2. 土壤与农业可持续发展国家重点实验室(中国科学院南京土壤研究所),南京 210008
小肽(small peptide),宽泛地是指在长度上少于100个氨基酸的蛋白[1],但也有人定义为5 ~ 60个氨基酸长度[2]。小肽参与了细胞增殖[3-4]、根系发育[5-10]、花粉育性[11-14]、气孔开关[15-16]、矿质元素的吸收和调控[9]、抵御病虫害[17-19]等生长发育和环境适应等诸多过程。同传统的植物激素一样,小肽发挥作用的位置和其合成产生的最初细胞和组织可以相同也可以距离很远,表明小肽可以作为长距离系统信号发挥作用。小肽发挥其生理功能的浓度很低,甚至在飞摩尔浓度(10–15 mol/L)下也可发挥生物学功能。同传统的植物激素不同的是,小肽本质上由氨基酸组成,外源施加不会对环境构成风险,而过多施加传统激素如生长素的人工合成产物2, 4-D会对环境构成风险;另外,几乎所有植物都会产生植物激素,而小肽具有物种特异性和环境诱导性的特征。尽管如此,小肽作为激素或称为信号分子,已经成为近年来的研究热点[1]。本文主要针对近30 a来植物小肽的来源、鉴定和调控方面的研究进展作一个初步总结,同时就这些方面仍然存在的问题进行讨论,为未来的研究提供参考。
1 植物小肽的来源历史上,有关小肽的研究已经获得过多次诺贝尔奖。早在1900年代,在寻求糖尿病治疗的过程中,有科学家提出小肽可以作为信号的设想。1921年班廷(Banting)和他的同事发现并纯化了胰岛素(insulin),并因此荣膺1923年的诺贝尔生理学或医学奖。1926年,Sanger解析了胰岛素的蛋白结构,发现其由长度分别为21和30个氨基酸的两条小肽通过二硫键形成,并因此获得1958年诺贝尔化学奖[20]。但直到1980年代,科学家才成功克隆了胰岛素的编码基因,发现胰岛素的两条小肽都由同一个基因编码,经过翻译后加工形成长度不同的两条小肽[21]。基于对胰岛素的深入研究,科学家提出了作为小肽激素必须满足的3个条件:①小(< 60个氨基酸);②可以分泌;③影响生理学过程[20]。虽然随着胰岛素的发现,动物中小肽的研究得到蓬勃发展,但直到1991年,才在番茄中首次报道了植物源的第一个功能小肽——系统素(systemin);成熟的系统素由18个氨基酸组成,由200个氨基酸长度的原系统素酶解加工而来;系统素参与调控了番茄对病虫害引起的伤害反应[22-23]。
如图 1所示,小肽从来源上可以分成3类:①原前体蛋白加工而成;②由独立的小ORF(蛋白阅读框)直接翻译而来;③由位于编码正常大小蛋白的5’-端或3’-端非翻译区(utr)上的小ORF编码而来。
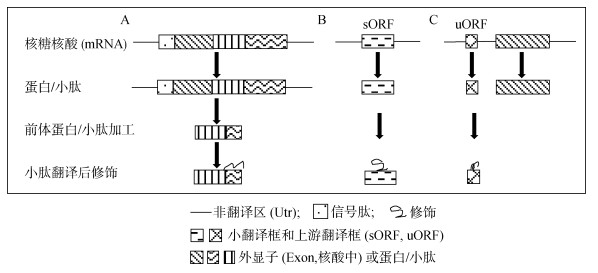
|
图 1 小肽的三种来源 Fig. 1 Sources of plant peptides |
对于第一类小肽,一般从原分泌蛋白靠近羧基端(C端)酶解加工而来。这类蛋白的氨基端往往含有由16 ~ 30个氨基酸构成的信号肽,指引其进入到内质网和高尔基体中进行进一步加工,包括切除信号肽和多轮蛋白降解和/或翻译后修饰,或分泌到胞外由胞外肽酶进一步降解切割,最终产生长度不等的活性肽。分泌型小肽一般长度为5 ~ 30个氨基酸,但如果是富含半胱氨酸的小肽,长度也可达60个氨基酸[2-24]。
虽然第二和第三类小肽都是来自于短的ORF,但其本质不同。对于第二类小肽,其编码基因是一个独立完整的基因。如果该基因含有较长的启动子和utr,那么该基因总长度可能比正常蛋白基因的长度还要长。例如,拟南芥小肽IMA1只有50个氨基酸,但其含有较长启动子和utr,仅RNA就达470个碱基对,启动子长度2 490个碱基对。通过氨基酸保守结构域比对发现拟南芥中共有8个IMA1类小肽基因[25]。由于这类小肽基因ORF较短,很难获得T-DNA插入缺失突变体,同时加上功能冗余,极大地阻碍了对该类小肽的鉴定。拟南芥基因组中共含有这类小ORF(sORF) 606 285个,其中570 948个位于基因间[26],这些基因间隔区的序列曾一度被认为是“垃圾DNA”。转录组数据分析也发现木棉中有12 852个sORF[27]。目前的研究表明,植物基因组中含有海量的小ORF,这也为研究小ORF的功能带来了难度。是否这些预测的小ORF都可以转录?转录组发现的小ORF是否可以翻译成小肽?还是绝大多数鉴定或预测的小ORF都是假阳性?由于小ORF序列短,通过T-DNA随机插入技术而获得相应突变体的成功率较低,也为从正向遗传学来解答这些小ORF的功能带来了挑战。现今基因靶向编辑技术的发展则为揭示这一类小ORF及其蛋白产物(小肽)的生物学功能提供了契机。
第三类小肽也是来自于小的ORF,但和第二类不同的是,其编码的小ORF不是独立基因,而是位于一个正常基因的utr区。目前在植物中发现的主要是位于5’UTR起始密码子上游的小ORF (uORF),而位于3’端下游的小ORF(dORF)目前在植物中还基本未见报道。该小ORF的翻译往往影响到其正常ORF的翻译,在功能上起到翻译调控作用,或者小ORF翻译产生的小肽和其正常ORF翻译产生的蛋白竞争同一个靶蛋白,对正常蛋白的生理功能起到调控作用。目前发现最短的uORF是由一个起始密码子紧接着一个终止密码子构成,这个uORF对下游主要蛋白的翻译以及RNA的降解起到调控作用。例如,拟南芥硼转运通道蛋白NIP5;1是低硼条件植株生长所必需的,但过多的硼会导致植物毒害,因此NIP5;1的表达量受到严格的调控。研究发现NIP5;1基因的5’UTR区一个由6个核苷酸构成的小ORF(AUG- UAA)参与了NIP5;1的调控:当硼过多时,蛋白翻译机器核糖体停转在AUG-STOP上,反复起始翻译,抑制了NIP5;1自身的翻译,同时导致NIP5;1RNA的降解,这样uORF以硼浓度依赖的方式调控了NIP5;1的表达,最终调控胞内硼稳态[28]。但是在高硼下,为什么上游uORF反复起始翻译的效率急剧升高的分子机制目前还不是很明确。
2 小肽的鉴定随着系统素的成功鉴定,基于生物分析为指导的生化纯化技术陆续鉴定到参与细胞增殖的小肽PSK[29]、富含羟脯氨酸的系统素类似物HypSys[30]以及维管系统干细胞命运调节小肽TDIF[31]等。从系统素在植物系统中首次报道以来的近30 a,陆续发现和报道参与植物生长发育和环境适应的多种小肽。但是相较于动物中鉴定到的小肽数量,植物中鉴定到的信号肽总体较少[32-34],极大限制了小肽的开发应用。如图 2所示,植物小肽鉴定的主要方法有以下几种,分别作一简述。
|
图 2 植物小肽的鉴定技术 Fig. 2 Techniques applied to identify plant peptides |
由于小肽分子量小、含量低,给传统的分离纯化方法带来了极大的挑战。比如,植物中第一个小肽,番茄系统素的研究中,仅对其生物活性的测试就用了3万多株番茄幼苗。运用传统的生化分析纯化技术,经过多步反向液相色谱和强阳离子交换液相色谱分析,最终从27 kg番茄叶片中获得大约1 μg的活性肽,随后通过氨基酸含量和序列分析,阐明了系统素的氨基酸组成(AVQSKPPSKRDPPKMQTD)和序列[23]。由此可见,虽然传统的生化分析具有鉴定到成熟小肽及其氨基酸序列的优势,但是传统的液相分离纯化和生化分析来研究植物小肽技术难度大、耗时费力、成本高、通量低。
随着高通量蛋白组学技术的发展,专门研究小肽的肽组学(peptidomics)技术已经应运而生,在动物组织中方兴未艾,成功鉴定到了新型具有生物活性的小肽,并且该技术可以鉴定到小肽的翻译后修饰,以及揭示蛋白降解途径[35-38]。但是该技术在植物小肽的鉴定中远没有发挥作用,主要有以下几个因素:①植物组织复杂,富含各种次级代谢产物和极低的植物信号肽浓度,给小肽的分离带来了困难;②小肽小,可能不含有胰肽酶的切点,或者切出来的片段过小,超出了质谱检测限;③新的蛋白质或肽段的质谱鉴定在很大程度上取决于对蛋白数据库的检索和比对,植物小肽数据库的局限性是肽组学技术的瓶颈之一。直到2014年,随着质谱仪器灵敏度的提高,Chen等[39]在植物中建立了新型肽组学技术,并应用该技术从番茄叶片中不仅成功鉴定到受伤害诱导的系统素,而且鉴定到14个新型的受伤害和茉莉酸甲酯诱导的、参与防御反应的信号肽;进一步对一个来自于病程相关蛋白1(PR-1b)C端的小肽CAPE1研究,发现外源添加ng级的该小肽就可以显著诱导抗病反应,为将来作物保护的生物防治提供了基础[39]。
虽然肽组学技术一次可以鉴定十几到几百个小肽,并且可以鉴定小肽翻译后修饰,大大提高了鉴定的通量,但植物小肽含量低,又没有完善的小肽数据库,极大增加了假阳性率。为了降低假阳性率,往往需要更严谨的搜库参数以及后续的验证实验,增加了工作量。另外,肽组学鉴定的小肽只能表明其存在,但无法判断其有无活性,还需要生物学、分子遗传学等实验来证明其生物学功能,这进一步增加了工作量。而且,由于不知道小肽产生的原蛋白到底在何处降解,因此目前还没有标准的小肽数据库,如果用某一物种的整个蛋白数据库来搜库的话,极大地增加了搜库时间和假阳性率,为了降低搜库时间和假阳性率,往往需要建立一个由整个物种蛋白C端50个氨基酸构成的一个假定的小肽数据库,并且产生一个由该库生成的随机库,然后用小肽质谱数据同时搜索这两个数据库,根据搜库分数和统计分析最后得到可能的小肽分子。尽管增加了搜库的严谨度,但鉴定到的小肽数量也很有限[39],同时肽组学技术对质谱仪的灵敏度和精确性都要求较高,因此,该技术在植物小肽信号分子鉴定中应用还需要进一步完善。
2.2 小肽鉴定的传统遗传学法基于突变体表型筛选的传统正向遗传学对基因功能的研究功不可没,但是对于小肽的鉴定效果并不理想,只鉴定到为数不多的小肽,主要是由于小肽及其前体来源广、复杂程度高,并且存在功能冗余,极大限制了通过正向遗传学的突变体表型研究来发现小肽和其生理功能的研究。1999年,在揭示拟南芥茎尖干细胞命运决定因子过程中,国外科学家通过正向遗传学克隆了拟南芥编码小肽CLV3(CLAVATA3)的基因[40],该基因调控了茎尖干细胞的数目[40-42],但成熟CLV3的功能结构还是通过生化分析和质谱技术才得以阐明[41-42]。2003年,另一个小肽基因INFLORESCENCE DEFICIENT IN ABSCISSION(IDA)也是通过该方法得以鉴定,但是IDA小肽结构和序列仍然未知[43]。从2003年以后,拟南芥中再没有通过经典遗传学鉴定到小肽信号,表明具有明显表型的、功能没有冗余的小肽基因的突变体已经筛选完毕、不复存在[2]。前期,我们通过基因编辑缺失IMA1小肽及其同源基因IMA3都没有发现任何表型,直到缺失拟南芥中包括IMA1在内的所有8个小肽基因才发现IMA参与了植物对铁的吸收,表明IMA各个成员间存在一定的功能冗余,通过过表达各个成员来研究其功能可能比基因突变的效果更好[25]。
2.3 小肽鉴定的生物信息学分析如上所述,不论传统的生化分析、经典遗传学,还是新近的肽组学技术,目前鉴定到的小肽数量都非常有限。拟南芥基因组分析揭示植物比动物含有10倍以上的可能的肽转运蛋白和受体,推测植物应该比动物含有更多的小肽,但目前情况恰恰相反,动物中鉴定到的小肽数量远远大于植物中鉴定的数量,考虑到植物的复杂性、不可移动性和无时无刻需要适应外界环境,有理由相信植物中还有大量的小肽迄待挖掘。
随着植物中鉴定到的小肽数量增加,对小肽自身和前体蛋白序列结构及其降解位点的分析,一些保守的序列特征已被揭示,加上越来越多的植物基因组被测序解析,转录组技术的发展,这些前期研究和组学技术为生物信息学从全基因组水平来鉴定已知小肽的同源物和预测新型小肽奠定了基础。早在2001年,有研究报道从全基因组水平上对LCV3的同源物进行了生物信息学分析,鉴定到一组信号肽,统称为CLE肽。CLE肽的C端拥有一个含有14个氨基酸的功能域——CLE功能域[44-45]。随后,在鉴定DEVIL和ROTUNDIFOLIA4小肽基础上,通过生物信息学的同源比对在拟南芥中和其他21个物种中鉴定DVL/RTFL的基因家族[46]。
除了同源比对,还可以预测新型的小肽。Hanada等[47]通过搜寻拟南芥基因组中所有介于30 ~ 100个密码子(也就是编码10 ~ 33个氨基酸长度的小肽)的小ORF,最后从基因间隔区找到7 901个小ORF。同样的,通过查询小于120个密码子的小ORF,在大豆中预测到766个小ORF[48]。Yang等[27]通过分析小于200个密码子的转录组学数据,在木棉中鉴定到1 282个小ORF,并且611个得到蛋白组学数据支持。
通过生物信息学分析结合其他技术手段,不仅可以有效鉴定小肽而且可以验证小肽的生物学功能。Hara等[49]首先通过生物信息学分析,从拟南芥全基因组水平上筛选出氨基酸数目少于150个,预测为分泌蛋白的基因153个,然后通过过表达所有153个基因,鉴定到编码小肽EPF1基因;过表达该基因显著降低气孔密度。Ohyama等[50]根据已知分泌小肽的原蛋白结构特征,结合生物信息学分析,筛选出那些结构相似的、预测为分泌蛋白的基因,然后再结合质谱分析,从而成功鉴定到C端编码小肽1(CEP1);该小肽参与了拟南芥侧根发育。Matsuzaki等[51]结合生物信息学分析和生物学分析,成功鉴定了拟南芥根尖生长因子(RGF)家族的相关小肽,这些小肽可以成功挽救由于TPST基因功能丧失导致的根尖缺失。近来,通过对单细胞转录组学数据的分析,成功鉴定到一些参与生殖过程的富含半胱氨酸的小肽信号[12-52]。
这些研究显示在小肽鉴定中,基于生物信息学的筛选再结合其他技术手段的强大功能,其成功克服了由于基因功能冗余和丰度低对小肽鉴定带来的障碍,同时也避免了传统生化分析技术的繁琐、耗时、技术要求高的缺点,是未来小肽鉴定和功能研究的发展方向。
3 小肽的调控随着对植物小肽研究的深入,不仅对小肽的来源和鉴定有了一定的理解,而且对小肽的调控也有了进一步的认识。植物小肽总体上可以分为两大类:分泌型和非分泌型。非分泌型小肽主要在胞内起作用,少数情况下也可以被送到胞外,担任细胞–细胞间的信号分子。如系统素虽然是非分泌型小肽,但随着伤害发生,系统素从伤害细胞释放出来,在质外体空间随蒸腾流到达非伤害部位引起抗性反应[23]。分泌型小肽又可以进一步分为翻译后修饰小肽和富含半胱氨酸小肽两类。富含半胱氨酸小肽一般含有偶数个半胱氨酸残基(典型的为6个或8个),用于分子内二硫键的形成。形成分子内二硫键的小肽结构稳定,不易被蛋白酶进一步降解。翻译后修饰类小肽一般较富含半胱氨酸小肽小,在成为有活性的功能小肽之前,往往需要一系列的成熟过程。首先是长度调控,这类小肽一般少于20个氨基酸,典型长度一般在10个氨基酸左右,由蛋白酶多次降解加工而成。和动物及酵母相似,这类植物小肽由较大的前体蛋白降解而来。这些前体蛋白在N端含有分泌信号序列,蛋白翻译后指导该类蛋白进入内质网和高尔基体,随后在这些细胞器内,切除N端信号序列,完成第一步加工,然后蛋白酶在靠近C端发生多轮降解加工,产生特定长度的小肽[2]。在动物中,前体蛋白的切割一般发生在C端成对碱性氨基酸之间,如赖氨酸-赖氨酸、赖氨酸-精氨酸、精氨酸-赖氨酸及精氨酸-精氨酸之间,肽键的切割由subtilisin/kexin-like原激素转化酶完成[53-54],切割完成后,末端碱性氨基酸由羧肽酶移除[53]。但研究发现植物小肽的加工过程和动物及酵母显著不同,主要体现在3个方面:①靠近成熟小肽N端的前体蛋白并没有发现成对的碱性氨基酸;②体外实验发现植物蛋白酶可以切割CLV3前体蛋白单个精氨酸的N端,也有报道位于三叶草成熟小肽CLE36 N端上游两个氨基酸处的甲硫氨酸和丝氨酸是前体蛋白的切割点;③拟南芥中的一个AtSBT1.1在体内负责PSK4的起始加工,但是切割位点是位于成熟小肽上游的亮氨酸和组氨酸之间。
这些研究表明起始加工位点和最后成熟小肽的边界序列没有特定的关系,植物小肽信号的蛋白降解加工是一系列复杂的生化过程:首先是蛋白酶内切产生初步小肽,随后外切蛋白酶对小肽进一步修剪,如去除末端几个氨基酸。发挥修剪作用的外切酶可能为含锌的羧肽酶SOL1[55]。迄今,植物体内有哪些蛋白酶来切割哪些原蛋白,以及某个或某类蛋白酶又如何决定切割位点的分子机制还不清楚。有一种解释是由于原蛋白翻译后修饰,阻止了体内复杂的蛋白降解系统对原蛋白的随机降解,只允许特定蛋白酶(类)可以接近特定位点,产生切割。蛋白内切酶产生的初级小肽又是如何面对如此丰富的蛋白外切酶的挑战而保持一定长度的呢?一种解释是成熟小肽末端的脯氨酸可能赋予初级小肽抵挡外切酶的降解,例如,CLE类小肽在第4、7和9位都含有保守的脯氨酸(图 3)。

|
图 3 CLE类小肽的氨基酸序列保守性 Fig. 3 Conserved amino acids of CLE peptide family |
小肽除了长度调控外,分泌型小肽还通常含有翻译后修饰,其中主要的翻译后修饰有3类:①酪氨酸硫酸化(tyrosine sulfation);②脯氨酸羟基化(proline hydroxylation);③羟脯氨酸阿拉伯糖基化(hydroxy-proline arabinosylation)。分泌蛋白的酪氨酸硫酸化修饰动植物中都存在。植物中带有该修饰的小肽有PSK[56]、PSY[57]和RGF[51]等,其中PSK是植物中第一个报道有修饰的小肽。介导该修饰的酶为TPST (tyrosylprotein sulfotransferase),催化了硫酸根从3′-磷酸腺苷-5′-磷酰硫酸(3′-phosphoadenosine 5′-phos-pho-sulfate,PAPS)向酪氨酸苯环转移[58]。
缺失TPST基因的拟南芥地上部矮小、叶片白化、早衰,根部表现为根极短、根尖原基活性显著下降,究其原因就是突变体丧失了对RGF小肽的硫酸化修饰,从而无法维持根尖干细胞的活性。这些研究表明小肽酪氨酸硫酸化的重要性。目前对于硫酸化修饰的具体分子机制还不是十分明确,但研究发现酪氨酸硫酸化的最基本要求是酪氨酸的N端必须连接一个天冬氨酸(也就是DYxxxxxx),如果一个酪氨酸附近的酸性氨基酸越多,该酪氨酸硫酸化的可能性就极显著地增加[58]。
除了PSK小肽家族,目前发现植物中几乎所有修饰小肽都发生脯氨酸的羟基化修饰。脯氨酸羟基化由2-酮戊二酸依赖性双加氧酶家族(2-oxoglutarate- dependent dioxygenase)的脯氨酰-4-羟化酶(prolyl-4- hydroxylase)介导完成,需要2-酮戊二酸和氧气为共同的底物[59]。脯氨酰-4-羟化酶是一个跨膜蛋白,亚细胞定位于内质网和高尔基体复合物中。拟南芥中总共有13个该酶的编码基因,缺失该酶基因导致根毛极性生长丧失[60]。
一些小肽如PSY1、CLV3、CLE2、CLE9和CLE-RS2的羟脯氨酸进一步发生修饰,主要是羟基和阿拉伯糖反应形成O型阿拉伯糖链[61]。羟脯氨酸O-阿拉伯糖基转移酶(hydroxyproline o-arabinosyl-transferase,HPAT)负责羟脯氨酸4位上β糖苷键的连接。最近发现拟南芥HPAT是一个跨膜蛋白,亚细胞定位于高尔基体上,属于糖基转移酶(glycosyl-transferase,GT)8号家族[62]。缺失拟南芥HPAT基因导致多种表型:下胚轴变长、细胞壁加厚受阻、早花、早衰。HPAT1和HPAT3双突变体显著影响花粉管生长,进而导致雄性不育。缺失豌豆和三叶草中拟南芥HPAT的同源基因NOD3和RDN1导致高度结瘤的表型,有力支持了CLE小肽的羟脯氨酸O型阿拉伯糖基化可以抑制根际微生物的过度结瘤[63]。综上所述,小肽的糖基化修饰在植物生长发育和防御中起到多种多样的作用。
4 问题与展望小肽作为信号分子,正如传统植物激素一样,往往起到“四两拨千斤”的作用。外源添加纳克级甚至飞克级小肽,就可以提高番茄抵抗病虫害的伤害,提高植物对氮的吸收,增加植物铁含量。植物信号肽还参与了其他生物学过程如细胞增殖、根系干细胞维持、侧根生长发育、根瘤形成等。本文初步总结了近30 a来植物小肽的来源、鉴定和调控,重点介绍了植物小肽分离鉴定的几种方法,指出了每种方法的优势和局限性。虽然植物小肽的研究已经取得一些进展,但挑战仍然巨大:①近10 a来,各种组学技术日新月异,尤其是近年来基因组和转录组学相关研究已从模式植物拟南芥、水稻等成功延伸到各种经济作物甚至目前看起来没什么经济价值的植物。随之而来的是越来越多的已知植物信号肽的同源物和新型小肽,特别是一些物种特异性和环境适应性的信号肽被进一步发掘和鉴定。如何高效地验证如此海量小肽的生物学功能并在生产中充分利用如此丰富的小肽资源是未来研究的一个重要方向和巨大挑战;②虽然植物源小肽越来越丰富,但在林木中,有关小肽的研究还鲜有报道。另外,正如前面所述,小肽小、浓度低,很难获得T-DNA插入突变体,通过传统遗传学和生物化学方法来鉴定小肽不仅难度大,而且费时耗力。未来包括小肽在内的新基因的功能研究,可能都将采用“生物信息学预测–突变体表型–基因身份鉴定”这一套综合的方法。总之,植物小肽是一个新兴的、极具前景的研究领域,但是其数量多、来源和加工成熟机制复杂、生物学功能研究难度大,目前植物小肽研究技术手段还不够成熟,给植物小肽的研究增加了难度,但同时也为植物小肽的研究提供了更大的契机。正如人类对土壤微生物的认知,有众多的空白等待去填补。一旦突破,将小肽成功应用到生产中就可以减少农药化肥等的施用量,起到减肥增效的效果,保护生态环境;同时可以提高果蔬的品质和林木的材质,为现代高质农业生产服务。
| [1] |
Hsu P Y, Benfey P N. Small but mighty: Functional peptides encoded by small ORFs in plants[J]. Proteomics, 2018, 18(10): e1700038 DOI:10.1002/pmic.201700038 (  0) 0) |
| [2] |
Matsubayashi Y. Posttranslationally modified small-peptide signals in plants[J]. Annu. Rev. Plant Biol., 2014, 65: 385-413 DOI:10.1146/annurev-arplant-050312-120122 (  0) 0) |
| [3] |
Djordjevic M A, Mohd-Radzman N A, Imin N. Small- peptide signals that control root nodule number, develop-ment, and symbiosis[J]. J. Exp. Bot., 2015, 66(17): 5171-5181 DOI:10.1093/jxb/erv357 (  0) 0) |
| [4] |
Imin N, Mohd-Radzman N A, Ogilvie H A, et al. The peptide-encoding cep1 gene modulates lateral root and nodule numbers in medicago truncatula[J]. J. Exp. Bot., 2013, 64(17): 5395-5409 DOI:10.1093/jxb/ert369 (  0) 0) |
| [5] |
Delay C, Imin N, Djordjevic M A. Cep genes regulate root and shoot development in response to environmental cues and are specific to seed plants[J]. J. Exp. Bot., 2013, 64(17): 5383-5394 DOI:10.1093/jxb/ert332 (  0) 0) |
| [6] |
Delay C, Imin N, Djordjevic M A. Regulation of arabidopsis root development by small signaling peptides[J]. Front Plant Sci., 2013, 4: 352 (  0) 0) |
| [7] |
Mohd-Radzman N A, Binos S, Truong T T, et al. Novel mtcep1 peptides produced in vivo differentially regulate root development in medicago truncatula[J]. J. Exp. Bot., 2015, 66(17): 5289-5300 DOI:10.1093/jxb/erv008 (  0) 0) |
| [8] |
Patel N, Mohd-Radzman N A, Corcilius L, et al. Diverse peptide hormones affecting root growth identified in the medicago truncatula secreted peptidome[J]. Mol. Cell Proteomics, 2018, 17(1): 160-174 DOI:10.1074/mcp.RA117.000168 (  0) 0) |
| [9] |
Taleski M, Imin N, Djordjevic M A. Cep peptide hormones: Key players in orchestrating nitrogen-demand signalling, root nodulation, and lateral root development[J]. J. Exp. Bot., 2018, 69(8): 1829-1836 DOI:10.1093/jxb/ery037 (  0) 0) |
| [10] |
Taleski M, Imin N, Djordjevic M A. New role for a cep peptide and its receptor: Complex control of lateral roots[J]. J. Exp. Bot., 2016, 67(16): 4797-4799 DOI:10.1093/jxb/erw306 (  0) 0) |
| [11] |
Higashiyama T, Takeuchi H. The mechanism and key molecules involved in pollen tube guidance[J]. Annu. Rev. Plant Biol., 2015, 66: 393-413 DOI:10.1146/annurev-arplant-043014-115635 (  0) 0) |
| [12] |
Okuda S, Tsutsui H, Shiina K, et al. Defensin-like polypeptide lures are pollen tube attractants secreted from synergid cells[J]. Nature, 2009, 458(7236): 357-361 DOI:10.1038/nature07882 (  0) 0) |
| [13] |
Takeuchi H, Higashiyama T. A species-specific cluster of defensin-like genes encodes diffusible pollen tube attractants in arabidopsis[J]. PLoS Biol., 2012, 10(12): e1001449 DOI:10.1371/journal.pbio.1001449 (  0) 0) |
| [14] |
Takeuchi H, Higashiyama T. Tip-localized receptors control pollen tube growth and lure sensing in arabidopsis[J]. Nature, 2016, 531(7593): 245-248 DOI:10.1038/nature17413 (  0) 0) |
| [15] |
Qu X, Cao B, Kang J, et al. Fine-tuning stomatal movement through small signaling peptides[J]. Front Plant Sci., 2019, 10: 69 DOI:10.3389/fpls.2019.00069 (  0) 0) |
| [16] |
Takahashi F, Suzuki T, Osakabe Y, et al. A small peptide modulates stomatal control via abscisic acid in long- distance signalling[J]. Nature, 2018, 556(7700): 235-238 DOI:10.1038/s41586-018-0009-2 (  0) 0) |
| [17] |
Stotz H U, Spence B, Wang Y. A defensin from tomato with dual function in defense and development[J]. Plant Mol. Biol., 2009, 71(1/2): 131-143 (  0) 0) |
| [18] |
Stotz H U, Thomson J G, Wang Y. Plant defensins: Defense, development and application[J]. Plant Signal Behav., 2009, 4(11): 1010-1012 DOI:10.4161/psb.4.11.9755 (  0) 0) |
| [19] |
Ziemann S, van der Linde K, Lahrmann U, et al. An apoplastic peptide activates salicylic acid signalling in maize[J]. Nat. Plants, 2018, 4(3): 172-180 DOI:10.1038/s41477-018-0116-y (  0) 0) |
| [20] |
Rosenfeld L. Insulin: Discovery and controversy[J]. Clin. Chem., 2002, 48(12): 2270-2288 (  0) 0) |
| [21] |
Bell G I, Pictet R L, Rutter W J, et al. Sequence of the human insulin gene[J]. Nature, 1980, 284(5751): 26-32 DOI:10.1038/284026a0 (  0) 0) |
| [22] |
McGurl B, Pearce G, Orozco-Cardenas M, et al. Structure, expression, and antisense inhibition of the systemin precursor gene[J]. Science, 1992, 255(5051): 1570-1573 DOI:10.1126/science.1549783 (  0) 0) |
| [23] |
Pearce G, Strydom D, Johnson S, et al. A polypeptide from tomato leaves induces wound-inducible proteinase inhibitor proteins[J]. Science, 1991, 253(5022): 895-897 DOI:10.1126/science.253.5022.895 (  0) 0) |
| [24] |
Tavormina P, De Coninck B, Nikonorova N, et al. The plant peptidome: An expanding repertoire of structural features and biological functions[J]. Plant Cell, 2015, 27(8): 2095-2118 DOI:10.1105/tpc.15.00440 (  0) 0) |
| [25] |
Grillet L, Lan P, Li W, et al. Iron man is a ubiquitous family of peptides that control iron transport in plants[J]. Nat Plants, 2018, 4(11): 953-963 DOI:10.1038/s41477-018-0266-y (  0) 0) |
| [26] |
Hanada K, Zhang X, Borevitz J O, et al. A large number of novel coding small open reading frames in the intergenic regions of the arabidopsis thaliana genome are transcribed and/or under purifying selection[J]. Genome Res., 2007, 17(5): 632-640 DOI:10.1101/gr.5836207 (  0) 0) |
| [27] |
Yang X, Tschaplinski T J, Hurst G B, et al. Discovery and annotation of small proteins using genomics, proteomics, and computational approaches[J]. Genome Res., 2011, 21(4): 634-641 (  0) 0) |
| [28] |
Tanaka M, Sotta N, Yamazumi Y, et al. The minimum open reading frame, aug-stop, induces boron-dependent ribosome stalling and mrna degradation[J]. Plant Cell, 2016, 28(11): 2830-2849 DOI:10.1105/tpc.16.00481 (  0) 0) |
| [29] |
Yang H, Matsubayashi Y, Nakamura K, et al. Oryza sativa psk gene encodes a precursor of phytosulfokine-alpha, a sulfated peptide growth factor found in plants[J]. Proc. Natl. Acad. Sci. USA, 1999, 96(23): 13560-13565 DOI:10.1073/pnas.96.23.13560 (  0) 0) |
| [30] |
Pearce G, Moura D S, Stratmann J, et al. Production of multiple plant hormones from a single polyprotein precursor[J]. Nature, 2001, 411(6839): 817-820 DOI:10.1038/35081107 (  0) 0) |
| [31] |
Ito Y, Nakanomyo I, Motose H, et al. Dodeca-cle peptides as suppressors of plant stem cell differentiation[J]. Science, 2006, 313(5788): 842-845 DOI:10.1126/science.1128436 (  0) 0) |
| [32] |
Butenko M A, Vie A K, Brembu T, et al. Plant peptides in signalling: Looking for new partners[J]. Trends Plant Sci., 2009, 14(5): 255-263 (  0) 0) |
| [33] |
Farrokhi N, Whitelegge J P, Brusslan J A. Plant peptides and peptidomics[J]. Plant Biotechnol. J., 2008, 6(2): 105-134 DOI:10.1111/j.1467-7652.2007.00315.x (  0) 0) |
| [34] |
Olsson V, Joos L, Zhu S S, et al. Look closely, the beautiful may be small: Precursor-derived peptides in plants[J]. Annu. Rev. Plant Biol., 2019, 70(1): 153-186 DOI:10.1146/annurev-arplant-042817-040413 (  0) 0) |
| [35] |
Che F Y, Lim J, Pan H, et al. Quantitative neuropeptidomics of microwave-irradiated mouse brain and pituitary[J]. Mol. Cell Proteomics, 2005, 4(9): 1391-1405 DOI:10.1074/mcp.T500010-MCP200 (  0) 0) |
| [36] |
Svensson M, Skold K, Svenningsson P, et al. Peptidomics-based discovery of novel neuropeptides[J]. J. Proteome Res., 2003, 2(2): 213-219 DOI:10.1021/pr020010u (  0) 0) |
| [37] |
Tinoco A D, Kim Y G, Tagore D M, et al. A peptidomics strategy to elucidate the proteolytic pathways that inactivate peptide hormones[J]. Biochemistry, 2011, 50(12): 2213-2222 DOI:10.1021/bi2000033 (  0) 0) |
| [38] |
Tinoco A D, Saghatelian A. Investigating endogenous peptides and peptidases using peptidomics[J]. Biochemistry, 2011, 50(35): 7447-7461 DOI:10.1021/bi200417k (  0) 0) |
| [39] |
Chen Y L, Lee C Y, Cheng K T, et al. Quantitative peptidomics study reveals that a wound-induced peptide from pr-1 regulates immune signaling in tomato[J]. Plant Cell, 2014, 26(10): 4135-4148 DOI:10.1105/tpc.114.131185 (  0) 0) |
| [40] |
Fletcher J C, Brand U, Running M P, et al. Signaling of cell fate decisions by clavata3 in arabidopsis shoot meristems[J]. Science, 1999, 283(5409): 1911-1914 DOI:10.1126/science.283.5409.1911 (  0) 0) |
| [41] |
Kondo T, Sawa S, Kinoshita A, et al. A plant peptide encoded by clv3 identified by in situ maldi-tof ms analysis[J]. Science, 2006, 313(5788): 845-848 DOI:10.1126/science.1128439 (  0) 0) |
| [42] |
Ohyama K, Shinohara H, Ogawa-Ohnishi M, et al. A glycopeptide regulating stem cell fate in arabidopsis thaliana[J]. Nat. Chem. Biol., 2009, 5(8): 578-580 DOI:10.1038/nchembio.182 (  0) 0) |
| [43] |
Butenko M A, Patterson S E, Grini P E, et al. Inflorescence deficient in abscission controls floral organ abscission in arabidopsis and identifies a novel family of putative ligands in plants[J]. Plant Cell, 2003, 15(10): 2296-2307 DOI:10.1105/tpc.014365 (  0) 0) |
| [44] |
Cock J M, McCormick S. A large family of genes that share homology with clavata3[J]. Plant Physiol., 2001, 126(3): 939-942 DOI:10.1104/pp.126.3.939 (  0) 0) |
| [45] |
Oelkers K, Goffard N, Weiller G F, et al. Bioinformatic analysis of the cle signaling peptide family[J]. BMC Plant Biol., 2008, 8: 1 DOI:10.1186/1471-2229-8-1 (  0) 0) |
| [46] |
Guo P, Yoshimura A, Ishikawa N, et al. Comparative analysis of the rtfl peptide family on the control of plant organogenesis[J]. J. Plant Res., 2015, 128(3): 497-510 DOI:10.1007/s10265-015-0703-1 (  0) 0) |
| [47] |
Hanada K, Higuchi-Takeuchi M, Okamoto M, et al. Small open reading frames associated with morphogenesis are hidden in plant genomes[J]. Proc. Natl. Acad. Sci. USA, 2013, 110(6): 2395-2400 DOI:10.1073/pnas.1213958110 (  0) 0) |
| [48] |
Guillen G, Diaz-Camino C, Loyola-Torres C A, et al. Detailed analysis of putative genes encoding small proteins in legume genomes[J]. Front Plant Sci., 2013, 4: 208 (  0) 0) |
| [49] |
Hara K, Kajita R, Torii K U, et al. The secretory peptide gene epf1 enforces the stomatal one-cell-spacing rule[J]. Genes. Dev., 2007, 21(14): 1720-1725 DOI:10.1101/gad.1550707 (  0) 0) |
| [50] |
Ohyama K, Ogawa M, Matsubayashi Y. Identification of a biologically active, small, secreted peptide in arabidopsis by in silico gene screening, followed by lc-ms-based structure analysis[J]. Plant J., 2008, 55(1): 152-160 DOI:10.1111/j.1365-313X.2008.03464.x (  0) 0) |
| [51] |
Matsuzaki Y, Ogawa-Ohnishi M, Mori A, et al. Secreted peptide signals required for maintenance of root stem cell niche in arabidopsis[J]. Science, 2010, 329(5995): 1065-1067 DOI:10.1126/science.1191132 (  0) 0) |
| [52] |
Sprunck S, Rademacher S, Vogler F, et al. Egg cell-secreted ec1 triggers sperm cell activation during double fertilization[J]. Science, 2012, 338(6110): 1093-1097 DOI:10.1126/science.1223944 (  0) 0) |
| [53] |
Fuller R S, Sterne R E, Thorner J. Enzymes required for yeast prohormone processing[J]. Annu. Rev. Physiol., 1988, 50: 345-362 DOI:10.1146/annurev.ph.50.030188.002021 (  0) 0) |
| [54] |
Rehemtulla A, Kaufman R J. Protein processing within the secretory pathway[J]. Curr. Opin. Biotechnol., 1992, 3(5): 560-565 DOI:10.1016/0958-1669(92)90086-X (  0) 0) |
| [55] |
Casamitjana-Martinez E, Hofhuis H F, Xu J, et al. Root-specific cle19 overexpression and the sol1/2 suppressors implicate a clv-like pathway in the control of arabidopsis root meristem maintenance[J]. Curr. Biol., 2003, 13(16): 1435-1441 DOI:10.1016/S0960-9822(03)00533-5 (  0) 0) |
| [56] |
Matsubayashi Y, Sakagami Y. Phytosulfokine, sulfated peptides that induce the proliferation of single mesophyll cells of asparagus officinalis l[J]. Proc. Natl. Acad. Sci. USA, 1996, 93(15): 7623-7627 DOI:10.1073/pnas.93.15.7623 (  0) 0) |
| [57] |
Amano Y, Tsubouchi H, Shinohara H, et al. Tyrosine-sulfated glycopeptide involved in cellular proliferation and expansion in arabidopsis[J]. Proc. Natl. Acad. Sci. USA, 2007, 104(46): 18333-18338 DOI:10.1073/pnas.0706403104 (  0) 0) |
| [58] |
Hanai H, Nakayama D, Yang H, et al. Existence of a plant tyrosylprotein sulfotransferase: Novel plant enzyme catalyzing tyrosine o-sulfation of preprophytosulfokine variants in vitro[J]. FEBS Lett., 2000, 470(2): 97-101 DOI:10.1016/S0014-5793(00)01299-0 (  0) 0) |
| [59] |
Myllyharju J. Prolyl 4-hydroxylases, the key enzymes of collagen biosynthesis[J]. Matrix. Biol., 2003, 22(1): 15-24 DOI:10.1016/S0945-053X(03)00006-4 (  0) 0) |
| [60] |
Velasquez S M, Ricardi M M, Dorosz J G, et al. O-glycosylated cell wall proteins are essential in root hair growth[J]. Science, 2011, 332(6036): 1401-1403 DOI:10.1126/science.1206657 (  0) 0) |
| [61] |
Okamoto S, Shinohara H, Mori T, et al. Root-derived cle glycopeptides control nodulation by direct binding to har1 receptor kinase[J]. Nat. Commun., 2013, 4: 2191 DOI:10.1038/ncomms3191 (  0) 0) |
| [62] |
Ogawa-Ohnishi M, Matsushita W, Matsubayashi Y. Identification of three hydroxyproline o-arabinosyltran-sferases in arabidopsis thaliana[J]. Nat. Chem. Biol., 2013, 9(11): 726-730 DOI:10.1038/nchembio.1351 (  0) 0) |
| [63] |
Okamoto S, Ohnishi E, Sato S, et al. Nod factor/nitrate- induced cle genes that drive har1-mediated systemic regulation of nodulation[J]. Plant Cell Physiol., 2009, 50(1): 67-77 (  0) 0) |
2. State Key Laboratory of Soil and Sustainable Agriculture, Institute of Soil Science, Chinese Academy of Sciences, Nanjing 210008, China
 2019, Vol. 51
2019, Vol. 51


