2. 河南商丘农田生态系统国家野外科学观测研究站, 河南商丘 476000
根际是植物与土壤物质和能量交换的活跃区域[1],植物通过根系分泌物为根际微生物的生长提供基质和信号[2-4],影响着微生物生长的生态位、细菌的形态和生理等特征[5]。而受根系分泌物形态的影响,根际微生物则能通过改变根际养分的有效性,影响植物对养分的吸收,协助抵御病原菌侵染和改变对土壤有机碳的分解速率[6-9]。土壤微生物是有机碳分解转化的驱动者,其中,细菌作为根际最丰富的微生物,它们对土壤有机碳分解转化的影响不可忽视[10-11]。
氮作为植物生长所需的大量元素,氮素水平成为土壤生产力的主要限制性因素。综合前人研究,施氮量对土壤细菌群落的组成和多样性影响各异。Li等[12]研究发现,种植玉米的砂质黏壤土细菌群落的多样性和丰富度随着氮肥用量的增加而减少;而也有研究发现施氮量对常年耕种的农田作物根际土壤细菌组成有显著影响,但对细菌多样性没有影响[13-14]。究其原因可能归为两个方面:①土壤类型或者同种土壤的不同种植制度都会引起土壤理化性质的差异和土壤细菌群落结构的改变,进而造成细菌种群对氮肥的响应各异[15];②土壤细菌群落结构的差异致其驱动土壤氮循环过程的不同,进而影响土壤中氮的形态,而氮形态可以通过影响植物根系沉积物形态来调节根际土壤细菌的活性和群落组成[16]。基于反馈调节机制,由根系分泌物引起的根际土壤细菌群落结构和多样性的差异也会直接影响土壤有机碳的分解代谢[17]。大量研究表明,细菌对有机碳形态具有选择利用性[18-19],相对于含–CH3的物质,含–COOH的有机物质更容易被细菌矿化;革兰氏阴性细菌菌群优先选择易分解态有机质,而革兰氏阳性细菌菌群对难分解态有机质的利用更有优势[8, 20]。由于参与有机碳分解代谢的细菌种群复杂多样,一种细菌可能含有参与有机碳分解的多种关键酶基因,单从细菌种群结构的水平解释土壤有机碳的分解过程远远不够[21]。而基于PICRUSt功能预测有助于深入挖掘参与有机质分解过程中的关键功能基因丰度的差异。综上,土壤性质和施氮水平调控细菌群落结构和参与有机质分解代谢的微生物过程,因此,深入了解不同茬口土壤和施氮水平对下季作物根际土壤细菌参与有机碳分解代谢过程关键功能基因丰度的影响,对改善种植制度和提高土壤有机碳的固定能力具有指导性意义。
基于上述目的,本研究以收获小麦和西瓜后种植的南瓜根际土壤为研究对象,以施氮水平为处理,通过南瓜盆栽试验研究根际土壤细菌群落结构对不同茬口土壤和施氮水平的响应,重点解析参与有机碳分解代谢细菌的关键功能基因表达特征,为深刻理解根际土壤细菌群落参与有机碳分解代谢的生理生态过程提供一定的理论依据。
1 材料与方法 1.1 试验设计南瓜盆栽试验在河南商丘农田生态系统国家野外科学观测研究站(34°35.22′ N,1115°34.52′ E)日光温室内开展。供试土壤采自于站内农田,为西瓜茬土壤和小麦茬土壤,土壤质地为砂壤土。采用多点采样法分别采集西瓜茬和小麦茬表层0 ~ 20 cm土样,去除动植物残体及碎石,风干后过5 mm筛,充分混合备用。供试土壤基本理化指标如表 1所示。
|
|
表 1 供试土壤基本理化性质 Table 1 Basic properties of tested soils |
供试白籽南瓜(日本铁甲)经消毒、漂洗以及催芽后选择健康且长势一致的幼苗移栽到温室中装有10.00 kg风干土的盆钵(直径25 cm ×高20 cm)。在南瓜苗移栽定植后(每盆2棵南瓜苗),按照相当于纯氮施入量分别为1.00和2.00 g/kg施入复合肥料(N∶P2O5∶K2O = 15∶15∶15)。按照正交试验设计设置4个处理,分别为:西瓜茬土+低氮、西瓜茬土+高氮、小麦茬土+低氮和小麦茬土+高氮,每个处理10个重复。在南瓜生长期间保持温室白天温度范围为18 ~ 30 ℃,夜间温度低于15 ℃。
1.2 样品采集与测定 1.2.1 样品采集在种植30 d后,先将南瓜植株从土壤中轻轻拔出,抖掉根系上的松散土,然后将与根系紧密结合的土壤刷下作为根际土。根际土混合均匀后,经过液氮速冻处理,保存在–80 ℃冰箱,用于细菌群落多样性分析。收集盆钵中剩余的土壤,混合均匀后用于土壤理化指标的测定。
1.2.2 样品测定采用Fast DNA®SPIN试剂盒(Qbiogene Inc., USA)提取土壤总DNA,DNA浓度和纯度利用NanoDrop2000进行检测,利用1% 琼脂糖凝胶电泳检测DNA提取质量。在设计测序引物时,在序列中添加bar-code序列以区分各个样品测序数据。用338F(5′-ACTCCTACGGGAGGCAGCAG-3′)和806R(5′-GGACTACHVGGGTWTCTAAT-3′)引物对V3 ~ V4可变区进行PCR扩增,扩增程序为:95 ℃预变性3 min,27个循环(95 ℃变性30 s,55 ℃退火30 s, 72 ℃延伸30 s),最后72 ℃延伸10 min(PCR仪:ABI GeneAmp® 9700型)。扩增体系为20 μl,4 μl 5×FastPfu缓冲液,2 μl 2.5 mmol/L dNTPs,0.8 μl引物(5 μmmol/L),0.4 μl FastPfu聚合酶;10 ng DNA模板。使用2% 琼脂糖凝胶回收PCR产物,利用AxyPrep DNA Gel Extraction Kit(Axygen Biosciences, Union City, CA, USA)进行纯化,Tris-HCl洗脱,2% 琼脂糖电泳检测。利用QuantiFluorTM-ST(Promega, USA)进行检测定量。根据定量结果和测序量要求,取PCR产物构建测序文库。构建好的文库在Illumina Miseq PE300测序平台测序。
基于《土壤农化分析》方法[22],称取新鲜土样,按照水土比10∶1(m/V),采用0.01 mol/L KCl浸提,AA3连续流动分析仪测定铵态氮和硝态氮含量;有效磷采用NaHCO3浸提–钼锑抗比色法测定;速效钾采用NH4OAc浸提–火焰光度法测定;土壤水分含量采用105 ℃烘干称重法测定。
1.3 数据分析数据处理在Excel 2007中完成。Shannon多样性指数利用QIME 1.80计算。细菌群落功能基因预测基于PICRUSt分析实现[23],通过PICRUSt(PICRUSt软件存储了greengene ID对应的KEGG Ortholog(KO)信息)对OTU丰度表进行标准化,并去除16S标记基因在物种基因组中的拷贝(copy)数目的影响;然后通过每个OTU对应的greengene ID,获得OTU对应的(KO)信息;并计算各功能基因对应的KO丰度,该分析可在http://huttenhower.sph.harvard.edu/galaxy/中在线实现。基于R 3.4.3的vegan包,采用非参数多元统计比对组间细菌群落结构差异;基于SPSS 16.0配对T检验研究组间土壤理化性质的差异;基于STAMP的Welch T检验对比组间微生物种属差异和组间参与氮循环的功能基因相对丰度差异;采用双因素方差分析揭示不同茬口土壤和施氮水平对南瓜根际细菌Shannon多样性以及微生物群落结构的影响。基于Canoco 4.5冗余分析揭示土壤理化性质对根际细菌群落结构的作用。所有作图在Origin 8.5中完成。
2 结果与分析 2.1 土壤理化性质的差异配对T检验的结果发现(表 2),不同茬口土壤和施氮水平并未影响土壤铵态氮和硝态氮的含量,且土壤中以硝态氮含量为主。但不同茬口土壤显著影响低氮处理条件下有效磷的含量,有效磷在小麦茬土壤中的含量约为在西瓜茬土壤中的1.7倍(P < 0.05)。在西瓜茬土壤中,高氮处理土壤有效磷的含量约为低氮处理土壤的2倍(P < 0.05)。在小麦茬土壤中,高氮处理土壤速效钾的含量为426.01 mg/kg,约为低氮处理土壤的1.5倍(P < 0.05)。
|
|
表 2 土壤理化性质差异 Table 2 Differences in soil physiochemical properties |
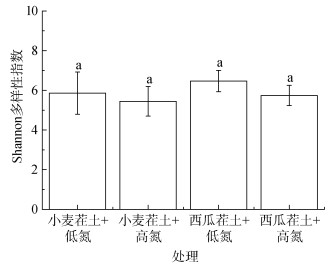
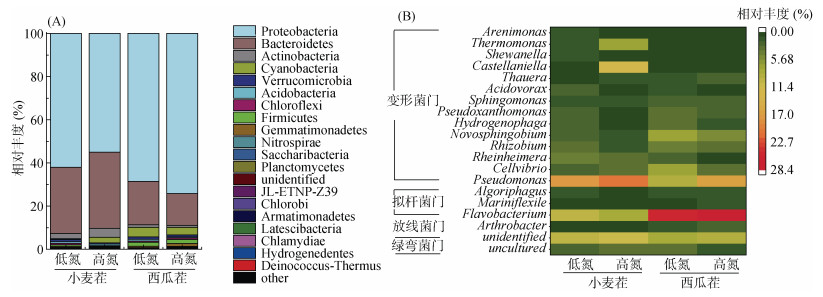
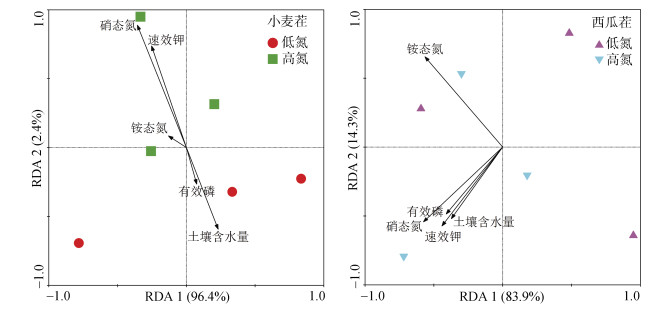
基于图 1的结果发现,不同茬口土壤和施氮水平并未改变根际土壤细菌Shannon多样性指数,且对细菌群落结构的影响不显著(表 3,P > 0.05)。在门水平上对土壤中细菌群落结构分析发现,所有处理细菌群落结构中相对丰度较高的群体为变形菌门(Proteobacteria)、拟杆菌门(Bacterodidetes)、放线菌门(Actinobacteria)和蓝藻细菌门(Cyanobacteria),四者之和约达到90%(图 3A)。与西瓜茬土壤高氮处理相比,小麦茬土壤高氮处理根际变形菌门的相对丰度降低,但拟杆菌门和放线菌门的相对丰度均升高(P < 0.05)。在属水平上,相对丰度前20位的菌属主要属于变形菌门、拟杆菌门和绿弯菌门(Chloroflexi),其中变形菌门假单胞菌属(Pseudomonas)和拟杆菌门黄杆菌属(Flavobacterium)为各处理的优势菌属(图 2B)。在西瓜茬土壤中,与低氮处理相比,高氮处理降低了氢噬胞菌属(Hydrogenophaga)的相对丰度(P < 0.05)。与西瓜茬土壤低氮处理相比,小麦茬土壤低氮处理明显提高了假单胞菌属的相对丰度(P < 0.05)。与西瓜茬土壤高氮处理相比,小麦茬土壤高氮处理明显提高了假黄单胞菌属(Pseudoxanthomonas)、根瘤菌属(Rhizobium)、黄杆菌属(Flavobacterium)、新鞘氨醇杆菌属(Novosphingobium)和节杆菌属(Arthrobacter)的相对丰度(图 2B,P < 0.05)。冗余分析的结果发现,土壤铵态氮、硝态氮、有效磷、速效钾以及含水量对小麦茬土壤和西瓜茬土壤根际细菌群落的解释率分别为98.8% 和98.2%,其中小麦茬土壤中硝态氮的作用大于其他指标,而在西瓜茬土壤中铵态氮和硝态氮的作用相对更大(图 3)。
|
图 1 基于OTU水平的细菌Shannon多样性指数 Fig. 1 Shannon index of bacterial diversity on OTUs level |
|
|
表 3 基于非参数多元统计方法(ANOSIM,Adonis和MRPP)的不同处理细菌群落结构差异 Table 3 Differences in bacterial community compositions under different treatments using non-parametric multivariate statistical approaches (ANOSIM, Adonis and MRPP)s |
|
(相对丰度前20位的菌门和属) 图 2 基于门(A)和属(B)水平的细菌微生物群落结构 Fig. 2 Bacterial community structures at phyla (A) and genus (B) levels (with relative abundance in top 20) |
|
图 3 基于冗余分析的环境因子与细菌微生物群落结构的关系 Fig. 3 Redundancy analysis plot showing relationship between environmental factors and bacterial community structure |
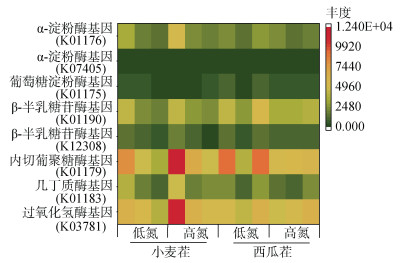
基于图 4和Welch's T检验的结果表明,在同种茬口土壤条件下,施氮水平对参与有机碳分解代谢的关键功能基因丰度的影响不大;但在相同施肥水平下,参与有机碳分解代谢的关键功能基因丰度受不同茬口土壤的影响显著。在低氮处理下,小麦茬土壤显著提高了根际含有α-淀粉酶基因(K01176)和β-半乳糖苷酶基因的丰度(P < 0.05)。在高氮处理下,小麦茬土壤显著提高了根际含有β-半乳糖苷酶基因的丰度(P < 0.05)。
|
图 4 参与碳分解代谢的关键功能基因丰度图 Fig. 4 Heat map showing key functional bacterial genes involved in carbon decomposition metabolism |
本研究结果发现,不同茬口和施氮水平并未改变根际土壤细菌Shannon多样性指数和细菌群落结构,说明南瓜经过为期1个月的生长,土壤根际细菌群落组成已经相当稳定。随着培养时间的延长,不同处理间影响根际细菌群落结构及多样性的关键因子差异逐渐消失,根际微环境的稳定,导致不同处理根际细菌所处的根际土壤微环境较为一致,并未引起不同处理间细菌群落多样性的改变[24-25]。冗余分析的结果发现,土壤铵态氮、硝态氮、有效磷、速效钾以及含水量对小麦茬土壤和西瓜茬土壤根际细菌群落的解释率分别为98.8% 和98.2%,其中小麦茬土壤中硝态氮的作用大于其他指标,而在西瓜茬土壤中铵态氮和硝态氮的作用相对更大,说明可利用氮源含量的高低在调控土壤根际细菌种群结构中起着至关重要的作用[12]。而各处理间南瓜根际土壤铵态氮与硝态氮含量差异不显著,这也是导致不同处理间细菌群落结构和多样性较稳定的原因之一。基于表 2的结果,小麦茬与西瓜茬南瓜根际土壤有效磷的含量存在差异,但这种差异不足以改变细菌群落结构组成,也侧面说明土壤有效磷不是调控细菌群落结构的限制因子。
3.2 根际土壤参与有机碳分解代谢细菌功能菌群的变化在门水平上对土壤中细菌群落结构分析发现,所有处理细菌群落结构中相对丰度较高的群体为变形菌门(Proteobacteria)、拟杆菌门(Bacterodidetes)和放线菌门(Actinobacteria),三者之和约达到90%,说明这3大门类的细菌种群具有较强的环境适应能力,无论是土壤环境的改变还是人为活动的影响,都不会影响细菌三大门类的主导地位[13, 26],但环境的差异会影响到其相对丰度的变化。大量研究认为,土壤可利用碳源含量及形态的改变都会影响参与碳分解的微生物种类和生物量[8, 18]。变形菌门在易分解有机碳组分的分解中占主导地位,拟杆菌门种群可以表达糖基水解酶参与纤维素和半纤维素分解的程度[27-28],而放线菌门属于主要参与难分解有机碳降解的门类[29]。在本研究中,高氮处理下,小麦茬南瓜根际土壤变形菌门的相对丰度低于西瓜茬土壤,但拟杆菌门和放线菌门的相对丰度高于西瓜茬土壤。这从侧面说明在高氮处理下,小麦茬南瓜根际土壤中易利用碳源的含量可能低于西瓜茬南瓜根际土壤[30],但参与难分解有机碳组分微生物种群的增加可能会加速小麦茬南瓜根际碳后期的分解速率。
基于图 4和Welch's T检验的结果,施氮水平对同种茬口南瓜根际土壤参与有机碳分解的关键功能基因丰度无显著影响,说明氮素含量虽然在细菌群落结构中的作用较大,但并不是限制有机碳分解的因子。但无论是在低氮处理还是在高氮处理,小麦茬土壤β-半乳糖苷酶基因(K01190)的丰度均显著高于西瓜茬土壤;另外,在低氮处理下,与西瓜茬南瓜根际土壤相比,小麦茬南瓜根际土壤显著提高了含有α-淀粉酶基因(K01176)丰度。研究表明β-半乳糖苷酶基因是编码β-半乳糖苷酶的基因,β-半乳糖苷酶是分解半纤维素的关键酶类,而α-淀粉酶基因编码形成α-淀粉酶,可以水解淀粉内部的α-1, 4-糖苷键,水解产物为糊精、低聚糖和单糖[30]。总之,这两种基因都是促进复杂有机物质分解转化成简单有机物质的关键,因而小麦茬土壤根际细菌可能会加速有机质分解过程[26]。
4 结论1) 不同茬口土壤和施氮水平并未改变南瓜根际土壤细菌Shannon多样性指数和群落结构,可利用氮源含量在调节南瓜根际土壤细菌群落结构中的作用至关重要。
2) 高氮处理下,小麦茬土壤根际细菌群落中参与难分解有机物质的种群丰度提升;且无论施氮高低,与西瓜茬土壤相比,小麦茬土壤的南瓜根际参与有机质分解的关键基因丰度显著提升,因此,与小麦茬土壤相比,西瓜茬土壤更有利于有机碳的固定。
| [1] |
Yang Z P, Yang W P, Li S C, et al. Variation of bacterial community diversity in rhizosphere soil of sole-cropped versus intercropped wheat field after harvest[J]. PLoS One, 2016, 11(3): e0150618 DOI:10.1371/journal.pone.0150618 (  0) 0) |
| [2] |
Chaparro J M, Badri D V, Vivanco J M. Rhizosphere microbiome assemblage is affected by plant development[J]. The ISME Journal, 2014, 8(4): 790-803 DOI:10.1038/ismej.2013.196 (  0) 0) |
| [3] |
Chaparro J M, Badri D V, Vivanco J M. Rhizosphere microbiome assemblage is affected by plant development[J]. The ISME Journal, 2014, 8(4): 790-803 DOI:10.1038/ismej.2013.196 (  0) 0) |
| [4] |
Huang X F, Chaparro J M, Reardon K F, et al. Rhizosphere interactions: root exudates, microbes, and microbial communities[J]. Botany, 2014, 92(4): 267-275 DOI:10.1139/cjb-2013-0225 (  0) 0) |
| [5] |
王晓婷, 陈瑞蕊, 井忠旺, 等. 水稻和小麦根际效应及细菌群落特征的比较研究[J]. 土壤学报, 2019, 56(2): 443-453 (  0) 0) |
| [6] |
Ibekwe A M, Poss J A, Grattan S R, et al. Bacterial diversity in cucumber (Cucumis sativus) rhizosphere in response to salinity, soil pH, and boron[J]. Soil Biology and Biochemistry, 2010, 42(4): 567-575 DOI:10.1016/j.soilbio.2009.11.033 (  0) 0) |
| [7] |
Morrissey J P, Dow J M, Mark G L, et al. Are microbes at the root of a solution to world food production?[J]. EMBO Reports, 2004, 5(10): 922-926 DOI:10.1038/sj.embor.7400263 (  0) 0) |
| [8] |
Mendes R, Kruijt M, de Bruijn I, et al. Deciphering the rhizosphere microbiome for disease-suppressive bacteria[J]. Science, 2011, 332(6033): 1097-1100 DOI:10.1126/science.1203980 (  0) 0) |
| [9] |
Bird J A, Herman D J, Firestone M K. Rhizosphere priming of soil organic matter by bacterial groups in a grassland soil[J]. Soil Biology and Biochemistry, 2011, 43(4): 718-725 DOI:10.1016/j.soilbio.2010.08.010 (  0) 0) |
| [10] |
杨珍, 戴传超, 王兴祥, 等. 作物土传真菌病害发生的根际微生物机制研究进展[J]. 土壤学报, 2019, 56(1): 12-22 (  0) 0) |
| [11] |
Six J, Frey S D, Thiet R K, et al. Bacterial and fungal contributions to carbon sequestration in agroecosystems[J]. Soil Science Society of America Journal, 2006, 70(2): 555-569 DOI:10.2136/sssaj2004.0347 (  0) 0) |
| [12] |
Prashar P, Kapoor N, Sachdeva S. Rhizosphere: its structure, bacterial diversity and significance[J]. Reviews in Environmental Science and Bio, 2014, 13(1): 63-77 (  0) 0) |
| [13] |
Li J G, Shen M C, Hou J F, et al. Effect of different levels of nitrogen on rhizosphere bacterial community structure in intensive monoculture of greenhouse lettuce[J]. Scientific Reports, 2016, 6: 25305 DOI:10.1038/srep25305 (  0) 0) |
| [14] |
Fierer N, Bradford M A, Jackson R B. Toward an ecological classification of soil bacteria[J]. Ecology, 2007, 88(6): 1354-1364 DOI:10.1890/05-1839 (  0) 0) |
| [15] |
Wang R Q, Xiao Y P, Lv F, et al. Bacterial community structure and functional potential of rhizosphere soils as influenced by nitrogen addition and bacterial wilt disease under continuous sesame cropping[J]. Applied Soil Ecology, 2018, 125: 117-127 DOI:10.1016/j.apsoil.2017.12.014 (  0) 0) |
| [16] |
Souza R C, Hungria M, Cantão M E, et al. Metagenomic analysis reveals microbial functional redundancies and specificities in a soil under different tillage and crop-management regimes[J]. Applied Soil Ecology, 2015, 86: 106-112 DOI:10.1016/j.apsoil.2014.10.010 (  0) 0) |
| [17] |
Giagnoni L, Pastorelli R, Mocali S, et al. Availability of different nitrogen forms changes the microbial communities and enzyme activities in the rhizosphere of maize lines with different nitrogen use efficiency[J]. Applied Soil Ecology, 2016, 98: 30-38 DOI:10.1016/j.apsoil.2015.09.004 (  0) 0) |
| [18] |
Wang X J, Tang C X, Severi J, et al. Rhizosphere priming effect on soil organic carbon decomposition under plant species differing in soil acidification and root exudation[J]. New Phytologist, 2016, 211(3): 864-873 DOI:10.1111/nph.13966 (  0) 0) |
| [19] |
Qiu H S, Zheng X D, Ge T, et al. Weaker priming and mineralisation of low molecular weight organic substances in paddy than in upland soil[J]. European Journal of Soil Biology, 2017, 83: 9-17 DOI:10.1016/j.ejsobi.2017.09.008 (  0) 0) |
| [20] |
(  0) 0) |
| [21] |
Derrien D, Plain C, Courty P E, et al. Does the addition of labile substrate destabilise old soil organic matter?[J]. Soil Biology and Biochemistry, 2014, 76: 149-160 DOI:10.1016/j.soilbio.2014.04.030 (  0) 0) |
| [22] |
(  0) 0) |
| [23] |
鲍士旦. 土壤农化分析(3版)[M].
中国农业出版社, 北京, 2000
(  0) 0) |
| [24] |
Langille M G I, Zaneveld J, Caporaso J G, et al. Predictive functional profiling of microbial communities using 16S rRNA marker gene sequences[J]. Nature Biotechnology, 2013, 31(9): 814-821 DOI:10.1038/nbt.2676 (  0) 0) |
| [25] |
Zhalnina K, Louie K B, Hao Z, et al. Dynamic root exudate chemistry and microbial substrate preferences drive patterns in rhizosphere microbial community assembly[J]. Nature Microbiology, 2018, 3(4): 470-480 DOI:10.1038/s41564-018-0129-3 (  0) 0) |
| [26] |
Brimecombe M J, De Leij F A A M, Lynch J M. Rhizodeposition and microbial populations//Pinton R, Veranini Z, Nannipieri P. The rhizosphere biochemistry and organic substances at the soil-plant interface. New York, USA: Taylor & Francis Group, 2007: 73-98.
(  0) 0) |
| [27] |
Zheng W, Zhao Z Y, Gong Q L, et al. Effects of cover crop in an apple orchard on microbial community composition, networks, and potential genes involved with degradation of crop residues in soil[J]. Biology and Fertility of Soils, 2018, 54(6): 743-759 DOI:10.1007/s00374-018-1298-1 (  0) 0) |
| [28] |
Wegner C E, Liesack W. Microbial community dynamics during the early stages of plant polymer breakdown in paddy soil[J]. Environmental Microbiology, 2016, 18(9): 2825-2842 DOI:10.1111/1462-2920.12815 (  0) 0) |
| [29] |
Wang G Z, Wang Y R, Yang P L, et al. Molecular detection and diversity of xylanase genes in alpine tundra soil[J]. Applied Microbiology and Biotechnology, 2010, 87(4): 1383-1393 DOI:10.1007/s00253-010-2564-9 (  0) 0) |
| [30] |
Sonia M T, Hafedh B, Abdennaceur H, et al. Studies on the ecology of actinomycetes in an agricultural soil amended with organic residues: II. Assessment of enzymatic activities of Actinomycetales isolates[J]. World Journal of Microbiology and Biotechnology, 2011, 27(10): 2251-2259 DOI:10.1007/s11274-011-0688-4 (  0) 0) |
| [31] |
Trivedi P, Anderson I C, Singh B K. Microbial modulators of soil carbon storage: integrating genomic and metabolic knowledge for global prediction[J]. Trends in Microbiology, 2013, 21(12): 641-651 DOI:10.1016/j.tim.2013.09.005 (  0) 0) |
2. National Agro-ecological System Observation and Research Station of Shangqiu, Shangqiu, Henan 476000, China
 2021, Vol. 53
2021, Vol. 53


