2. 中国科学院大学, 北京 100049
我国酸性土壤(pH < 5.5)面积为217.96万km2,占全国土地面积的22.7%,主要分布在南方热带和亚热带地区,具有丰富的光、热、水、土和生物资源[1]。近年来,由于氨态氮肥过量施用和工业化导致的酸沉降等人为活动强度的不断增加,土壤酸化程度在加剧,酸化面积在不断扩大[2],严重限制了土壤的生产潜力,并对生态环境造成严重危害。
土壤微生物是土壤生态系统的重要组成部分,主导的硝化作用不仅是元素生物地球化学循环的重要组成部分[3-5],也是土壤酸化的重要驱动者[2],因此越来越受到研究人员的重视。经典硝化作用分为两步,第一步是氨氧化过程,即将氨(NH3)氧化为亚硝酸根(NO2-);第二步是亚硝酸盐氧化过程,即将NO2-氧化为硝酸根(NO3-);其中氨氧化过程是硝化作用的限速步骤,在氮循环中扮演着重要角色[6]。上百年来,氨氧化细菌(ammonia-oxidizing bacteria, AOB)一直被认为是土壤氨氧化过程的主要驱动者[7]。但是,2005年Könneke等人[8]在海洋中分离出一株可以进行氨氧化过程的泉古菌,其属于化能无机自养代谢氨氧化古菌(ammonia-oxidizing archaea, AOA)。进一步研究证实,AOB和AOA都参与氨氧化过程,其贡献能力取决于特定的环境和微生物类群组成[9-10]。例如,AOA和AOB对土壤pH和底物NH3的敏感性不同[11]。Wang等[12]研究发现,由于AOA和AOB在4种环境(稻田、河口、浅滩和湿地)中群落组成明显不同,它们的氨氧化活性差异较大。基于目前对土壤酸化的高度重视,研究认为AOA,尤其是奇古菌和泉古菌,是酸性土壤氨氧化过程主要驱动者,原因在于其能够较好地适应酸性土壤的环境特点,特别是较低土壤pH[11]。然而,一些研究发现,酸性土壤中AOB的某些种属类群(如Neutrophilic bacteria和Nitrosospira cluster 8a等)也可能发挥重要作用[13-14]。即使在相似的土壤pH下,一些特定的AOA或AOB类群也表现较大差异的功能。例如,AOA中Group 1.1a的古菌微生物类群在酸性水稻土壤(pH = 4.8)和森林土壤(pH = 4.2)氨氧化过程中发挥重要的作用,而AOB中Nitrosospira cluster 3-like的微生物类群主要参与酸性旱作土壤(pH = 4.7)中的氨氧化过程[15]。可见,由于受多种因素的影响,AOB和AOA对酸性土壤氨氧化过程的相对贡献仍存在较大争议,发挥作用机理需要进一步阐明。
早期研究认为酸性土壤硝化作用很弱,但是目前很多证据证明了在某些特定的酸性土壤中,其硝化作用也很强烈。基于本实验室前期对我国南方酸性红壤区采集的51个土壤样品[16],发现样品间硝化作用潜能存在较大差异,本研究选择其中两个pH相近且硝化能力差异较大的土壤样品,期望通过测定氨氧化微生物的丰度和群落组成,分析AOA和AOB在酸性土壤硝化过程中所扮演的角色,并探索发挥作用的机制。
1 材料与方法 1.1 土壤样品培养试验所用酸性土壤样品分别采自江西鹰潭柑桔园和旱地农田,根据我们前期研究,依次命名为JX-3和JX-7[16]。两个土壤样品起始基本理化性质见表 1。
|
|
表 1 供试土壤基本理化性质 Table 1 Physicochemical properties of tested soil |
称取50 g过2 mm筛的风干土放于250 ml培养瓶中,加去离子水至最大持水量的40%,用塑料膜将培养瓶封口。于28 ℃环境中恒温黑暗预培养7 d后,按照N 250 mg/kg干土加入硫酸铵,最后将土壤水分含量调至最大持水量的60%,用塑料膜将培养瓶封口,于28 ℃环境中恒温黑暗培养。培养期间每隔1 d采用称重法补水1次,使土壤水分保持基本不变。培养开始后的0 d和50 d分别取样。
土壤pH采用风干土测定,用pH计(Mettler Toledo FE20, 上海, 中国)在土水比为1:2.5的条件下测定;土壤铵态氮和硝态氮采用鲜土测定,2 mol/L氯化钾浸提后,用流动分析仪(San++, Skalar, Holland)测定。
1.3 土壤DNA提取和基因丰度测定称取0.50 g新鲜土壤样品,采用Fast®DNA SPIN Kit (MP Biomedicals, CA, USA)提取土壤DNA,并采用PowerClean®DNA Clean-up Kit (MoBio, CA, USA)纯化土壤DNA,具体操作步骤按照试剂盒说明书进行。每个土壤样品连续提取两次DNA,将其混合在一起作为一个土壤样品DNA。
在LightCycler 480 Real-Time定量PCR仪(Roche Diagnostics, Mannheim, Germany)上进行实时荧光定量PCR,测定与氨氧化过程相关基因(古菌amoA和细菌amoA)的丰度。氨氧化微生物功能基因古菌amoA和细菌amoA扩增引物分别为Arch-amoA-F (5’-STAATGGTCTGGCTTAGACG-3’)/Arch-amoA-R(5’-GCGGCCATCCATCTGTATGT-3’)[8]和Bac-amoA-F (5’-GGGGTTTCTACTGGTGGT-3’) / Bac-amoA-R (5’-CCCCTCKGSAAAGCCTTCTTC-3’)[17]。定量PCR反应体系为10 μl,包括5 μl TB GreenTM Premix Ex TaqTM (2x),上下引物各0.4 μl,2 μl DNA模板(稀释20倍),2.2 μl超纯无菌水。定量PCR反应条件如下:95 ℃预变性30 s,95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸30 s,40个循环。每个土壤DNA样品扩增3次。对含有目的基因的质粒进行10倍梯度稀释,于同一条件下进行qPCR,得到标准曲线。古菌amoA和细菌amoA的qPCR扩增效率分别为94.0%(R2= 0.999)和102.5% (R2 = 0.991)。
1.4 AOA和AOB中amoA基因高通量测序以提取的土壤DNA为模板,采用Arch-amoA-F/R和Bac-amoA-F/R引物对目的片段进行PCR扩增。获得PCR产物后,对其进行纯化、建立文库和文库质检,最后利用454 GS FLX+仪器进行测序。所有原始下机数据首先运用QIIME软件识别疑问序列并去除[18];随后通过QIIME软件调用USEARCH检查并剔除嵌合体序列。剩余的高质量序列利用UCLUST序列比对工具按照97% 的相似度划分操作分类单元(operational taxonomic unit, OTU),每个OTU中数量最多的序列被选为该OTU的代表序列[19]。最后将所有代表序列与NCBI GenBank中具有分类信息的序列相比对,获得每个OTU所对应的分类学信息。
1.5 数据处理与统计分析土壤pH变化量是培养开始后50 d的土壤pH减去0 d的pH。土壤硝化速率是单位时间内土壤硝态氮含量的增加量,即培养开始后50 d的土壤硝态氮含量与0 d土壤硝态氮含量的差值除以50 d[20]。
AOA和AOB前20位OTUs的绝对丰度是高通量测序数据中相对丰度前20位OTUs的相对丰度乘以对应基因的总丰度。
数据处理采用Microsoft Excel 2019,并采用SPSS 22.0进行统计分析。采用单因素方差分析检验AOA和AOB中amoA基因丰度的差异显著性。多重比较采用邓肯(Ducan)法,差异显著性水平为0.05。采用独立样本t-检验分析供试土壤之间土壤pH变化量、硝化速率,同一供试土壤下培养开始后0 d和50 d之间微生物前20位OTUs绝对丰度差异显著性。应用R语言vegan包对微生物群落结构进行主成分分析(principal component analysis, PCA)。作图使用Origin 2019软件。
2 结果 2.1 土壤pH变化量和硝化速率培养50 d后,JX-3土壤pH基本稳定不变,而JX-7土壤pH显著下降(图 1A)。两个土壤硝化速率存在显著差异,即JX-7 (0.78 mg/(kg·d)) >JX-3 (0.11 mg/(kg·d)) (图 1B)。可见,土壤硝化作用越强,土壤酸化越明显,但不同土壤间存在较大差异。
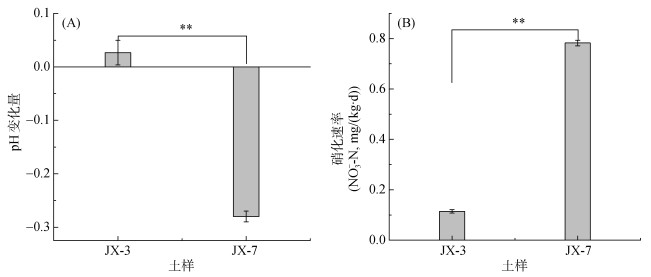
|
(** 表示土壤pH变化量或硝化速率在不同供试土壤间差异达P < 0.01显著水平) 图 1 培养50 d后土壤pH变化量(A)和硝化速率(B) Fig. 1 Soil pH changes (A) and nitrification rates (B) after 50 d incubation |
两个土壤本身AOB的amoA基因丰度存在显著差异,但AOA并没有显著差异(图 2)。培养50 d后,JX-7土壤AOA的amoA基因丰度显著增加(图 2A),但是JX-3无显著增加。两个土壤AOB的amoA基因丰度也均显著增加,但样品间无明显差异(图 2B)。可以推测,AOA可能是硝化作用的主要驱动者。
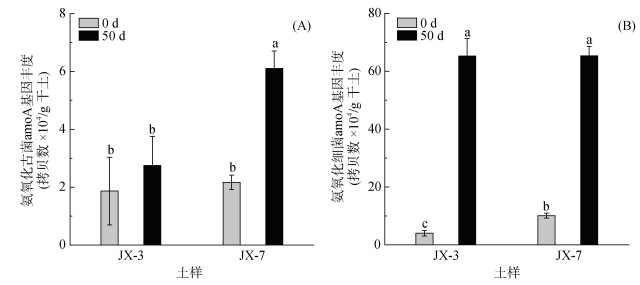
|
(图中小写字母不同表示土壤AOA或AOB的amoA基因丰度在不同土壤间差异达显著水平(P < 0.05)) 图 2 土壤AOA (A)和AOB (B)的amoA基因丰度 Fig. 2 Soil archaeal amoA (A) and bacterial amoA (B) genes abundances |
土壤氨氧化微生物群落组成分析结果(图 3)显示,酸性土壤AOA群落主要的菌属是Nitrososphaera和Candidatus Nitrosocosmicus;AOB群落主要的菌属是Nitrosospira、Nitrosovibrio、Nitrosomonas和Sphingopyxis。这些菌属相对丰度的占比在样品间存在差异。
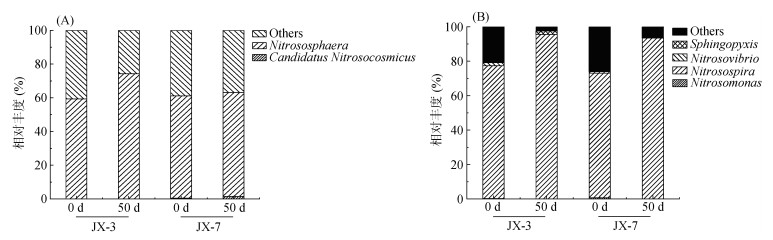
|
图 3 土壤AOA (A)和AOB (B)微生物群落组成 Fig. 3 Soil AOA (A) and AOB (B) microbial community compositions |
土壤AOA群落结构分析结果(图 4A)显示,主成分1和主成分2分别解释变量方差的49.83% 和29.09%;JX-7和JX-3在主成分轴1上有很好的分异,但是这两个土壤中AOA群落在培养前后(0 d和50 d)没有显著分异。土壤AOB群落结构分析结果(图 4B)显示,主成分1和主成分2分别解释变量方差的89.55% 和9.13%;AOB群落在JX-3和JX-7间没有分异,但这两个土壤AOB群落在培养前后(0 d和50 d)表现出很好的分异。
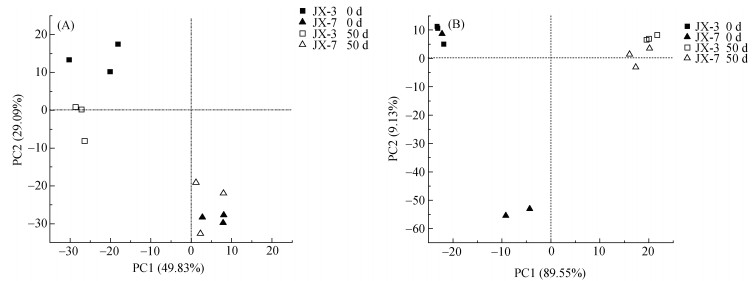
|
图 4 土壤AOA(A)和AOB (B)微生物群落结构主成分分析 Fig. 4 Principal component analyses (PCA) for soil AOA (A) and AOB (B) microbial communities |
土壤AOA绝对丰度前20位OTUs分析结果(表 2)显示,JX-7中otu1138和otu563绝对丰度在培养后显著增加,而JX-3中没有显著变化的OTUs。土壤AOB绝对丰度前20位OTUs分析结果(表 3)显示,JX-3中otu1618、otu487、otu2315、otu1460、otu1552、otu847、otu830和otu388绝对丰度在培养前后(0 d和50 d)存在显著变化;JX-7土壤中otu1618绝对丰度在培养前后(0 d和50 d)存在显著变化。
|
|
表 2 土壤AOA前20位OTUs的绝对丰度 Table 2 Absolute abundances of top 20 OTUs of soil AOA |
|
|
表 3 土壤AOB前20位OTUs的绝对丰度 Table 3 Absolute abundances of top 20 OTUs of soil AOB |
氨氧化微生物调控的硝化作用是土壤酸化加速的主要驱动者,且硝化速率越快,土壤pH下降越多[21]。本研究中JX-7土壤硝化速率高于JX-3,相应的土壤pH下降越多(图 1)。有学者对我国酸性土壤中AOA和AOB群落与土壤硝化潜势相关性进行了调查,发现氨氧化过程主要由AOA驱动[11-22]。但也有大量关于AOB主导酸性土壤硝化作用的报道[13-15]。而本研究通过分析氨氧化微生物丰度特征,发现所研究的酸性土壤硝化作用由AOA主导发生(图 2和图 4)。虽然两个土壤AOA和AOB丰度在培养前后都有所变化,但AOA的变化更符合与硝化速率的关系。明显的,JX-3土壤经过50 d的培养后,其硝化作用很弱,AOA丰度没有显著变化,但AOB丰度显著增加。推测供试土壤只是提供了AOB生长的环境,但并没有刺激其硝化能力[22]。类似的,He等[23]报道,施加肥料后酸性土壤AOB丰度增加,但AOB并不是硝化作用的主要驱动者。这可能是由于土壤pH是影响AOB发挥硝化能力的一个主要因素[23-24]。
Gubry-Rangin等[24]研究结果表明,AOA群落结构改变与酸性土壤硝化作用的发生密切相关。然而本研究结果显示,两个土壤AOA群落组成在培养前后没有显著分异(图 4A),暗示AOA不是主要通过改变其整体群落结构来参与硝化作用。而某些AOA的OTUs绝对丰度在培养前后有显著增加(表 2),说明AOA发挥其氨氧化功能主要是通过个别微生物。有学者研究表明,AOA群落中Thaumarchaea group 1.1a和1.1b与酸性土壤硝化作用密切相关,且与土壤pH和底物NH3浓度有关[12, 25-27]。He等[23]和Leininger等[27]研究发现,泉古菌Crenarchaeote clone是提高酸性土壤硝化作用的关键氨氧化微生物。本研究中,JX-7土壤AOA中otu1138和otu563绝对丰度的显著增加说明,供试土壤AOA中Nitrososphaera属的一些个别微生物种类主要参与土壤硝化过程。
前期研究中,所有可培养的Nitrososphaera最适生长pH均为中性(pH 6.0~8.0)[28-29]。分子学研究表明,在中性和碱性土壤AOA中Nitrososphaera是优势属[20, 30-31]。然而,越来越多的研究发现,酸性土壤氨氧化作用与Nitrososphaera密切相关[32-33]。Lin等[14]和Zhang等[32]分别对酸性土壤氨氧化微生物丰度进行了调查,发现AOA的绝大部分序列属于Nitrososphaera。Wang等[33]利用稳定同位素核酸标记探针技术,研究表明Nitrososphaera是酸性土壤上发挥氨氧化功能的主要AOA类群,且其在酸性环境中是化能自养型微生物。这可能是由于AOA含有编码酸碱平衡的基因[34]以及AOA对底物的亲和力高于AOB[33];或者可能是由于该菌属微生物在适应酸性环境过程中具备了某种代谢能力以缓解低pH对其毒害作用[34-35]。同时,这些结果也说明了酸性土壤上氨氧化过程的复杂性,需要我们进一步阐明。
本研究对初始pH相近的两个酸性土壤中硝化作用及氨氧化微生物的丰度特征和群落结构的研究结果表明,AOA应该在所研究的酸性土壤硝化作用中扮演着关键角色,AOA调控硝化功能的实现主要是通过增加个别微生物物种的丰度而不是改变整个群落组成。
| [1] |
赵其国. 我国红壤的退化问题[J]. 土壤, 1995, 27(6): 281-285 (  0) 0) |
| [2] |
Guo J H, Liu X J, Zhang Y, et al. Significant acidification in major Chinese croplands[J]. Science, 2010, 327(5968): 1008-1010 DOI:10.1126/science.1182570 (  0) 0) |
| [3] |
Gruber N, Galloway J N. An Earth-system perspective of the global nitrogen cycle[J]. Nature, 2008, 451(7176): 293-296 DOI:10.1038/nature06592 (  0) 0) |
| [4] |
Luo G W, Friman V P, Chen H, et al. Long-term fertilization regimes drive the abundance and composition of N-cycling-related prokaryotic groups via soil particle-size differentiation[J]. Soil Biology and Biochemistry, 2018, 116: 213-223 DOI:10.1016/j.soilbio.2017.10.015 (  0) 0) |
| [5] |
Pereg L, Morugán-Coronado A, McMillan M, et al. Restoration of nitrogen cycling community in grapevine soil by a decade of organic fertilization[J]. Soil and Tillage Research, 2018, 179: 11-19 DOI:10.1016/j.still.2018.01.007 (  0) 0) |
| [6] |
Daims H, Lücker S, Wagner M. A new perspective on microbes formerly known as nitrite-oxidizing bacteria[J]. Trends in Microbiology, 2016, 24(9): 699-712 DOI:10.1016/j.tim.2016.05.004 (  0) 0) |
| [7] |
Purkhold U, PommereningRöser A, Juretschko S, et al. Phylogeny of all recognized species of ammonia oxidizers based on comparative 16S rRNA and amoA sequence analysis: implications for molecular diversity surveys[J]. Applied and Environmental Microbiology, 2000, 66(12): 5368-5382 DOI:10.1128/AEM.66.12.5368-5382.2000 (  0) 0) |
| [8] |
Könneke M, Bernhard A E, de la Torre J R, et al. Isolation of an autotrophic ammonia-oxidizing marine archaeon[J]. Nature, 2005, 437(7058): 543-546 DOI:10.1038/nature03911 (  0) 0) |
| [9] |
Carey C J, Dove N C, Beman J M, et al. Meta-analysis reveals ammonia-oxidizing bacteria respond more strongly to nitrogen addition than ammonia-oxidizing Archaea[J]. Soil Biology and Biochemistry, 2016, 99: 158-166 DOI:10.1016/j.soilbio.2016.05.014 (  0) 0) |
| [10] |
Kuypers M M M, Marchant H K, Kartal B. The microbial nitrogen-cycling network[J]. Nature Reviews. Microbiology, 2018, 16(5): 263-276 DOI:10.1038/nrmicro.2018.9 (  0) 0) |
| [11] |
Li Y Y, Chapman S J, Nicol G W, et al. Nitrification and nitrifiers in acidic soils[J]. Soil Biology and Biochemistry, 2018, 116: 290-301 DOI:10.1016/j.soilbio.2017.10.023 (  0) 0) |
| [12] |
Wang C, Tang S Y, He X J, et al. The abundance and community structure of active ammonia-oxidizing Archaea and ammonia-oxidizing bacteria shape their activities and contributions in coastal wetlands[J]. Water Research, 2020, 171: 115464 DOI:10.1016/j.watres.2019.115464 (  0) 0) |
| [13] |
Huang X R, Zhao J, Su J, et al. Neutrophilic bacteria are responsible for autotrophic ammonia oxidation in an acidic forest soil[J]. Soil Biology and Biochemistry, 2018, 119: 83-89 DOI:10.1016/j.soilbio.2018.01.016 (  0) 0) |
| [14] |
Lin Y X, Ye G P, Luo J F, et al. Nitrosospira cluster 8a plays a predominant role in the nitrification process of a subtropical ultisol under long-term inorganic and organic fertilization[J]. Applied and Environmental Microbiology, 2018, 84(18): e01031-e01018 (  0) 0) |
| [15] |
Dai S Y, Liu Q, Zhao J, et al. Ecological niche differentiation of ammonia-oxidising Archaea and bacteria in acidic soils due to land use change[J]. Soil Research, 2018, 56(1): 71 DOI:10.1071/SR16356 (  0) 0) |
| [16] |
Zheng M M, Wang C, Li W X, et al. Soil nutrients drive function and composition of phoC-harboring bacterial community in acidic soils of Southern China[J]. Frontiers in Microbiology, 2019, 10: 2654 DOI:10.3389/fmicb.2019.02654 (  0) 0) |
| [17] |
Rasche F, Knapp D, Kaiser C, et al. Seasonality and resource availability control bacterial and archaeal communities in soils of a temperate beech forest[J]. The ISME Journal, 2011, 5(3): 389-402 DOI:10.1038/ismej.2010.138 (  0) 0) |
| [18] |
Caporaso J G, Kuczynski J, Stombaugh J, et al. QIIME allows analysis of high-throughput community sequencing data[J]. Nature Methods, 2010, 7(5): 335-336 DOI:10.1038/nmeth.f.303 (  0) 0) |
| [19] |
Edgar R C. Search and clustering orders of magnitude faster than BLAST[J]. Bioinformatics, 2010, 26(19): 2460-2461 DOI:10.1093/bioinformatics/btq461 (  0) 0) |
| [20] |
蔡祖聪, 赵维. 土地利用方式对湿润亚热带土壤硝化作用的影响[J]. 土壤学报, 2009, 46(5): 795-801 (  0) 0) |
| [21] |
He J Z, Hu H W, Zhang L M. Current insights into the autotrophic thaumarchaeal ammonia oxidation in acidic soils[J]. Soil Biology and Biochemistry, 2012, 55: 146-154 DOI:10.1016/j.soilbio.2012.06.006 (  0) 0) |
| [22] |
Hu H W, Xu Z H, He J Z. Ammonia-oxidizing Archaea play a predominant role in acid soil nitrification[J]. 2014, 125: 261-302.
(  0) 0) |
| [23] |
He J Z, Shen J P, Zhang L M, et al. Quantitative analyses of the abundance and composition of ammonia-oxidizing bacteria and ammonia-oxidizing Archaea of a Chinese upland red soil under long-term fertilization practices[J]. Environmental Microbiology, 2007, 9(9): 2364-2374 DOI:10.1111/j.1462-2920.2007.01358.x (  0) 0) |
| [24] |
Gubry-Rangin C, Nicol G W, Prosser J I. Archaea rather than bacteria control nitrification in two agricultural acidic soils[J]. FEMS Microbiology Ecology, 2010, 74(3): 566-574 DOI:10.1111/j.1574-6941.2010.00971.x (  0) 0) |
| [25] |
Offre P, Prosser J I, Nicol G W. Growth of ammonia-oxidizing Archaea in soil microcosms is inhibited by acetylene[J]. FEMS Microbiology Ecology, 2009, 70(1): 99-108 DOI:10.1111/j.1574-6941.2009.00725.x (  0) 0) |
| [26] |
Zhang L M, Offre P R, He J Z, et al. Autotrophic ammonia oxidation by soil thaumarchaea[J]. PNAS, 2010, 107(40): 17240-17245 DOI:10.1073/pnas.1004947107 (  0) 0) |
| [27] |
Leininger S, Urich T, Schloter M, et al. Archaea predominate among ammonia-oxidizing prokaryotes in soils[J]. Nature, 2006, 442(7104): 806-809 DOI:10.1038/nature04983 (  0) 0) |
| [28] |
Kim J G, Jung M Y, Park S J, et al. Cultivation of a highly enriched ammonia-oxidizing archaeon of thaumarchaeotal group I.1b from an agricultural soil[J]. Environmental Microbiology, 2012, 14(6): 1528-1543 DOI:10.1111/j.1462-2920.2012.02740.x (  0) 0) |
| [29] |
Gubry-Rangin C, Hai B, Quince C, et al. Niche specialization of terrestrial archaeal ammonia oxidizers[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(52): 21206-21211 DOI:10.1073/pnas.1109000108 (  0) 0) |
| [30] |
Pester M, Rattei T, Flechl S, et al. amoA-based consensus phylogeny of ammonia-oxidizing Archaea and deep sequencing of amoA genes from soils of four different geographic regions[J]. Environmental Microbiology, 2012, 14(2): 525-539 DOI:10.1111/j.1462-2920.2011.02666.x (  0) 0) |
| [31] |
Huang R, Wu Y C, Zhang J B, et al. Nitrification activity and putative ammonia-oxidizing Archaea in acidic red soils[J]. Journal of Soils and Sediments, 2012, 12(3): 420-428 DOI:10.1007/s11368-011-0450-4 (  0) 0) |
| [32] |
Zhang M M, Alves R J E, Zhang D D, et al. Time-dependent shifts in populations and activity of bacterial and archaeal ammonia oxidizers in response to liming in acidic soils[J]. Soil Biology and Biochemistry, 2017(52): 77-89 (  0) 0) |
| [33] |
Wang B Z, Zheng Y, Huang R, et al. Active ammonia oxidizers in an acidic soil are phylogenetically closely related to neutrophilic archaeon[J]. Applied and Environmental Microbiology, 2014, 80(5): 1684-1691 DOI:10.1128/AEM.03633-13 (  0) 0) |
| [34] |
Lehtovirta-Morley L E, Sayavedra-Soto L A, Gallois N, et al. Identifying potential mechanisms enabling acidophily in the ammonia-oxidizing archaeon "candidatus nitrosotalea devanaterra"[J]. Applied and Environmental Microbiology, 2016, 82(9): 2608-2619 DOI:10.1128/AEM.04031-15 (  0) 0) |
| [35] |
Lehtovirta-Morley L E, Ge C R, Ross J, et al. Characterisation of terrestrial acidophilic archaeal ammonia oxidisers and their inhibition and stimulation by organic compounds[J]. FEMS Microbiology Ecology, 2014, 89(3): 542-552 DOI:10.1111/1574-6941.12353 (  0) 0) |
2. University of Chinese Academy of Sciences, Beijing 100049, China
 2021, Vol. 53
2021, Vol. 53


