2. 中国科学院大学, 北京 100049;
3. 南京林业大学林学院, 南京 210008
水稻是人类所需的三大主要粮食作物之一,据估计,水稻养活了全世界50%、亚洲85% 的人口[1]。随着全世界人口不断增长,粮食产量也需要进一步提高。水稻产量的提升受到稻田土壤缺氮的限制,为补充农田中的氮素,大量的化学氮肥被施入,导致了高浪费和严重的环境污染[2]。而固氮微生物自身固定的氮素能被植物有效利用,且不产生环境污染问题。自身固氮也是许多生态系统氮的主要输入途径,维持着全球植物的生产力[3]。在Haber-Bosch发明工业合成氮肥以前,水稻田中的氮素来源主要是生物固氮,所以生物固定的氮被认为是谷物生产中潜在且有吸引力的替代氮源[4]。
种植水稻甚至种植不同品种的水稻均会改变稻田土壤微生物生长环境,从而改变稻田微生物群落结构。固氮菌作为稻田中广泛存在的细菌,其群落结构及其固氮能力也会受到种植水稻的影响[5-6]。如Yoshida和Ancajas[7]通过乙炔还原法研究发现,无论是在雨季还是旱季,无论淹水还是干旱稻田,整个生长季种水稻处理比不种水稻处理生物固氮量均高出2倍左右。Bei等[8]通过15N2直接标记法研究发现,培养70 d种水稻处理15N固定量均高于不种水稻处理。不同水稻品种也会影响稻田生物固氮,研究发现杂交稻品种(IIY)比自交稻品种(W23)对稻田生物固氮促进作用更大[9-10]。Ito等[11]用两种水稻品种进行水培,发现其固氮活性相差近1倍。可见,水稻种植与否及其品种对稻田固氮量有很大影响,因此在估算稻田固氮潜力时,必须要考虑水稻种植情况及其品种。而在1年时间内,撂荒和水稻生长季稻田土壤对生物固氮量的贡献分别是多少,目前还不清楚。
固氮量由活跃固氮微生物的数量以及固氮活性决定。编码固氮酶铁蛋白亚基的nifH基因通常被用来研究固氮微生物和评估固氮潜力[12]。前期研究大多数基于nifH DNA基因进行分析,这些研究提供了固氮微生物潜在固氮活性的很多有价值的信息[13-15]。但是研究发现,nifH基因拷贝数与固氮活性并没有显著的相关性[16-17]。固氮菌基因拷贝数仅能代表土壤中固氮菌的数量,不能直接说明其参与了固氮过程。而从土壤中直接提取RNA,再反转录成cDNA,然后对cDNA的nifH基因进行定量扩增,得出nifH转录拷贝数被认为更能代表固氮微生物活性[15, 18]。
研究稻田生物固氮,首先要对稻田固氮量进行测定。目前在水稻土壤中测定固氮量常用的方法有:乙炔还原法[19-20]、15N2自然丰度法[21]、15N2同位素稀释法[20]、氮素平衡法[22]等非直接的测定方法,这些方法由于缺少准确的转换因子,或者要考虑氮源、氮损失等各种因素而存在很大的不确定性。测定固氮量最直接的方法是15N2气体标记法,该方法不需要转换系数,不受外来氮源干扰,从而能够准确测定固氮量[23-24]。该方法难点在于要长时间保持系统密闭性,且为了反映田间水稻生长的真实环境,需要实现标记箱内CO2浓度、O2浓度、温度和光照条件等环境与大田环境相似;另外,高纯度高丰度15N2气体价格十分昂贵,这些都限制了该方法在自然环境条件下的广泛应用[25]。
本试验利用15N2标记密闭生长箱系统[8],在田间环境下,自插秧后进行15N2直接标记整个生长季,测定下位砂姜土在种水稻和不种水稻情况下的生物固氮量,并对土壤DNA和cDNA的nifH基因进行实时荧光定量PCR扩增,测定固氮菌数量以及固氮酶基因表达量,从而计算其固氮活性。
1 材料与方法 1.1 试验地描述和土壤理化性质试验地位于江苏省扬州市小纪镇马凌村良种场(32°35′5″ N,119°42′0″ E),该地区气候条件为典型亚热带季风气候,年降水量1 000 mm左右,年蒸发量大于1 100 mm,年均气温15 ℃,年日照时间大于2 000 h,年无霜220 d。试验地种植模式为稻麦轮作,土壤类型为下位砂姜土发育的简育水耕人为土[26]。2017年水稻季后,随机取试验地耕作层(0 ~ 15 cm)土壤,风干过4 mm筛备用。土壤pH为6.1,砂粒质量占比7.4%,粉粒质量占比78.0%,黏粒质量占比14.6%。更多土壤理化性质详见Wang等[27]。
1.2 田间15N2标记盆栽试验田间15N2标记盆栽试验时间为2018年7月23日至2018年10月23日。试验设种水稻和不种水稻、标记和不标记4个处理,每个处理3次重复。标记的3个重复分别放在3个标记箱中,即每个箱中只放置1个重复。不标记处理的3个重复放在箱外进行(作为自然丰度的对照处理,用于计算标记箱内样品和空气的15N原子百分超)。3个15N2标记密闭生长箱(长×宽×高= 1 160 mm× 680 mm× 890 mm)平行放置于田间,3个箱体间隔为1 m。用40 L 15N2 (99 atom% 15N)置换箱内40 L空气,使得3个箱子内的15N丰度为9% 左右。15N2根据Ohyama和Kumazawa[28]描述的方法进行制备和纯化。箱内温度设定为外界温度正负1℃,箱内CO2浓度设定为400 μl/L ± 20 μl/L。箱内由于植物光合作用产生的多余氧气通过除氧剂去除,使得箱内的O2浓度维持在21% 左右。更多关于15N2标记密闭生长箱的设计及其控制系统的信息详见Bei等[8]。
试验时,将土壤样品风干过筛(< 5 mm)后称取1.3 kg装入盆(长×宽×高= 9 cm × 9 cm × 20 cm)中15 cm高;在盆中浇水至高于土面1 ~ 2 cm,浸泡1周后,每盆施116.37 mg的KH2PO4和32.55 mg KCl(相当于P2O575 kg/hm2和K2O 75 kg/hm2),不施氮肥;然后从大田移栽两个月大的水稻苗(Oryza sativa L.,品种为武运粳23)至盆中,每盆种1棵。所有盆栽均放置在户外1周,确保秧苗存活后,再放入标记箱内。整个生长季保持盆内1 cm高水层(烤田期除外)。每周采集1次箱内气体,用于观测箱内15N2变化情况。
1.3 样品采集及15N2丰度测定经过90 d的15N2持续标记后,打开标记箱,采集土壤和植株样品。水稻植株分两部分采集:地上部(茎叶穗)和地下部(根)。每个盆栽的土样分三层(0 ~ 1、1 ~ 5、5 ~ 15 cm)采集。一部分土样作为鲜土放入液氮后,带回实验室保存在-80 ℃冰箱用作后续分子实验;另一部分土样风干、磨细、过筛(Retsch MM 400 mixer mill,Retsch, Haan, Germany),用元素分析仪和同位素质谱联用仪测定其全氮含量和15N丰度(Thermo Finnigan Flash EA-Delta V IRMS联用仪)。
1.4 土壤DNA及RNA提取使用RNeasy® PowerSoil® Total RNA Kit(Qiagen) 试剂盒提取土壤总RNA,用RNeasy® MinElute® Cleanup(Qiagen)对总RNA进一步纯化,用Prime ScriptTM RT reagent Kit with gDNA Eraser(Takara)试剂盒对提取的总RNA进行DNA进一步去除和反转录,反转录的cDNA保存到-80 ℃冰箱备用。使用RNeasy® PowerSoil® DNA Elution Kit(Qiagen) 试剂盒提取土壤总DNA,提取好的土壤DNA样品用超微量紫外可见分光光度计(NanoVue Plus,USA)测定纯度及浓度,然后保存于-20 ℃备用。
1.5 实时荧光定量PCR (qPCR)对样品的DNA及cDNA的nifH基因用引物polF (TGCGAYCCSAARGCBGACTC) / polR (ATSGCCAT CATYTCRCCGGA) 进行定量PCR扩增,程序为:95 ℃,10 min;35循环(95 ℃,30 s;58 ℃,35 s;72℃,45 s);72℃,10 min。
1.6 数据处理与分析1) 计算公式。从空气中固定的氮素占整个水稻-土壤系统全部氮素的比例(%N fixation)可以通过下式计算:
| $ \% {\rm{N}}\;{\rm{fixation}} = \frac{{{\rm{atom}}\;\% {\;^{15}}{{\rm{N}}_{{\rm{excess}}}}\;{\rm{samples}}}}{{{\rm{atom}}\;\% {\;^{15}}{{\rm{N}}_{{\rm{excess}}}}\;{\rm{gas}}}} \times 100 $ |
式中:atom% 15Nexcess samples为标记箱内样品的15N原子百分超;atom%15Nexcess gas为标记箱内空气中的15N原子百分超。atom% 15Nexcess samples= 15N2标记箱内样品的15N丰度(15N atom%)- 箱外对照样品的15N丰度(15N atom%)。atom%15Nexcess gas= 15N2标记箱内空气的15N丰度-箱外空气的15N丰度。
整个水稻-土壤系统的固氮量=样品总氮(N mg/盆)× %N fixation
固氮活性=固氮量(mg)/nifH RNA基因拷贝数
2) 数据统计分析。文中所有数据均采用SPSS 20.0统计软件作单因素方差分析,用Turkey法进行多重比较。
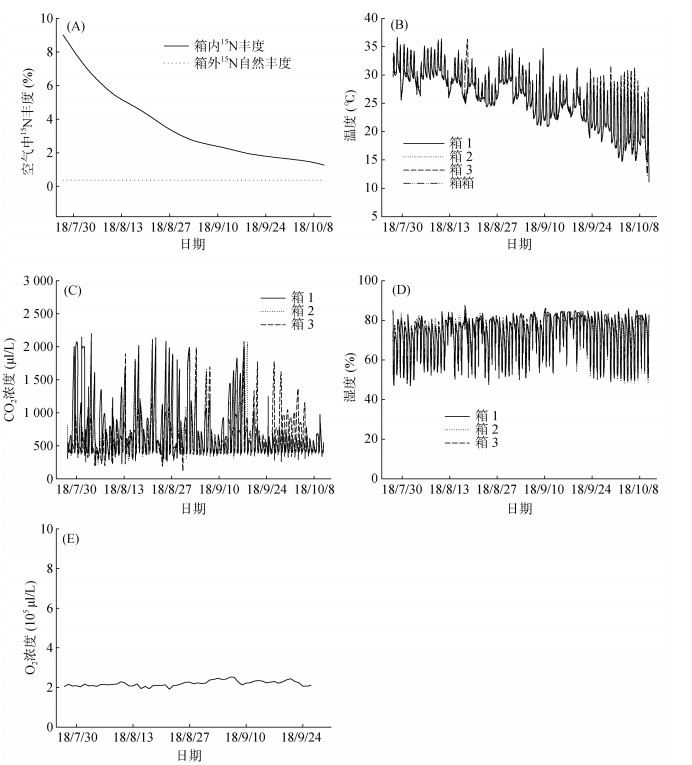
2 结果 2.1 15N2标记箱田间运行情况3个15N2标记密闭生长箱平行置于水稻田(间隔1 m),标记时间从2018年7月23日至2018年10月23日,自水稻生长的苗期至成熟期。标记箱内气体中15N2丰度最高为9.37%,最低为0.80%,平均为3.73%(图 1A)。自动控制密闭标记生长箱内的温度基本与田间温度保持一致(图 1B)。箱内CO2浓度在经过一晚上水稻呼吸作用累积后,早上最高达约2 000 μl/L,在经过大约1 h的光合作用消耗后,浓度逐渐降至设定的400 μl/L ± 20 μl/L范围,随后白天一直维持在这个范围(图 1C)。箱内相对湿度在50% ~ 80%(图 1D)。箱内光合作用产生的O2通过定期测量和利用除氧剂(铁粉)消除,浓度始终维持在2.1×105 μl/L (图 1E)。
|
(A:标记箱内外15N2丰度的动态变化,B:标记箱及箱外田间空气温度小时变化值,C:白天标记箱内CO2浓度小时变化值,D:白天标记箱内相对湿度小时变化值,E:标记箱内O2浓度变化值) 图 1 整个水稻生长季田间15N标记生长箱运行情况 Fig. 1 Operation of 15N2-labelling growth chambers in the whole rice growing season |
经过90 d 15N2标记试验,种水稻处理0 ~ 1 cm土层土壤的总氮量显著高于不种水稻处理;而其他土层(1 ~ 5、5 ~ 15 cm)以及整个盆中土壤,种水稻和不种水稻两处理的总氮量没有显著差异(表 1)。在所有土层(0 ~ 1、1 ~ 5、5 ~ 15 cm)中,种水稻处理的15N原子百分超和固氮量均显著高于不种水稻处理。种水稻处理的固氮量在土壤中为4.229 mg/盆,占水稻-土壤系统总固氮量的68.52%;0 ~ 1、1 ~ 5、5 ~ 15 cm各土层的固氮量分别占土壤中总固氮量的39.91%、16.46% 和占43.63%。不种水稻处理的总固氮量为0.599 mg/盆,0 ~ 1、1 ~ 5、5 ~ 15 cm各土层的固氮量分别占土壤总固氮量的53.76%、5.68% 和40.57%。
|
|
表 1 15N2标记试验90 d后,种水稻和不种水稻处理不同土层的总氮量、15N原子百分超和固氮量 Table 1 Total N, 15N atom % excess N fixation at different soil depths in pots with or without rice in 15N2-labelled chambers after labelled for 90 days |
水稻根部的总氮量显著低于地上部,而水稻根部的15N原子百分超显著高于地上部(表 2)。水稻植株固氮量在根部和地上部则没有显著差异。生物固氮量在整个水稻植株中的分配为1.943 mg/盆,占种水稻处理水稻-土壤系统总固氮量的31.48%,其中,水稻植株中根占56.18%,地上部分占43.82%。
|
|
表 2 15N2标记试验90 d后,种水稻处理水稻根和地上部的生物量、总氮量、15N原子百分超和固氮量 Table 2 Dry weight, total N, 15N atom % excess N fixation in rice roots, rice above ground parts and whole plant in 15N2-labelled chambers after labelled for 90 days |
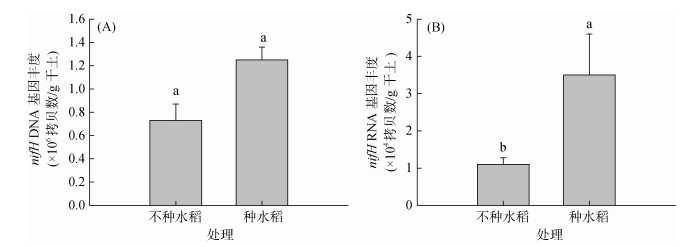
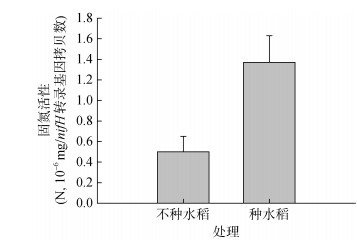
由图 2A可知,种水稻处理的nifH基因丰度为1.25×106拷贝数/g干土,高于不种水稻处理的0.73×106拷贝数/g干土,但二者差异未达显著水平。而种水稻处理的nifH反转录基因丰度为3.53×104拷贝数/g干土,显著高于不种水稻处理的1.14×104拷贝数/g干土(图 2B)。种水稻显著提高了固氮微生物平均固氮活性(图 3),其固氮活性是不种水稻处理的2.74倍。
|
(图中误差线表示标准误差(n=3),条形柱上不同小写字母表示不同处理间差异显著(P < 0.05);下同) 图 2 种水稻和不种水稻盆栽中0 ~ 1cm土壤中nifH DNA基因(A)和nifH RNA基因(B)拷贝数 Fig. 2 Copy numbers of nifH DNA gene(A) and nifH RNA gene (B) in 0-1 cm soil layers in pots with or without rice |
|
图 3 种水稻和不种水稻盆栽中0 ~ 1cm土壤中固氮菌固氮活性 Fig. 3 Average nitrogen fixation activities of nitrogen-fixing bacteria in 0-1cm soil layers in pots with or without rice |
与不种水稻处理相比,种水稻处理在所有土层(0 ~ 1、1 ~ 5、5 ~ 15 cm)均显著增加了固氮量(表 1),种水稻处理的总固氮量是不种水稻处理固氮量的10.3倍。生物固氮是一个极耗能的过程,因而也是一个需要碳源较多的过程[29-30]。水稻种植提高生物固氮量,一方面可能是由于水稻根系分泌的大量有机物质为生物固氮过程提供了能量来源[18, 31-32]。虽然固氮量因固氮微生物及植物种类的变化而差异较大,但植物通过为细菌生长和固氮菌提供碳源而发挥决定性作用[4]。植物固定的碳有很大一部分被分配到地下部,平均40% 的光合产物被转移到地下生物量中,而其中通常又有12% 被转化为根分泌物及其衍生代谢物和微生物生物量碳[33]。另一方面,水稻根系吸收了部分氮素,从而为固氮微生物创造了一定的低氮环境,减少氨抑制作用[34-35]。而且水稻能把大气中的N2转运到根部再扩散到根际,从而被根际固氮菌利用[36]。
不论是种水稻处理,还是不种水稻处理,0 ~ 1 cm土层的15N原子百分超均显著高于1 ~ 5 cm和5 ~ 15 cm,说明距离土壤表面越近,固氮作用越明显。种水稻处理在0 ~ 1 cm土层的固氮量显著高于1 ~ 5 cm土层,而与5 ~ 15 cm土层没有显著差异,说明0 ~ 1 cm土层虽然固氮作用强烈,但是土壤体积有限,而在5 ~ 15 cm土层,虽然固氮作用不如0 ~ 1 cm层强烈,但是体量庞大,最终在总的固氮量上并不比0 ~ 1 cm土层少。另外,种水稻处理的1 ~ 5 cm土层和5 ~ 15 cm土层固氮量对土壤-水稻系统总固氮量的贡献高于不种植水稻处理,尤其是1 ~ 5 cm土层。种水稻处理1 ~ 5 cm土层的贡献为16.46%,而不种水稻处理仅为5.68%。这可能是因为在相对较深层土壤中,异养微生物占绝大多数,而异养微生物不能自己产生能源,水稻分泌的能源物质则正好补充了这一点,说明种水稻对异养固氮微生物的促进作用在深层更加明显[8]。
种水稻显著提高了nifH RNA基因数量而对nifH基因数量的增加不显著,说明种水稻没有显著增加固氮微生物的数量,而是促进了固氮微生物固氮基因nifH的表达,显著提高了固氮微生物的活性(图 2、图 3)。这可能与水稻种植对土壤环境的改变及水稻根系分泌物有关。植物通过根系生长影响土壤空间结构[37],并通过根际分泌物为微生物生长提供基质[38]。植物根系分泌物由糖、有机酸和黏液等低分子化合物组成,是土壤微生物的活性碳源[39]。土壤碳源有效性及其组成是影响土壤微生物活性的关键,充足的土壤有效碳源,为生物固氮过程提供了能源,促进了固氮微生物固氮基因nifH的表达[40]。因此,水稻种植虽然没有增加固氮菌的数量,但是显著增加了固氮菌固氮基因nifH的表达量,从而显著提高了稻田生物固氮量。
4 结论本研究利用15N2直接标记技术测定了稻田固氮量,利用实时荧光定量PCR技术测定了固氮菌nifH基因及其反转录基因拷贝数,结果表明,种水稻显著增加了稻田各层土壤(0 ~ 1、1 ~ 5、5 ~ 15 cm)的固氮量。种水稻和不种水稻的固氮作用都是越靠近土壤表层(0 ~ 1 cm)越强烈,但是种水稻对较深土壤(1 ~ 5 cm和5 ~ 15 cm)固氮作用的促进作用更加明显。水稻种植没有增加固氮菌的数量,稻田固氮量的增加是因为水稻种植极大地促进了固氮菌nifH基因的表达,提高了固氮菌的固氮活性。
致谢: 本研究的大田试验得到了江都FACE实验站的朱国新和实验室王志玲的帮助,得到了南京土壤研究所刘钢高级工程师的技术指导,在此表示衷心感谢。
| [1] |
Sahoo R K, Ansari M W, Dangar T K, et al. Phenotypic and molecular characterisation of efficient nitrogen-fixing Azotobacter strains from rice fields for crop improvement[J]. Protoplasma, 2014, 251(3): 511-523 DOI:10.1007/s00709-013-0547-2 (  0) 0) |
| [2] |
Xie G H, Cai M Y, Tao G C, et al. Cultivable heterotrophic N2-fixing bacterial diversity in rice fields in the Yangtze River Plain[J]. Biology and Fertility of Soils, 2003, 37(1): 29-38 DOI:10.1007/s00374-002-0565-2 (  0) 0) |
| [3] |
Dynarski K A, Houlton B Z. Nutrient limitation of terrestrial free-living nitrogen fixation[J]. The New Phytologist, 2018, 217(3): 1050-1061 DOI:10.1111/nph.14905 (  0) 0) |
| [4] |
Rosenblueth M, Ormeño-Orrillo E, López-López A, et al. Nitrogen fixation in cereals[J]. Frontiers in Microbiology, 2018, 9: 1794 DOI:10.3389/fmicb.2018.01794 (  0) 0) |
| [5] |
Ladha J K, Reddy P M. Extension of nitrogen fixation to rice-Necessity and possibilities[J]. GeoJournal, 1995, 35(3): 363-372 DOI:10.1007/BF00989144 (  0) 0) |
| [6] |
Yoneyama T, Terakado-Tonooka J, Minamisawa K. Exploration of bacterial N2-fixation systems in association with soil-grown sugarcane, sweet potato, and paddy rice: A review and synthesis[J]. Soil Science and Plant Nutrition, 2017, 63(6): 578-590 DOI:10.1080/00380768.2017.1407625 (  0) 0) |
| [7] |
Yoshida T, Ancajas R R. Nitrogen-fixing activity in upland and flooded rice fields[J]. Soil Science Society of America Journal, 1973, 37(1): 42-46 DOI:10.2136/sssaj1973.03615995003700010017x (  0) 0) |
| [8] |
Bei Q C, Liu G, Tang H Y, et al. Heterotrophic and phototrophic 15N2 fixation and distribution of fixed 15N in a flooded rice-soil system[J]. Soil Biology and Biochemistry, 2013, 59: 25-31 DOI:10.1016/j.soilbio.2013.01.008 (  0) 0) |
| [9] |
Ladha J K, Reddy P M. Nitrogen fixation in rice systems: State of knowledge and future prospects[J]. Plant and Soil, 2003, 252(1): 151-167 DOI:10.1023/A:1024175307238 (  0) 0) |
| [10] |
Ma J, Bei Q C, Wang X J, et al. Paddy system with a hybrid rice enhances cyanobacteria Nostoc and increases N2 fixation[J]. Pedosphere, 2019, 29(3): 374-387 DOI:10.1016/S1002-0160(19)60809-X (  0) 0) |
| [11] |
Ito O, Cabrera D, Watanabe I. Fixation of dinitrogen-15 associated with rice plants[J]. Applied and Environmental Microbiology, 1980, 39(3): 554-558 DOI:10.1128/aem.39.3.554-558.1980 (  0) 0) |
| [12] |
Raymond J, Siefert J L, Staples C R, et al. The natural history of nitrogen fixation[J]. Molecular Biology and Evolution, 2004, 21(3): 541-554 DOI:10.1093/molbev/msh047 (  0) 0) |
| [13] |
Hsu S F, Buckley D H. Evidence for the functional significance of diazotroph community structure in soil[J]. The ISME Journal, 2009, 3(1): 124-136 DOI:10.1038/ismej.2008.82 (  0) 0) |
| [14] |
Nelson D R, Mele P M. The impact of crop residue amendments and lime on microbial community structure and nitrogen-fixing bacteria in the wheat rhizosphere[J]. Soil Research, 2006, 44(4): 319 DOI:10.1071/SR06022 (  0) 0) |
| [15] |
Tang Y F, Zhang M M, Chen A L, et al. Impact of fertilization regimes on diazotroph community compositions and N2-fixation activity in paddy soil[J]. Agriculture, Ecosystems & Environment, 2017, 247: 1-8 (  0) 0) |
| [16] |
Knelman J E, Legg T M, O'Neill S P, et al. Bacterial community structure and function change in association with colonizer plants during early primary succession in a glacier forefield[J]. Soil Biology and Biochemistry, 2012, 46: 172-180 DOI:10.1016/j.soilbio.2011.12.001 (  0) 0) |
| [17] |
Patra A K, Abbadie L, Clays-Josserand A, et al. Effects of management regime and plant species on the enzyme activity and genetic structure of N-fixing, denitrifying and nitrifying bacterial communities in grassland soils[J]. Environmental Microbiology, 2006, 8(6): 1005-1016 DOI:10.1111/j.1462-2920.2006.00992.x (  0) 0) |
| [18] |
Bürgmann H, Meier S, Bunge M, et al. Effects of model root exudates on structure and activity of a soil diazotroph community[J]. Environmental Microbiology, 2005, 7(11): 1711-1724 DOI:10.1111/j.1462-2920.2005.00818.x (  0) 0) |
| [19] |
Bergersen F J. The quantitative relationship between nitrogen fixation and the acetylene-reduction assay[J]. Australian Journal of Biological Sciences, 1970, 23(4): 1015 DOI:10.1071/BI9701015 (  0) 0) |
| [20] |
Mariotti A. Atmospheric nitrogen is a reliable standard for natural 15N abundance measurements[J]. Nature, 1983, 303(5919): 685-687 DOI:10.1038/303685a0 (  0) 0) |
| [21] |
Bremer E, van Kessel C. Appraisal of the nitrogen-15 natural-abundance method for quantifying dinitrogen fixation[J]. Soil Science Society of America Journal, 1990, 54(2): 404-411 DOI:10.2136/sssaj1990.03615995005400020018x (  0) 0) |
| [22] |
Giller K E, Day J M, Dart P J, et al. A method for measuring the transfer of fixed nitrogen from free-living bacteria to higher plants using 15N2[J]. Journal of Microbiological Methods, 1984, 2(6): 307-316 DOI:10.1016/0167-7012(84)90049-6 (  0) 0) |
| [23] |
Burris R H, Miller C E. Application of n15 to the study of biological nitrogen fixation[J]. Science, 1941, 93(2405): 114-115 DOI:10.1126/science.93.2405.114 (  0) 0) |
| [24] |
朱兆良, 陈德立, 张绍林, 等. 稻田非共生固氮对当季水稻吸收氮的贡献[J]. 土壤, 1986, 18(5): 225-229 (  0) 0) |
| [25] |
Unkovich M J, Peoples M B, David F H, et al. Measuring plant-associated nitrogen fixation in agricultural systems[J]. Australian Journal of Agricultural Research, 2008, 54: 333-340 (  0) 0) |
| [26] |
中国科学院南京土壤研究所土壤系统分类. 中国土壤系统分类检索(3版)[M].
中国科学技术大学, 合肥, 2001
(  0) 0) |
| [27] |
Wang X J, Liu B J, Ma J, et al. Soil aluminum oxides determine biological nitrogen fixation and diazotrophic communities across major types of paddy soils in China[J]. Soil Biology and Biochemistry, 2019, 131: 81-89 DOI:10.1016/j.soilbio.2018.12.028 (  0) 0) |
| [28] |
Ohyama T, Kumazawa K. A simple method for the preparation, purification and storage of 15N2 gas for biological nitrogen fixation studies[J]. Soil Science and Plant Nutrition, 1981, 27(2): 263-265 DOI:10.1080/00380768.1981.10431278 (  0) 0) |
| [29] |
Carnahan J E, Mortenson L E, Mower H F, et al. Nitrogen fixation in cell-free extracts of Clostridium pasteurianum[J]. Biochimica et Biophysica Acta, 1960, 44: 520-535 DOI:10.1016/0006-3002(60)91606-1 (  0) 0) |
| [30] |
Mortenson L E. Ferredoxin and atp, requirements for nitrogen fixation in cell-free extracts of Clostridium pasteurianum[J]. PNAS, 1964, 52(2): 272-279 DOI:10.1073/pnas.52.2.272 (  0) 0) |
| [31] |
Knauth S, Hurek T, Brar D, et al. Influence of different Oryza cultivars on expression of nifH gene pools in roots of rice[J]. Environmental Microbiology, 2005, 7(11): 1725-1733 DOI:10.1111/j.1462-2920.2005.00841.x (  0) 0) |
| [32] |
Stewart W D. Biological and ecological aspects of nitrogen fixation by free-living micro-organisms[J]. Proceedings of the Royal Society of London Series B, Biological Sciences, 1969, 172(1029): 367-388 (  0) 0) |
| [33] |
Jones D L, Nguyen C, Finlay R D. Carbon flow in the rhizosphere: Carbon trading at the soil-root interface[J]. Plant and Soil, 2009, 321(1/2): 5-33 (  0) 0) |
| [34] |
Reed S C, Cleveland C C, Townsend A R. Functional ecology of free-living nitrogen fixation: A contemporary perspective[J]. Annual Review of Ecology, Evolution, and Systematics, 2011, 42(1): 489-512 DOI:10.1146/annurev-ecolsys-102710-145034 (  0) 0) |
| [35] |
York L M, Carminati A, Mooney S J, et al. The holistic rhizosphere: Integrating zones, processes, and semantics in the soil influenced by roots[J]. Journal of Experimental Botany, 2016, 67(12): 3629-3643 DOI:10.1093/jxb/erw108 (  0) 0) |
| [36] |
Yoshida T, Broadbent F E. Movement of atmospheric nitrogen in rice plants[J]. Soil Science, 1975, 120(4): 288-291 DOI:10.1097/00010694-197510000-00007 (  0) 0) |
| [37] |
Angers D A, Caron J. Plant-induced changes in soil structure: Processes and feedbacks[J]. Biogeochemistry, 1998, 42(1/2): 55-72 DOI:10.1023/A:1005944025343 (  0) 0) |
| [38] |
Lynch J M, Whipps J M. Substrate flow in the rhizosphere[J]. Plant and Soil, 1990, 129(1): 1-10 (  0) 0) |
| [39] |
Bais H P, Weir T L, Perry L G, et al. The role of root exudates in rhizosphere interactions with plants and other organisms[J]. Annual Review of Plant Biology, 2006, 57(1): 233-266 DOI:10.1146/annurev.arplant.57.032905.105159 (  0) 0) |
| [40] |
Smercina D N, Evans S E, Friesen M L, et al. Erratum for smercina et al., "to fix or not to fix: Controls on free-living nitrogen fixation in the rhizosphere"[J]. Applied and Environmental Microbiology, 2019, 85(22): e02546-18.
(  0) 0) |
2. University of Chinese Academy of Sciences, Beijing 100049, China;
3. College of forestry, Nanjing Forestry University, Nanjing 210008, China
 2021, Vol. 53
2021, Vol. 53


