2. 中国科学院土壤环境与污染修复重点实验室(南京土壤研究所), 南京 210008;
3. 中国科学院生态环境研究中心, 北京 100085;
4. 江苏省农业科学研究院食品质量安全与检测研究所, 南京 210014
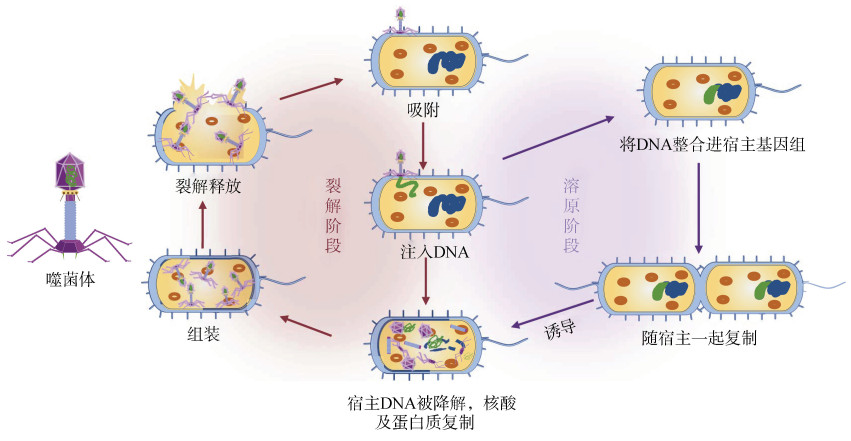
病毒是生物圈中数量最多的生命体,广泛分布在土壤、河流、海洋、湖泊、荒漠、森林等生境中。据估算,地球上总病毒大于1031数量级[1],有研究发现在沉积物和土壤中病毒数量分别占全球病毒粒子数量的87% 和10%,而在海洋水体中只占2.7%,说明土壤环境中病毒数量十分巨大[2]。土壤中栖息着各类生物,包括细菌、古菌、真菌在内的微生物,原生动物,线虫和蚯蚓等,而其中细菌、古菌的数量最多,侵染它们的病毒被称为噬菌体。因此,噬菌体是土壤中的主要病毒类群[3]。按形态划分,可将噬菌体分为有尾噬菌体(tailed phage)、无尾噬菌体(nontailed phage)和丝状噬菌体(filamen tous phage)[4]。依据核酸类型,可分为DNA型噬菌体和RNA型噬菌体。根据复制周期的不同,可将噬菌体分为烈性噬菌体(lyticphage)和溶原性噬菌体(lysogenic phage)[5]。以DNA型噬菌体为例,烈性噬菌体将自身DNA注入宿主细胞内,利用宿主的物质与能量,快速进行核酸及蛋白质的复制,并通过裂解细胞释放子代噬菌体。溶原性噬菌体又称为温和噬菌体(temperate phage),在溶原阶段,噬菌体不会迅速增殖及裂解宿主细菌,而是将DNA插入宿主染色体或在染色体外维持且随着宿主DNA一起复制,直到噬菌体被诱导进入烈性生活史的复制周期[6](图 1)。当外界条件不利时,为了躲避不良因子胁迫,噬菌体普遍以溶原状态存在,以确保其在寄主种群中能够长期存活并繁殖[7]。
|
图 1 噬菌体溶原裂解阶段示意图 Fig. 1 Schematic diagram of lytic and lysogenic stage of phage |
细菌在土壤关键功能中发挥的作用已逐渐受到重视,但作为土壤中数量最多的病毒体,也是细菌群落结构的重要影响因子,针对噬菌体生态功能的研究还有待加强。因此本文聚焦噬菌体对土壤碳氮循环转化影响的相关研究,试图对噬菌体在土壤中的功能有更加明确的认识。
1 噬菌体调控土壤营养元素循环的机制噬菌体与宿主的相互作用在土壤生物地球化学循环中发挥着重要功能[8]。根据噬菌体溶原及裂解两种生存策略以及形成的与宿主之间特定相互作用关系,可将噬菌体调控土壤营养元素的生物地球化学循环机制分为两类,即:下行调控(top-down regulation)和上行调控(bottom-up regulation)[9-10]。首先,噬菌体通过自上而下的机制侵染微生物宿主,改变土壤环境中微生物数量、群落结构以及功能。宿主细胞裂解,内容物释放到环境中,土壤中其他的生物可以利用内容物中营养元素进行新陈代谢,促进养分循环。其次,噬菌体携带辅助代谢基因(auxiliary metabolic genes,AMGs),造成自下而上的反馈调控机制。噬菌体可以作为土壤微生物间的穿梭载体,促进宿主间基因的水平转移(horizontal gene transfer,HGT),促使宿主菌群获得更加多元的功能基因[11]。AMGs可以在噬菌体感染周期中表达,改变宿主的新陈代谢,进而影响土壤养分元素在生物地球化学循环的效率和周期[12]。
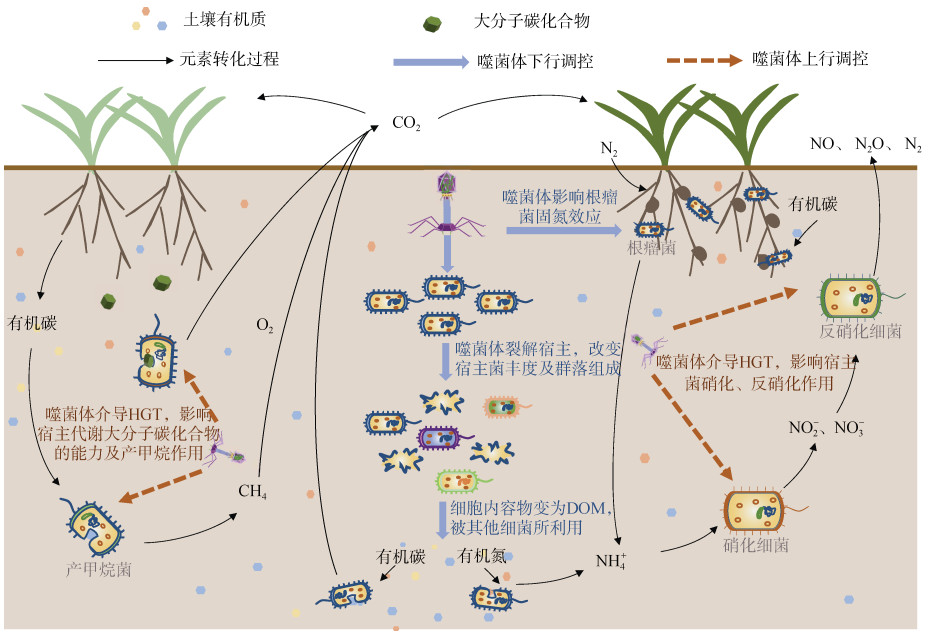
2 噬菌体对土壤碳元素循环的影响土壤是重要的陆地碳库,虽然微生物生物量碳(microbial biomass carbon, MBC)在土壤碳库中占比较小(1% ~ 4%),但却与土壤营养元素循环密切相关。微生物将有机质分解为简单无机物并释放出能量,为自身生长代谢使用,最终将大分子有机物分解为CO2,是土壤中微生物参与碳循环的重要环节,因此微生物是土壤有效养分的重要源和库[13]。基于噬菌体与宿主的密切联系,噬菌体可通过调控微生物群落组成及丰度或宿主菌基因来对土壤碳元素循环产生影响(图 2)。
|
图 2 噬菌体影响土壤碳氮循环示意图 Fig. 2 Phages'effects on soil carbon and nitrogen cycles |
Trubl等[10]在冻土中发现了噬菌体的预测宿主,这些宿主归属3个门4个属(疣微菌门:Pedosphaera;酸杆菌门:Acidobacterium及Candidatus Solibacter;变形菌门:Smithella)。这4种预测宿主在微生物群落中数量丰富,并且对土壤碳循环均有重要影响。其中Pedosphaera在土壤中普遍存在,是利用纤维素和葡萄糖的微生物[14];Acidobacterium和Candidatus Solibacter是多糖类物质的主要降解者[15-16];Smithella是专性厌氧菌,与产甲烷菌具有已知的共营养关系[17]。同样,Emerson等[18]在冻土中发现了噬菌体的预测宿主包括产甲烷菌和甲烷氧化菌,说明噬菌体可以通过调控产甲烷菌和甲烷氧化菌来影响土壤中的碳循环;另外,发现对于酸性细菌(Acidobacteriia)和氮螺旋菌(Nitrospirae)这两种细菌,病毒/宿主丰度与溶解性有机碳(dissolved organic carbon,DOC)浓度显著相关。上述这些病毒-宿主的联系为噬菌体通过调控与碳代谢有关微生物宿主种群数量,进而影响土壤碳循环提供了证据。而Allen等[19]的研究直接证明了噬菌体通过捕食作用对微生物群落的下行控制。其通过在土壤中添加含硫酸亚铁的茶叶提取物作为抗噬菌体剂,同时设置等量蒸馏水及乙酸盐做对照(乙酸盐处理提供了与抗噬菌体处理等量的碳),观察到相比于对照,添加茶叶提取物溶液4 d后,噬菌体平均丰度从3.53×108 PFU/m3土壤下降到1.58×107 PFU/m3土壤,而微生物细胞的平均计数有明显增加;虽然乙酸盐处理由于外源性碳的加入在初期短暂提高了微生物呼吸速率,但缺乏持续性的影响;然而加入茶叶提取物后的50 ~ 100 h,微生物呼吸速率稳定增加,并大于对照处理,表明噬菌体数量减少使得对宿主微生物的捕食作用减弱,宿主的代谢活性显著提高。上述案例说明噬菌体可以通过控制微生物的数量及活性来调节营养物质碳的流动。
2.1.2 裂解细菌,释放细胞内容物噬菌体裂解细胞,细胞内容物里的碳素释放到环境中变成DOC,由于微生物体积小、比表面积大,它们能在浓度较低时吸收养分物质,在与浮游植物等生物争夺养分时有较大竞争优势。因此,被裂解细菌内容物里的碳素很少能被传递到更高的营养水平,大部分DOC被微生物呼吸所消耗,不断地以CO2的形式排出。该过程也被称为“微生物-噬菌体-溶解性有机碳循环” (bacterium-phage-DOC loop) [20]。在该过程中病毒分流作用(viral shunt)扮演了重要角色:病毒作为一个分流器将来自异养微生物和光自养生物的养分物质运移到颗粒有机物(particulate organic matter, POM)和溶解性有机物(dissolved organic matter, DOM)库中,通过微生物的呼吸作用和光降解作用,其中大部分转化为CO2[21]。病毒分流是深海生态系统中不稳定有机碎屑的重要来源[22]。这一概念起源于海洋噬菌体,Roy等[23]利用透射电子显微镜(transmission electron microscope, TEM)估计病毒感染细胞频率(frequency of viral infected cells, FVIC)以推测由病毒介导的细菌死亡率,发现在羊牧区土壤中,测定的FVIC平均为23%,显著高于海洋或淡水环境报告的FVIC值(0.7% ~ 9%),这一结果在一定程度上说明噬菌体裂解细菌可能在土壤养分循环中也同样发挥了重要作用。而Li等[24]利用稳定性同位素探测技术证明了噬菌体介导的微生物环(phage-mediated microbial loop)在土壤中的存在。他们以13C标记水稻愈伤组织细胞,并加入土壤中培养,随后在T4噬菌体g23基因碎片中检测到了13C,追踪到在土壤环境条件下从愈伤组织细胞到T4型噬菌体的碳分流状态,证明噬菌体介导了分解根冠细胞细菌的裂解,促使碳素释放。噬菌体对微生物的裂解作用,虽然造成了个体微生物的死亡,但是随着细胞内容物的释放,减少了被土壤矿物质和土壤孔隙固定的碳素数量,使碳素变为有利于微生物利用的非稳定形态。因此,从整个微生物群落的角度分析,噬菌体裂解作用能反馈促进微生物的生长代谢和呼吸作用[25]。微生物群落的繁荣,又能促进噬菌体的裂解增殖,从而提高其对土壤中养分元素生物地球化学循环过程的影响力。在Starr等[26]的试验中,研究了土壤中RNA病毒的多样性及生态功能,试验结果显示,在一年生草原野生燕麦(Avena fatua)根际土壤中检测到的RNA噬菌体大多属于光滑噬菌体科(Leviviridae),使用标记基因来寻找这些噬菌体中依赖RNA为模板的RNA聚合酶序列(RNA- dependent RNA polymerase sequences,RdRps),共确定了1 338条序列,大大增加了对已有光滑噬菌体科RdRp序列多样性的认识;并且在种植野生燕麦盆栽试验的第3~ 22天,分别测定噬菌体群落变化,通过非度量多维尺度分析(一种将多维空间的研究对象简化到低维空间进行定位、分析和归类,同时又保留对象间原始关系的数据分析方法)发现光滑噬菌体群落中差异显著的聚类,随时间产生了明显的分化。由于光滑噬菌体没有一个已知的溶原阶段,噬菌体侵染会让微生物迅速裂解。因此,很大程度上噬菌体种群的改变能反映出在宿主中的复制状况,从而体现出微生物群落数量、组成的变化。高度的RNA病毒多样性和丰度以及它们种群的动态变化,表明存在大量病毒复制,随烈性噬菌体裂解释放到土壤中的微生物细胞内容物数量也十分巨大。基于此作者推测,烈性RNA噬菌体的增殖将对土壤中碳元素的形态、丰度和分布产生显著影响,从而对土壤中碳素循环过程具有塑造能力。虽然在该研究中没有定量证明RNA病毒对土壤碳循环的影响程度,但为今后研究土壤中RNA型噬菌体对生物地球化学循环的影响提供了思路。总之,噬菌体裂解细菌释放细胞内容物,可以认为提高了碳素在微生物群落中的利用效率。
2.2 对宿主基因的影响噬菌体可以通过辅助代谢基因或噬菌体编码的代谢基因来间接调控宿主群落生命过程,从而促进碳素的生物地球化学循环。Bi等[27]对玉米-大麦轮作农田生态系统中玉米抽穗期根际土壤的病毒群落进行了研究,通过功能预测,在病毒中发现了大量与碳水化合物代谢相关的功能;另外,在宏病毒组中发现了确认属于病毒基因组的48个碳水化合物活性酶(carbohydrate-active enzyme,CAZyme)辅助基因,功能包括编码糖苷水解酶(glycoside hydrolases)、碳水化合物酯酶(carbohydrate esterases)、碳水化合物结合模块(carbohydrate-binding modules),其中分解复杂有机物的糖苷水解酶基因数量最多,说明在农业生态系统中,噬菌体有助于碳循环的调节。Jin等[28]研究红树林土壤病毒,同样也发现了与其他辅助代谢功能基因相比,与“碳水化合物运输和代谢(carbohydrate transport and metabolism) ”相关的基因在红树林病毒中数量较多,其中大部分CAZyme基因参与红树林土壤生物碎屑中各类复合多糖的生物分解,并且系统发育分析显示,病毒CAZyme基因具有多样性,说明辅助碳水化合物代谢基因可能在红树林土壤病毒中广泛存在,这些病毒能够感染不同门的细菌。同样,Emerson等[18]也在冻土病毒组中发现了对大分子糖类具有降解能力的糖苷水解酶基因,而且甘露聚糖内切酶(endomannanase)被证明具有功能活性。上述研究中提到的各类基因对于土壤中复杂有机物的分解十分重要。土壤中有机碳的重要组成包括植物凋落物和动物残体,它们以复合碳水化合物或多糖的形式存在,纤维素、木聚糖、果胶、淀粉、甘露聚糖和几丁质等,是植物细胞壁、甲壳类外壳的主要成分,需要多种酶参与降解[29],为了促进在宿主细胞中的复制,噬菌体将利用各类复杂碳水化合物分解酶基因,提高宿主细胞代谢能力,加快对复杂碳水化合物的分解利用[30]。这一过程让大分子难降解有机物能在微生物代谢过程中被分解为小分子无机矿物,使碳素从大分子有机物中分解释放,促进碳素周转循环。噬菌体将碳水化合物活性酶基因介导进入宿主菌体内,随噬菌体复制及进一步感染,扩大该类基因在细菌群落内的分布,因此细菌群落对碳素的代谢能力也随之提升。
3 噬菌体对土壤氮元素循环的影响氮素是氨基酸、蛋白质的组成元素,对生命体的构成具有重要意义。土壤中氮素水平是植物生长的重要限制因子,供应不足会导致农作物品质和产量下降,而环境中的氮素过量会造成水体富营养化、地下水硝态氮积累及毒害等,因此了解土壤中氮素的循环转化过程十分重要[31]。固氮作用、硝化作用以及反硝化作用是土壤中微生物参与的重要氮素循环过程[32],其中涉及的微生物包括菌根真菌、根瘤菌以及土壤中存在的硝化细菌和反硝化细菌。同样,可将噬菌体对土壤氮素生物地球化学循环的影响与对宿主菌群落水平和基因水平的作用相对应,如图 2所示。
3.1 对宿主群落的影响 3.1.1 通过影响固氮菌数量减少固氮根瘤菌是指能够侵入植物根部,刺激根部细胞增生形成瘤状物的一类原核固氮微生物,其能够将空气中的分子态氮固定为氨,供植物吸收。豆科植物与根瘤菌的固氮量占生物固氮总量的65%,是一种高效的共生固氮体系[33]。根瘤菌噬菌体(rhizobiophage)能够调控宿主群落从而影响根瘤菌对氮素的固定。早期的一些研究表明,根瘤菌噬菌体通过侵染作用产生了大量的无效根瘤菌,或是直接观察到根瘤数量及质量的下降,影响植物的产量[34-35]。但是可能由于当时研究手段的限制,这些研究都是从单菌株纯培养的角度阐明了噬菌体对根瘤菌的影响,未能进一步说明根瘤菌与噬菌体间的侵染关系。Msimbira等[36]在南非种植花生(Arachis hypogea L.)的土壤中分离和纯化了3种属于长尾噬菌体科(Siphoviridae)的烈性噬菌体菌株(PRSA-1, PRSA-2和PRSA-26),并鉴定出3种易感根瘤菌(TUTAHSA126, TUTAHSA155和TUTAHSA75),通过nifH和glnII序列分析鉴定出3种根瘤菌属于慢生根瘤菌属(Bradyrhizobium);空斑实验显示,根瘤菌对噬菌体表现出不同的敏感性,TUTAHSA126对3种噬菌体均敏感,TUTAHSA155对PRSA-1、PRSA-2敏感,而TUTAHSA75仅对PRSA-1敏感。以上结果说明,南非土壤中噬菌体可以寄生在慢生根瘤菌上,并降低其数量,从而影响结瘤和N2的固定。噬菌体PRSA-1可寄生于3种慢生根瘤菌,表明其在土壤微生物环境中通过遗传转导或水平基因转移增加了它的感染能力。即使一种噬菌体对土壤中非同源根瘤菌没有直接显著影响,非同源根瘤菌也可能在进化过程中产生突变,形成易感菌株,噬菌体与突变体菌株相互作用导致基因转移,从而导致根瘤菌容易被噬菌体侵染。Appunu和Dhar[37]运用转座子Tn5诱变技术从慢生型大豆根瘤菌(B. japonicum ASR011)中分离出同时具有噬菌体抗性及结瘤能力的抗性菌株,在实验室控制条件下,接种噬菌体会影响普通根瘤菌与大豆的共生效应,与未接种噬菌体相比,结瘤量、根瘤质量、固氮酶活性、叶片氮含量均下降,而抗性菌株的共生效应未受到影响;在野外条件下,低剂量噬菌体(100和500 μl,浓度为108微粒/ml)不会对普通菌株及抗性菌株的共生效应产生影响,高剂量噬菌体(1 000 μl,浓度为108微粒/ml)会使普通菌株共生效应下降,而抗性菌株的结瘤量及根瘤质量增加。该试验说明在自然土壤条件下,接种抗性菌株及一定浓度噬菌体会改变根瘤菌种群中抗性种及普通种之间的竞争关系,土壤中低效根瘤菌数量减少,从而削弱噬菌体对结瘤固氮过程的负面影响。除了根瘤菌,也有研究报道了噬菌体对土壤中其他固氮微生物的作用。Li等[38]以中国东北某长期施用尿素的水田为研究对象,研究了淹水和施肥条件下土壤病毒和细菌群落动态,以及多种环境因子对土壤病毒的影响,结果发现,在施用氮磷钾肥的处理中,冗余度分析(redundancy analysis,RDA)显示,病毒丰度是微生物群落组成的重要影响因素,并且Rhodoplanes和Dongia两种固氮微生物的相对丰度与病毒丰度呈显著负相关,说明噬菌体对土壤中细菌群落具有特异性调控,对固氮菌的影响将限制微生物群落的固氮能力。若噬菌体大量裂解固氮菌使其数量减少,势必会影响氮素进入土壤的过程,因此可以说明噬菌体在控制元素循环关键环节中起到的重要作用。
3.1.2 裂解氮循环关键微生物Braga等[39]从两种土壤中分离出噬菌体和微生物群落,在不同的群落装配场景下(细菌群落定殖中、定殖后或自然土壤中)进行交互移植试验,研究噬菌体对土壤中细菌群落以及氮素有效性的影响,对土壤无机氮库的定量分析表明,在无菌土壤中接种微生物群落28 d后再接种非本土噬菌体,与不接种噬菌体相比,土壤中NH4+含量增加了2倍多;同样在自然土壤中,接种非本土噬菌体的NH4+浓度与对照相比平均增加了约10倍;在这两个处理中,噬菌体也显著改变了宿主微生物群落的多样性,证明噬菌体不但影响微生物群落,也影响了微生物介导的氮循环过程。噬菌体对土壤中NH4+含量的影响,一方面是因为噬菌体对微生物群落施加压力使其多样性发生改变,相比普通的矿化作用,硝化、反硝化这些被特定微生物持有的功能对环境扰动更敏感[40],比较容易随群落多样性的改变而变化,土壤氮素含量也随之受到影响;另一方面,噬菌体对微生物的裂解也会导致细胞质内容物的释放,从而增加土壤中氮素含量。说明噬菌体的裂解不仅影响宿主菌数量,对细菌群落功能的影响以及随之带来的土壤性质及生态功能的改变还需要进一步研究。
3.2 对宿主基因的影响反硝化过程是指在厌氧条件下,微生物将硝酸盐(NO3-)和亚硝酸盐(NO2-)还原为气态的NO、N2O或N2并获取能量的过程,也是土壤中氮素转化的重要阶段。噬菌体介导的水平基因转移可以使遗传物质在不同的微生物个体间进行移动,有利于微生物获得对自身有益的功能基因。Jang等[41]为了证明噬菌体能通过水平基因转移让反硝化功能基因在微生物群落间传播,从水稻土中分离出了41个固氮螺菌属(Azospirillum sp.)菌株,通过分析这些反硝化菌株的基因组,确定了在TSH58菌株基因组中,反硝化功能基因nirK和norCBQD位于移动遗传元件(mobile genetic elements)上,并且在反硝化诱导条件下,nirK会积极转录。位于移动遗传元件的基因如果与前噬菌体或接合元件(如接合质粒)相联合,就可以在多个菌株的基因组之间转移。因为nirK是菌株TSH58中唯一的亚硝酸盐还原酶基因,所以证明该菌株很可能通过水平基因转移获得反硝化功能基因而获益。虽然没有后续试验验证,但移动遗传元件中的反硝化基因如果移动到前噬菌体区域,噬菌体被激活后这些基因就能随子代噬菌体释放到外界环境,随着下一次的感染转移到其他微生物细胞中,促进微生物群落的反硝化功能提升,从而提高土壤氮循环周转速率。González等[42]报道了蚕豆共生固氮根瘤菌Rhizobium etli的完整基因组序列,发现在该根瘤菌染色体中存在一些与噬菌体相关基因,如编码噬菌体末端转移酶大小亚基和噬菌体溶解酶素及重组酶类的基因,间接证明了噬菌体可以介导水平基因转移;确定了通过HGT转移到R. etli中的基因主要由小分子代谢、运输和转录调节功能类的基因构成,例如:编码寡肽转运子的appABCDF基因、编码葡萄糖胺生物合成的glms2nagA基因、编码铁运输的sfuAB基因等,证明了噬菌体介导的水平基因转移有助于提高该根瘤菌的代谢能力,而根瘤菌代谢能力的增强又进一步有利于植物共生固氮作用,增加输入土壤中的氮素通量。辅助代谢基因对细菌群落的代谢能力的提升至关重要,也因此反映出噬菌体促进细菌转化营养元素举足轻重的作用。
4 影响噬菌体调节土壤碳氮元素循环的因素 4.1 碳氮循环过程的交互作用土壤有机质中碳氮元素含量之间均存在一定范围内的计量关系(C/N),这一计量关系很大程度上决定了土壤氮循环和碳循环之间存在交互作用,一种元素的转变会对另一元素产生影响。由于氮素是土壤中的限制性元素,供氮效应好的土壤,其固碳能力高[43]。同时也有研究表明,外源性碳输入会增加土壤中氨氧化细菌、反硝化细菌丰度,促进氮素转化过程[44]。因此,由噬菌体介导的对碳素或氮素循环转化的单一影响,都可能在碳氮交互作用中产生更加深远的意义。例如,噬菌体裂解细胞释放易于细菌利用的有机碳,碳素的增加导致C/N变化,进而可能会对细菌硝化作用和反硝化作用产生不同的效果。今后的研究应该从碳氮循环的交互作用及耦合机制出发,从机理上理解土壤中噬菌体影响碳氮循环转化的过程,对土壤噬菌体在元素地球循环中发挥的生态作用有更加明确的认识。
4.2 土著噬菌体类型土著噬菌体可以影响宿主微生物的适应、捕食、竞争关系,进而影响其进化及群落组成[45],而不同类型的噬菌体具有不同的形态学和基因组特征以及生命周期和复制策略,因而会对微生物产生不一样的作用效果。如丝状噬菌体生命周期以慢性感染为特征,以溶原性策略在宿主体内复制,在感染后不立即裂解宿主细胞,能够增强宿主菌抵御非生物和生物胁迫,协助宿主菌提高入侵新栖息地和群落繁殖的能力[46]。而肌尾科(Myoviridae)T4噬菌体是一种烈性噬菌体,对于调控宿主微生物种群数量以及活性具有重要作用[47]。有研究假设噬菌体在海洋环境中多为烈性,而在土壤中则以溶原性为主[20],虽然这一假设有待进一步验证,但土壤中裂解、溶原噬菌体的比例,以及二者之间的转换关系,都会对土壤中养分元素的转化和循环产生不同的影响。
4.3 土著宿主菌群落噬菌体能对微生物群落施加压力,影响整个群落的生存发展,同样潜在宿主的群落结构和丰度发生改变能造成噬菌体群落的动态变化[48]。噬菌体只能感染特定的微生物宿主,只有噬菌体对宿主的感染事件发生,那么由噬菌体引发的对土壤碳氮循环的影响才能产生。另外,土壤具有微观异质性,微生物群落的分布状况甚至会在微米范围内出现明显差异,根际土壤微生物群落组成及数量就明显不同于非根际土壤。当宿主微生物数量越大时,就存在可能有更多的细胞被噬菌体裂解,因而细胞内容物释放并输入土壤中的碳氮元素量则越大。宿主微生物为了躲避噬菌体的感染,进化出了CRISPR-Cas系统用来免疫外来DNA或RNA的入侵;为了克服这一系统,噬菌体可能会突变或删除它们的CRISPR靶点,或者获得一种被称为anti-CRISPRs (ACRs)的抑制蛋白,干扰CRISPR-Cas的活性[49]。因此,不同种宿主菌表现出对噬菌体敏感性的差异。微生物群落中优势物种的类别也能影响噬菌体对宿主的捕食效果。“杀死获胜者(kill-the-winner)”假说表明,噬菌体通过攻击最活跃的优势原核生物种群,使有限的资源被其利用,并限制微生物群落单一化发展[50-51]。所以宿主菌在整个微生物群落中的竞争优势能影响噬菌体与之相互作用的强度。因而,土著微生物的群落组成、丰度、在土壤中的分布状况及其对噬菌体的敏感性等特征,会使宿主与噬菌体之间产生复杂多样的相互作用关系,进而对土壤碳氮循环产生程度各异的影响。
4.4 土壤理化性质土壤理化性质影响噬菌体及宿主微生物的群落结构和生长代谢状况,因此对噬菌体驱动的土壤碳氮循环起到重要调控作用。土壤pH可以影响噬菌体在土壤颗粒中的吸附解吸情况,并与噬菌体丰度显著相关,在一定范围内噬菌体丰度随pH升高而增大,但超过最适值之后,会随pH增大而减小[52],例如在德国海尼希地区森林、草地、农田土壤样本中,随采样点pH从3.5到5,噬菌体丰度增加,而噬菌体群落生长最适pH为5左右,证明了pH对噬菌体丰度具有显著影响[48];温度会影响土壤中噬菌体的生存能力及丰度,在低温下噬菌体能保持其活性持久,而高温条件容易造成病毒粒子的热衰变[53],在3 ~10 ℃实验室无菌培养条件下,沙土中可检测到病毒感染单位的时长达170 d,而超过30℃后,时长缩短到40 d,证明高温条件可能会让土壤中病毒更易失活[54];土壤水分状况也会影响噬菌体丰度,通常情况下土壤湿度大能提高微生物活性,进而促使更多噬菌体复制,因此有利于噬菌体存活[55];土壤有机质含量代表了土壤养分状况,如果噬菌体感染营养不良的宿主,病毒染色体不能直接复制而停留在宿主细胞内,所以高有机质含量有利于提高噬菌体丰度[56]。此外,土壤质地、容重、CEC等也会改变土壤中宿主微生物的生存环境,因此也可能会同步影响噬菌体群落丰度和组成。这些土壤理化性质通过直接影响噬菌体的生存状况,进而间接改变碳氮元素循环的进程。其次,噬菌体对宿主微生物代谢过程的调控作用也可以影响土壤碳氮循环转化,而氧气含量、pH、CEC等土壤理化性质能够决定养分元素在土壤中的赋存形态以及元素转化的反应条件,进而影响微生物对这些代谢底物的利用程度及宿主微生物的代谢方式。土壤深度变化也会影响噬菌体丰度以及生殖策略,随土层加深噬菌体数量减少,而溶原碎片数量增加,说明噬菌体溶原性策略的发生率与土壤深度呈正相关[57]。另外土层深度也决定了噬菌体的辅助代谢基因的数量,有研究显示,病毒组中AMGs在土壤次表层的丰度比表面的丰度高16倍[58]。土壤碳氮元素循环受到噬菌体的调控作用,而土壤理化性质能通过改变噬菌体的生长状况来影响碳氮循环过程。
4.5 地理环境因素土壤中病毒的丰度及多样性与地理环境也显著相关。有研究表明,森林和湿地土壤中的病毒丰度通常高于沙漠和农业土壤中的病毒丰度[25],且同一种土壤类型,土壤中病毒丰度、多样性也会随所处地理位置不同而发生改变。Han等[59]比较了中国不同省份种植水稻和玉米的6种农业土壤,噬菌体丰度在各土壤中表现出差异,山东种植玉米的土壤中病毒丰度最高(1.5×1010 PFU/g土壤),吉林种植水稻黑土病毒颗粒丰度最低(5.1×109 PFU/g土壤);6种土壤有各自独特的病毒种类分布,微小噬菌体科(Microviridae)在吉林水稻、山东玉米和江苏水稻土壤中含量最高,圆环病毒科(Circoviridae)在吉林玉米和湖南水稻土壤中最为丰富,长尾噬菌体科(Siphoviridae)在江西水稻土壤中最多;对6种土壤病毒功能基因进行注释,发现了不同土壤所含功能基因类型及丰度也有所不同。同样在玉米根际土壤与红树林土壤[27-28]病毒组中发现的大多数CAZyme基因编码不同种类多糖分解酶,证明环境气候条件影响土壤中碳水化合物[e1] 组成及噬菌体宿主微生物类型,造成土壤微生物代谢方式的改变,病毒中与碳水化合物相关的辅助代谢基因就出现差别。Gupta等[60]基于宏基因组方法揭示了土地利用方式对土壤微生物的影响,对比花园土壤,医院周围土壤中氮素代谢相关基因显著减少,并且花园土壤中根瘤菌数量相对更富集,说明土地利用方式的改变影响土壤中微生物对氮素的转化利用。由于噬菌体与宿主之间的密切联系,可以推断微生物对氮素利用的改变能影响噬菌体与相关宿主之间的相互作用,因此不同土地利用方式间接影响噬菌体参与的氮素转化过程。此外,喷洒杀虫剂、施加化肥以及降雨情况等其他环境因素也驱动着土壤微生物群落的改变[61],噬菌体对宿主的调控作用也会受到连带影响。综上,地理环境因素影响噬菌体数量、多样性、繁殖策略、代谢状况与宿主的关系,最终作用于噬菌体影响土壤碳氮循环的过程。
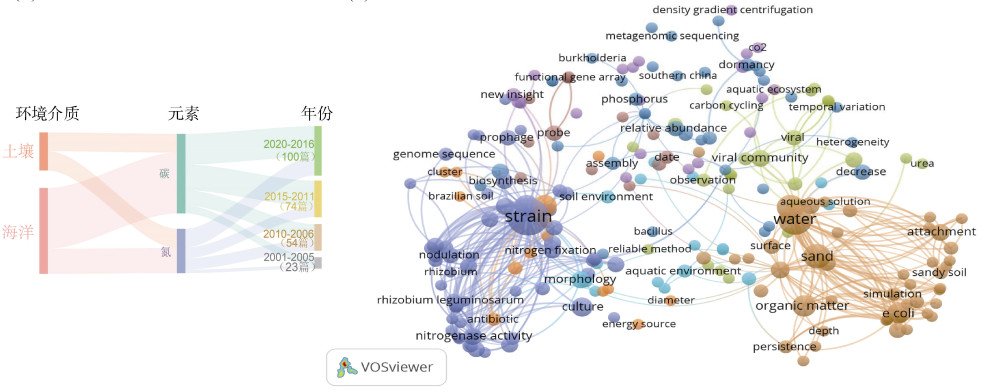
5 研究展望 5.1 现有研究难点为了解目前有关噬菌体影响土壤碳氮循环的研究情况及发展趋势,从Web of Science上采用主题词“(phage or bacteriophage)AND (soil or terrestrial) AND (carbon) OR(nitrogen)”进行检索,共获取到2001——2020年间的77篇文章,同时用“(phage or bacteriophage) AND (ocean or sea or marine) AND (carbon) OR (nitrogen)”检索共获取174篇文章(图 3A)。对文献量的统计可以看出,目前土壤环境中噬菌体影响碳氮循环的研究数量少于海洋环境,其中碳素、氮素研究的文章量分别为161和90,但近5年的研究数量相较于2001——2005年增长了约5倍,说明噬菌体影响元素化学循环这一功能越来越被研究者重视。然而,噬菌体影响土壤碳氮循环的研究依然存在巨大发展空间。对土壤中获取的77篇文献进行关键词共现分析(图 3B),发现“abundance”、“diversity”、“community”一类词出现的频次较大(总频次为28),证明目前的研究大多集中在对噬菌体群落的描述。另外,与碳氮循环直接相关的关键词如“carbon cycling”、“greenhouse”、“nitrogen fixation”等出现频次并不高(总频次为8),反映出现有研究直接与碳氮循环转化的关联度可能较低。并且由于文献数量过低,所形成的关键词聚类不能很好地反映现有研究之间的关系,也体现补充噬菌体影响碳氮循环转化研究空白的重要性。
|
图 3 近20年文献量统计及关键词共现分析 Fig. 3 Literature volume statistics and keyword co-occurrence analysis in recent 20 years |
基于文献统计,对目前研究中的难点进行总结:
1) 土壤中噬菌体具有种类和功能的多样性,噬菌体溶原及裂解两种复制策略也会对宿主产生不同的作用效果,但少有研究分类比较不同种类噬菌体或温和、烈性噬菌体的状态对土壤碳氮循环的影响。
2) 在研究过程中,大部分病毒序列由于缺少参比对象被列为未知序列,这一部分基因的缺失,可能减少土壤病毒被注释功能的数量及类型。因此,在评估噬菌体对土壤碳氮元素循环作用效果时会存在偏差。
3) 目前大部分研究都只揭示了与碳氮有关的病毒组中代谢基因或噬菌体潜在宿主,并得出噬菌体对碳氮元素生物地球化学循环具有潜在影响这一结论,然而未能验证相关基因的功能,或阐释相关宿主与噬菌体的相互作用关系。
4) 噬菌体对土壤碳氮循环的影响少有定量描述,而在海洋环境中,已有研究证明每年约有3亿t碳通过病毒分流释放到环境中[62],病毒基因的相对丰度也因此被认为是全球深海碳通量的最佳预测因子[63]。
5) 已有较多研究报道了土壤理化性质、气候条件会调控噬菌体或微生物群落,但气候、土壤条件变化对病毒-宿主关系的改变,以及对噬菌体驱动碳氮循环这一生态系统功能造成的影响程度尚不清楚,特别在全球变化的背景下,值得深入研究。
5.2 未来展望当前噬菌体生态功能的信息,大多是从海洋病毒的研究中获取,然而针对土壤噬菌体的认知还不够全面,基于此,未来关于噬菌体影响元素化学循环的研究,可以从以下几方面加强:
1) 目前病毒基因数据库较为匮乏,应加强对病毒分离纯化及基因组注释的研究,构建并拓展面向阐释噬菌体宿主预测及对应碳氮循环相关功能基因数据库。
2) 利用同位素标记的方法追踪元素在土著噬菌体群落中的流动,了解病毒粒子对不同元素的利用比例关系,探究噬菌体影响土壤碳氮循环的分子机制,阐释噬菌体影响土壤养分元素转化的过程。
3) 未来的研究应该重视不同条件土壤中噬菌体与宿主菌的相互作用关系,揭示在协同进化过程中的主导因子[64],并建立模型,实现在非试验研究条件下对噬菌体宿主之间相互作用的预测。
4) 比较不同时间、空间尺度噬菌体对土壤碳氮元素循环的影响,同时比较溶原、烈性噬菌体对碳氮元素转化的效率差异和作用机制,强化RNA类型噬菌体的研究。
5) 建立能够直观地对噬菌体影响生物地球化学循环这一生态功能进行评估的研究方法,为噬菌体影响土壤碳氮循环提供直接证据。加强对噬菌体影响碳氮循环程度的定量描述,进一步明确噬菌体在土壤碳氮循环过程中发挥的生态作用。
6) 目前噬菌体影响土壤中其他元素的研究数量远远少于碳氮,所以应加强噬菌体对土壤中如磷、硫等元素循环转化的影响研究,比较噬菌体对不同元素转化作用权重大小,逐步实现利用噬菌体靶向调控土壤中养分元素,达到绿色可持续利用的目的。
| [1] |
Suttle C A. Viruses in the sea[J]. Nature, 2005, 437(7057): 356-361 DOI:10.1038/nature04160 (  0) 0) |
| [2] |
Cobián Güemes A G, Youle M, Cantú V A, et al. Viruses as winners in the game of life[J]. Annual Review of Virology, 2016, 3(1): 197-214 DOI:10.1146/annurev-virology-100114-054952 (  0) 0) |
| [3] |
王光华. 掀开土壤生物"暗物质"——土壤病毒的神秘面纱[J]. 中国科学院院刊, 2017, 32(6): 575-584 (  0) 0) |
| [4] |
付丽娜, 徐永平, 李晓宇, 等. 噬菌体控制致病性大肠杆菌的研究进展[J]. 畜牧与兽医, 2017, 49(12): 140-144 (  0) 0) |
| [5] |
Howard-Varona C, Hargreaves K R, Abedon S T, et al. Lysogeny in nature: Mechanisms, impact and ecology of temperate phages[J]. The ISME Journal, 2017, 11(7): 1511-1520 DOI:10.1038/ismej.2017.16 (  0) 0) |
| [6] |
Emerson J B. Soil viruses: A new hope[J]. mSystems, 2019, 4(3): SI e00120-19 (  0) 0) |
| [7] |
王光华, 刘俊杰, 朱冬, 等. 土壤病毒的研究进展与挑战[J]. 土壤学报, 2020, 57(6): 1319-1332 (  0) 0) |
| [8] |
Pratama A A, van Elsas J D. The 'neglected' soil virome -potential role and impact[J]. Trends in Microbiology, 2018, 26(8): 649-662 DOI:10.1016/j.tim.2017.12.004 (  0) 0) |
| [9] |
Brum J R, Sullivan M B. Rising to the challenge: Accelerated pace of discovery transforms marine virology[J]. Nature Reviews Microbiology, 2015, 13(3): 147-159 DOI:10.1038/nrmicro3404 (  0) 0) |
| [10] |
Trubl G, Jang H B, Roux S, et al. Soil viruses are underexplored players in ecosystem carbon processing[J]. mSystems, 2018, 3(5) (  0) 0) |
| [11] |
韩丽丽, 贺纪正. 病毒生态学研究进展[J]. 生态学报, 2016, 36(16): 4988-4996 (  0) 0) |
| [12] |
Breitbart M. Marine viruses: Truth or dare[J]. Annual Review of Marine Science, 2012, 4(1): 425-448 DOI:10.1146/annurev-marine-120709-142805 (  0) 0) |
| [13] |
齐边斌, 赵鑫勇, 吕德国. 影响土壤有机碳动态变化的因素研究进展[J]. 北方果树, 2019(4): 1-4 (  0) 0) |
| [14] |
Bergmann G T, Bates S T, Eilers K G, et al. The under-recognized dominance of Verrucomicrobia in soil bacterial communities[J]. Soil Biology and Biochemistry, 2011, 43(7): 1450-1455 DOI:10.1016/j.soilbio.2011.03.012 (  0) 0) |
| [15] |
Woodcroft B J, Singleton C M, Boyd J A, et al. Genome-centric view of carbon processing in thawing permafrost[J]. Nature, 2018, 560(7716): 49-54 DOI:10.1038/s41586-018-0338-1 (  0) 0) |
| [16] |
Pearce D A, Newsham K K, Thorne M A, et al. Metagenomic analysis of a southern maritime Antarctic soil[J]. Frontiers in Microbiology, 2012, 3: 403 (  0) 0) |
| [17] |
Wawrik B, Marks C R, Davidova I A, et al. Methanogenic paraffin degradation proceeds via alkane addition to fumarate by 'Smithella' spp. mediated by a syntrophic coupling with hydrogenotrophic methanogens[J]. Environmental Microbiology, 2016, 18(8): 2604-2619 DOI:10.1111/1462-2920.13374 (  0) 0) |
| [18] |
Emerson J B, Roux S, Brum J R, et al. Host-linked soil viral ecology along a permafrost thaw gradient[J]. Nature Microbiology, 2018, 3(8): 870-880 DOI:10.1038/s41564-018-0190-y (  0) 0) |
| [19] |
Allen B, Willner D, Oechel W C, et al. Top-down control of microbial activity and biomass in an Arctic soil ecosystem[J]. Environmental Microbiology, 2010, 12(3): 642-648 DOI:10.1111/j.1462-2920.2009.02104.x (  0) 0) |
| [20] |
Mara P, Vik D, Pachiadaki M G, et al. Viral elements and their potential influence on microbial processes along the permanently stratified Cariaco Basin redoxcline[J]. The ISME Journal, 2020, 14(12): 3079-3092 DOI:10.1038/s41396-020-00739-3 (  0) 0) |
| [21] |
Suttle C A. Marine viruses-major players in the global ecosystem[J]. Nature Reviews Microbiology, 2007, 5(10): 801-812 DOI:10.1038/nrmicro1750 (  0) 0) |
| [22] |
Danovaro R, Dell'Anno A, Corinaldesi C, et al. Major viral impact on the functioning of benthic deep-sea ecosystems[J]. Nature, 2008, 454(7208): 1084-1087 DOI:10.1038/nature07268 (  0) 0) |
| [23] |
Roy K, Ghosh D, DeBruyn J M, et al. Temporal dynamics of soil virus and bacterial populations in agricultural and early plant successional soils[J]. Frontiers in Microbiology, 2020, 11: 1494 DOI:10.3389/fmicb.2020.01494 (  0) 0) |
| [24] |
Li Y, Watanabe T, Murase J, et al. Identification of the major capsid gene (g23) of T4-type bacteriophages that assimilate substrates from root cap cells under aerobic and anaerobic soil conditions using a DNA-SIP approach[J]. Soil Biology and Biochemistry, 2013, 63: 97-105 DOI:10.1016/j.soilbio.2013.03.026 (  0) 0) |
| [25] |
Williamson K E, Fuhrmann J J, Wommack K E, et al. Viruses in soil ecosystems: An unknown quantity within an unexplored territory[J]. Annual Review of Virology, 2017, 4(1): 201-219 DOI:10.1146/annurev-virology-101416-041639 (  0) 0) |
| [26] |
Starr E P, Nuccio E E, Pett-Ridge J, et al. Metatranscriptomic reconstruction reveals RNA viruses with the potential to shape carbon cycling in soil[J]. Proceedings of the National Academy of Sciences of the United States of America, 2019, 116(51): 25900-25908 DOI:10.1073/pnas.1908291116 (  0) 0) |
| [27] |
Bi L, Yu D T, Du S, et al. Diversity and potential biogeochemical impacts of viruses in bulk and rhizosphere soils[J]. Environmental Microbiology, 2021, 23(2): 588-599 DOI:10.1111/1462-2920.15010 (  0) 0) |
| [28] |
Jin M, Guo X, Zhang R, et al. Diversities and potential biogeochemical impacts of mangrove soil viruses[J]. Microbiome, 2019, 7(1): 1-15 DOI:10.1186/s40168-018-0604-3 (  0) 0) |
| [29] |
Gao B L, Jin M, Li L, et al. Genome sequencing reveals the complex polysaccharide-degrading ability of novel deep-sea bacterium Flammeovirga pacifica WPAGA1[J]. Frontiers in Microbiology, 2017, 8: 600 (  0) 0) |
| [30] |
Hurwitz B L, U'Ren J M. Viral metabolic reprogramming in marine ecosystems[J]. Current Opinion in Microbiology, 2016, 31: 161-168 DOI:10.1016/j.mib.2016.04.002 (  0) 0) |
| [31] |
黄巧云. 土壤学[M].
中国农业出版社, 北京, 2016
(  0) 0) |
| [32] |
王朱珺, 王尚, 刘洋荧, 等. 宏基因组技术在氮循环功能微生物分子检测研究中的应用[J]. 生物技术通报, 2018, 34(1): 1-14 (  0) 0) |
| [33] |
沈仁芳, 赵学强. 土壤微生物在植物获得养分中的作用[J]. 生态学报, 2015, 35(20): 6584-6591 (  0) 0) |
| [34] |
Evans J, Barnet Y M, Vincent J M. Effect of a bacteriophage on colonisation and nodulation of clover roots by paired strains of Rhizobium trifolii[J]. Canadian Journal of Microbiology, 1979, 25(9): 974-978 DOI:10.1139/m79-149 (  0) 0) |
| [35] |
Hashem F M, Angle J S. Rhizobiophage effects on nodulation, nitrogen fixation, and yield of field-grown soybeans (Glycine max L. Merr.)[J]. Biology and Fertility of Soils, 1990, 9(4): 330-334 DOI:10.1007/BF00634110 (  0) 0) |
| [36] |
Msimbira L A, Jaiswal S K, Dakora F D. Identification and characterization of phages parasitic on bradyrhizobia nodulating groundnut (Arachis hypogaea L.) in South Africa[J]. Applied Soil Ecology, 2016, 108: 334-340 DOI:10.1016/j.apsoil.2016.09.010 (  0) 0) |
| [37] |
Appunu C, Dhar B. Isolation and symbiotic characteristics of two Tn5-derived phage-resistant Bradyrhizobium japonicum strains that nodulate soybean[J]. Current Microbiology, 2008, 57(3): 212-217 DOI:10.1007/s00284-008-9176-y (  0) 0) |
| [38] |
Li Y T, Sun H, Yang W C, et al. Dynamics of bacterial and viral communities in paddy soil with irrigation and urea application[J]. Viruses, 2019, 11(4): 347 DOI:10.3390/v11040347 (  0) 0) |
| [39] |
Braga L P P, Spor A, Kot W, et al. Impact of phages on soil bacterial communities and nitrogen availability under different assembly scenarios[J]. Microbiome, 2020, 8(1): 52 DOI:10.1186/s40168-020-00822-z (  0) 0) |
| [40] |
Schimel J P, Schaeffer S M. Microbial control over carbon cycling in soil[J]. Frontiers in Microbiology, 2012, 3: 348 (  0) 0) |
| [41] |
Jang J, Sakai Y, Senoo K, et al. Potentially mobile denitrification genes identified in Azospirillum sp. strain TSH58[J]. Applied and Environmental Microbiology, 2019, 85(2): e02474-e02418 (  0) 0) |
| [42] |
González V, Santamaría R I, Bustos P, et al. The partitioned Rhizobium etli genome: Genetic and metabolic redundancy in seven interacting replicons[J]. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(10): 3834-3839 DOI:10.1073/pnas.0508502103 (  0) 0) |
| [43] |
Li D J, Niu S L, Luo Y Q. Global patterns of the dynamics of soil carbon and nitrogen stocks following afforestation: A meta-analysis[J]. The New Phytologist, 2012, 195(1): 172-181 DOI:10.1111/j.1469-8137.2012.04150.x (  0) 0) |
| [44] |
高思齐, 宋艳宇, 宋长春, 等. 增温和外源碳输入对泥炭地土壤碳氮循环关键微生物功能基因丰度的影响[J]. 生态学报, 2020, 40(13): 4617-4627 (  0) 0) |
| [45] |
Mai-Prochnow A, Hui J G K, Kjelleberg S, et al. 'Big things in small packages: The genetics of filamentous phage and effects on fitness of their host'[J]. FEMS Microbiology Reviews, 2015, 39(4): 465-487 DOI:10.1093/femsre/fuu007 (  0) 0) |
| [46] |
Li Y, Liu H Y, Pan H, et al. T4-type viruses: Important impacts on shaping bacterial community along a chronosequence of 2000-year old paddy soils[J]. Soil Biology and Biochemistry, 2019, 128: 89-99 DOI:10.1016/j.soilbio.2018.10.007 (  0) 0) |
| [47] |
Sharma R S, Karmakar S, Kumar P, et al. Application of filamentous phages in environment: A tectonic shift in the science and practice of ecorestoration[J]. Ecology and Evolution, 2019, 9(4): 2263-2304 (  0) 0) |
| [48] |
Narr A, Nawaz A, Wick L Y, et al. Soil viral communities vary temporally and along a land use transect as revealed by virus-like particle counting and a modified community fingerprinting approach (fRAPD)[J]. Frontiers in Microbiology, 2017, 8: 1975 DOI:10.3389/fmicb.2017.01975 (  0) 0) |
| [49] |
Uribe R V, van der Helm E, Misiakou M A, et al. Discovery and characterization of Cas9 inhibitors disseminated across seven bacterial Phyla[J]. Cell Host & Microbe, 2019, 25(2): 233-241 (  0) 0) |
| [50] |
van Goethem M W, Swenson T L, Trubl G, et al. Characteristics of wetting-induced bacteriophage blooms in biological soil crust[J]. mBio, 2019, 10(6): e02287-19 (  0) 0) |
| [51] |
Winter C, Bouvier T, Weinbauer M G, et al. Trade-offs between competition and defense specialists among unicellular planktonic organisms: The "killing the winner" hypothesis revisited[J]. Microbiology and Molecular Biology Reviews, 2010, 74(1): 42-57 DOI:10.1128/MMBR.00034-09 (  0) 0) |
| [52] |
Chen L, Xun W B, Sun L, et al. Effect of different long-term fertilization regimes on the viral community in an agricultural soil of Southern China[J]. European Journal of Soil Biology, 2014, 62: 121-126 DOI:10.1016/j.ejsobi.2014.03.006 (  0) 0) |
| [53] |
Wen K, Ortmann A C, Suttle C A. Accurate estimation of viral abundance by epifluorescence microscopy[J]. Applied and Environmental Microbiology, 2004, 70(7): 3862-3867 DOI:10.1128/AEM.70.7.3862-3867.2004 (  0) 0) |
| [54] |
Duboise S M, Moore B E, Sorber C A, et al. Viruses in soil systems[J]. CRC Critical Reviews in Microbiology, 1979, 7(3): 245-301 DOI:10.3109/10408417909082016 (  0) 0) |
| [55] |
Williamson K E, Corzo K A, Drissi C L, et al. Estimates of viral abundance in soils are strongly influenced by extraction and enumeration methods[J]. Biology and Fertility of Soils, 2013, 49(7): 857-869 DOI:10.1007/s00374-013-0780-z (  0) 0) |
| [56] |
Williamson K E, Radosevich M, Wommack K E. Abundance and diversity of viruses in six Delaware soils[J]. Applied and Environmental Microbiology, 2005, 71(6): 3119-3125 DOI:10.1128/AEM.71.6.3119-3125.2005 (  0) 0) |
| [57] |
Liang X L, Zhang Y Y, Wommack K E, et al. Lysogenic reproductive strategies of viral communities vary with soil depth and are correlated with bacterial diversity[J]. Soil Biology and Biochemistry, 2020, 144: 107767 DOI:10.1016/j.soilbio.2020.107767 (  0) 0) |
| [58] |
Liang X L, Wagner R E, Zhuang J, et al. Viral abundance and diversity vary with depth in a southeastern United States agricultural ultisol[J]. Soil Biology and Biochemistry, 2019, 137: 107546 DOI:10.1016/j.soilbio.2019.107546 (  0) 0) |
| [59] |
Han L L, Yu D T, Zhang L M, et al. Genetic and functional diversity of ubiquitous DNA viruses in selected Chinese agricultural soils[J]. Scientific Reports, 2017, 7: 45142 (  0) 0) |
| [60] |
Gupta S, Kumar M, Kumar J, et al. Systemic analysis of soil microbiome deciphers anthropogenic influence on soil ecology and ecosystem functioning[J]. International Journal of Environmental Science and Technology, 2017, 14(10): 2229-2238 DOI:10.1007/s13762-017-1301-7 (  0) 0) |
| [61] |
Wakelin S A, MacDonald L M, Rogers S L, et al. Habitat selective factors influencing the structural composition and functional capacity of microbial communities in agricultural soils[J]. Soil Biology and Biochemistry, 2008, 40(3): 803-813 DOI:10.1016/j.soilbio.2007.10.015 (  0) 0) |
| [62] |
Breitbart M, Bonnain C, Malki K, et al. Phage puppet Masters of the marine microbial realm[J]. Nature Microbiology, 2018, 3(7): 754-766 (  0) 0) |
| [63] |
Guidi L, Chaffron S, Bittner L, et al. Plankton networks driving carbon export in the oligotrophic ocean[J]. Nature, 2016, 532(7600): 465-470 (  0) 0) |
| [64] |
Kuzyakov Y, Mason-Jones K. Viruses in soil: Nano-scale undead drivers of microbial life, biogeochemical turnover and ecosystem functions[J]. Soil Biology and Biochemistry, 2018, 127: 305-317 (  0) 0) |
2. Key Laboratory of Soil Environment and Pollution Remediation, Institute of Soil Science, Chinese Academy of Sciences, Nanjing 210008, China;
3. Research Center for Eco-Environmental Sciences, Chinese Academy of Sciences, Beijing 100085, China;
4. Institute of Food Safety and Quality, Jiangsu Academy of Agricultural Sciences, Nanjing 210014, China
 2021, Vol. 53
2021, Vol. 53


