2. 中国科学院大学, 北京 100049;
3. 江苏省环境工程技术有限公司, 南京 210019;
4. 生态环境部南京环境科学研究所, 南京 210042
随着中国“退二进三”、“退城进园”等经济战略实施,大批工业企业关闭搬迁遗留了上千个之多的有机氯农药污染场地,这些场地面临用地功能转换及二次开发[1-2]。这些潜在高风险污染场地土壤严重威胁人体健康和社会安全,是当前亟需解决的环境问题[3]。1,4-二氯苯是化工行业污染场地土壤和钢铁烧结烟气中典型的挥发性有机污染物,其在污染场地土壤最高检出浓度达到2 340.0 mg/kg,具有强挥发性和难降解性,其可通过大气传输,在食物链中富集,严重影响人体健康和生态系统稳定[3-5]。因此,利用经济有效的土壤修复技术去除土壤中残留的1,4-二氯苯十分必要。
生物修复方式是一种能耗低、环保、处理效果稳定的污染土壤治理技术。在有机氯农药生物修复过程中,微生物生长,尤其是高活性降解微生物与土壤中有机氯农药降解密切相关[6-8]。但是在土壤环境中,土壤异质性、营养物质传质缓慢、有机质对微生物的吸附等影响了土著微生物的降解效率[6, 9-10]。因而,提高土著菌群降解活性、强化微生物生存能力,可以有效改善土壤微生物降解有机氯农药效率慢的问题。向原有土壤生态系统中引入特定功能的营养物质或者微生物菌剂(106 ~ 108 CFU/µl),可以改善原有环境体系中微生物组成,增强土著菌群对有机污染物的适应能力和降解效率[8, 10-12]。前人研究发现,从污染土壤筛选、分离和改造土著降解微生物,而后重新外源加入原环境体系,可以用来生物强化原土壤中微生物降解效率,并且这种体系具有强竞争力和应对外部压力的抵御能力[6, 11-12]。同时,土著微生物降解有机氯农药活性与其降解基因含量,以及携带降解基因的微生物种类等密切相关[13-14]。目前,针对生物强化土著微生物降解活性相关研究,主要为外源添加或经生物工程技术改造的土著菌群,很少涉及外源添加降解基因DNA片段后土著菌群降解活性方面的研究。因此,外源添加降解基因或携带降解基因的菌群后,对土著微生物群落组成、群落动态、降解基因动态变化等进行全面识别,深入分析微生物降解活性动态变化,可以用来评估土著微生物的后续降解活性。
本研究对外源添加降解基因和表达降解基因的模式菌——大肠杆菌后,土著菌群组成、多样性及降解活性演变规律进行探究。首先采集了中国长三角典型1,4-二氯苯污染场地土壤,对添加降解基因或菌群前后,土壤中农药降解动力学进行分析;其次,结合高通量测序技术及共现网络分析方法,对外源添加降解基因后微生物群落组成及关键种群进行分析,阐释微生物群落及关键种群活性变迁;然后,通过荧光定量PCR分析土壤微生物群落降解基因含量动态变化,重点关注微生物降解活性演变规律,以探究微生物群落与降解活性间的联系。本研究结果剖析外源添加降解基因后土壤微生物多样性及降解活性动态变化,有助于探明外源降解基因刺激土著关键物种抵御有机氯农药胁迫的机制,为调控优化1,4-二氯苯污染土壤修复过程及效果提供新科学认知。
1 材料与方法 1.1 供试污染土壤和降解基因/降解菌的获取供试污染土壤采集自江苏省江阴市青阳镇某1,4-二氯苯污染场地(31°45′N,120°14′E)。该场地由有机氯农药生产企业搬迁产生,有30多年农药生产历史,主要有机氯农药污染种类是氯苯、二氯苯和硝基氯苯类农药。土壤样品均采集自表层0 ~ 20 cm,经自然风干、混合均匀后,使用气相色谱仪测定其中1,4-二氯苯浓度[5]。降解基因选择了专属性降解二氯苯农药的xylH、dmpB和catE 3种基因,并在NCBI(National Center for Biotechnology Information, http://www.ncbi.nlm.nih.gov/)网站下载了它们的标准序列,使用标准质粒pUC19对这些基因进行合成,获取到降解基因DNA片段(表 1);同时,使用商用大肠杆菌E.coli DH5α对这些降解基因进行单独表达,获取到含有降解基因片段菌群[15-16]。
|
|
表 1 基因序列及其降解功能 Table 1 Gene sequences and their degradation function |
将污染土壤放置于25 ℃恒温箱中培养7 d,并定期补水,设置预试验,分别设定外源含降解菌群添加量为105 ~ 106 CFU/μl,降解基因质粒DNA添加量为102 ~ 103 copies/μl。最终设定了CK(对照组)、PL-1(xylH质粒DNA)、PL-2(dmpB质粒DNA)、PL-3(catE质粒DNA)、BA-1(含xylH菌群)、BA-2(含dmpB菌群)、BA-3(含catE菌群)等7个处理组,并分别取第0、30、60、90、120、150、180、210天的土壤样品分析1,4-二氯苯浓度,同时将所有采集的样品置于4 ℃冰箱保存。1,4-二氯苯浓度采用气相色谱-质谱方法检测,其回收率介于64.7% ~ 77.1%。
1.3 土壤DNA提取及高通量测序使用DNeasy PowerSoil试剂盒(QIAGEN)提取所有土壤样品DNA,–80 ℃下储存,并使用Qubit 3.0测定DNA样品浓度,总量达到要求后使用Illumina Truseq Nano DNA LT Library Prep Kit试剂盒进行文库构建,采用Miseq平台进行双端测序。带有Barcode的特异性引物对样本16S rRNA进行扩增:前端引物为341F(5’-CCTAYGGGRBGCASCAG-3’),反向引物为806R(5’-GGACTACNNGGGTATCTAAT-3’)。测序得到的PE reads首先根据overlap关系进行拼接,同时对序列质量进行质控和过滤,并使用Usearch软件和gold数据库,采用denovo和reference结合的方式去除嵌合体,得到31 790 ~ 46 643条序列(表 2)。按照97% 相似性对非重复序列进行OTU聚类,采用RDP classifier贝叶斯算法对OUT代表序列进行物种分类学分析[17]。
|
|
表 2 土壤菌群测序序列长度(bp) Table 2 Illumina sequence length of soil indigenous flora |
利用Cytoscape 3.9.0中的CoNet算法对注释物种之间相互关系网络进行构建[18]。在网络构建之前,对所有样本进行过滤,除去少于1/3样本中存在的物种,然后计算每个物种相对丰度。基于整体网络方法判定注释物种之间的共存与排斥关系,两个相关性度量为Pearson和Spearman,两个不相似性度量设定为Bray-Curtis和Kullback-Leibler。通过选择相关系数绝对值最高的1 000个连线进行后续分析,并对相关系数进行标准化处理,使用1 000次迭代,并将置信区间设置为99%[19]。将Cytoscape处理完成的数据Excel表格导入Gephi 0.9.2,对网络平均度、平均加权度、平均聚类系数等进行模块化计算后,将其中中介中心度值≤0.5、紧密中心性值≥0.5、连接度≥3的物种判定为关键物种[20-23],最后,利用Gephi软件中Fruchterman Reingold布局方式对网络分析图进行可视化[24]。
1.5 实时荧光定量PCR分析依托软件Primer 6.0设计各基因专一性引物,每个基因都设计了4对引物,并使用PCR测试各引物对样品中各基因的专一性,后选择扩增效果最好、条带最清晰的一对引物进行实时荧光定量PCR试验(quantitative polymerase chain reaction, qPCR)(表 3)。反应体系在96-孔板上准备,每个样品准备3个平行。在25 μl体系中,包含了10 μl TB Green Premix (Takara,T li RNaseH Plus 2×),10 μmol/L前端引物和后端引物各0.8 μl,DNA模板2 μl,StepOnePlus Real-Time PCR system(Applied Biosystem)用于qPCR定量,扩增程序如下:95 ℃变性30 s,95 ℃延伸5 s共计40个循环,60 ℃终延伸30 s。各基因标准质粒DNA连续稀释(10–1 ~ 10–7)配制标准曲线,以将Ct值转换为拷贝数。测试的样品Ct值低于34时,则确认样品不受污染[25]。
|
|
表 3 基因引物序列 Table 3 Gene primer sequences |
文中所有柱状图、折线图、热图等均使用GraphPad Prism 8.0.1实现可视化,相关系数图等通过RStudio 3.6.1的corrplot等包实现可视化。
2 结果 2.1 土壤中1,4-二氯苯的降解动力学为探究外源添加降解基因对土壤中有机氯农药降解效率影响,本研究分别测定了为期210 d中1,4-二氯苯的降解动力学(图 1)。在对照组CK中,1,4-二氯苯消减过程最缓慢,降解率为18.59%;外源添加降解基因DNA片段处理组中(PL-1, PL-2, PL-3),有机氯农药降解率为32.35% ~ 38.43%,是对照组降解率的1.74倍~ 2.07倍;外源添加携带降解基因菌群处理组中(BA-1, BA-2, BA-3),1,4-二氯苯降解率最高,为43.10% ~ 44.74%,是对照组降解率的2.32倍~ 2.41倍。该结果说明,外源添加含降解基因菌群或DNA片段会加速1,4-二氯苯降解,且外源添加携带降解基因菌群处理组中,有机氯农药降解最快。
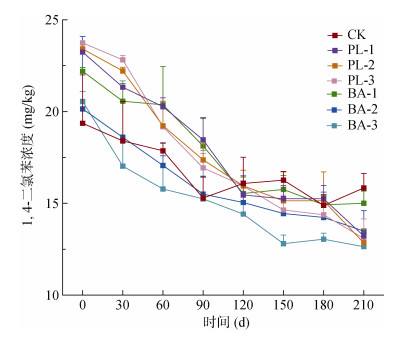
|
图 1 1,4-二氯苯降解动力学 Fig. 1 Degradation of 1,4-dichlorobenzene |
对不同土壤处理组进行高通量测序,发现在土壤菌群中都检测到了古菌和细菌群落,不同处理组中菌群结构与组成差异显著(图 2)。第0天时,在门水平上,各处理组中都包含的前5个优势细菌门分别是变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)、放线菌门(Actinobacteria)、酸杆菌门(Acidobacteria)和芽单胞菌门(Gemmatimonadetes);第210天时,各处理中都包含的前5个优势细菌门变化为Proteobacteria、Acidobacteria、绿弯菌门(Chloroflexi)、Bacteroidetes和疣微菌门(Verrucomicrobia)(图 2A)。在各处理组中,除PL-1中Proteobacteria和Acidobacteria外,其余处理中细菌门相对于第0天都表现出了显著差异(P < 0.05)。同时,相较于对照组,外源添加降解基因菌群或DNA片段后,土壤菌群多样性指数(Simpson指数)、丰富度指数(Chao1)并没有发生显著改变(图 2B,表 4)。该结果表明,外源添加含降解基因菌群或者DNA片段会显著改变土壤中优势菌门,但土壤菌群多样性并没有因此发生显著上升或者下降。
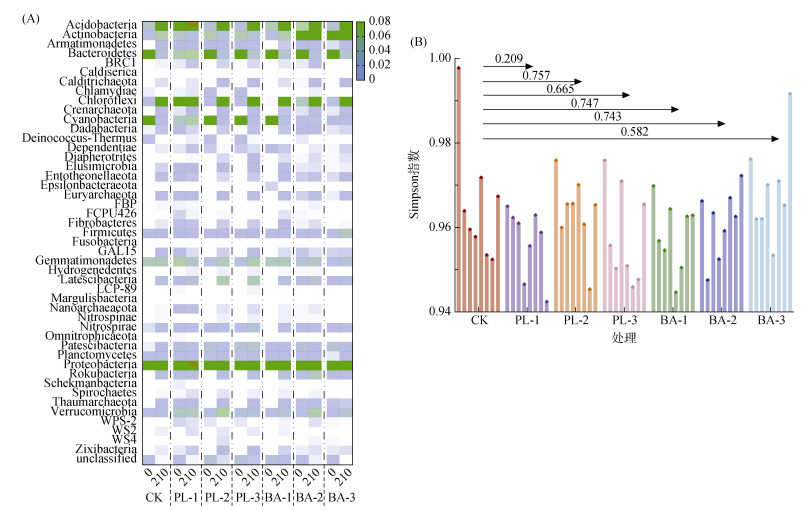
|
(A. 外源添加含降解基因菌群及质粒DNA后,在门水平上,第0天(0)和第210天(210)土壤中菌群相对丰度变化,其中,**标记为210 d后土壤菌群相对丰度显著上升;B. 各样本210 d内菌群Simpson指数变化柱状图,不同颜色代表不同处理组,且箭头上方的数字代表各处理组与对照组之间的Pearson系数) 图 2 土著菌群组成与多样性动态变化 Fig. 2 Variations of composition and diversity of soil indigenous flora |
|
|
表 4 土壤菌群Chao1指数变化 Table 4 Variations of diversity index (Chao1) of soil indigenous flora |
构建不同处理组中菌群共现网络,可以深度评估外源添加降解基因对农药污染土壤中微生物结构的潜在影响(图 3)。以对照组中各菌群之间共存与互斥关系占比作为基准,外源添加降解基因后,土壤菌群间共存关系分别增加了10.85% ~ 40.06%(DNA)和36.64% ~ 58.46%(菌群);该结果说明,外源添加降解基因后,菌群间共存关系发生的频次显著高于对照组中共存关系发生的频次(P < 0.01);并且,外源添加含降解基因菌群后,菌群间共存关系增加幅度显著高于添加降解基因片段的处理组(P < 0.05)。同时,与对照组相比,各处理组中共现网络特征值,如平均连接度和平均中介中心度等均有显著下降(表 5)。
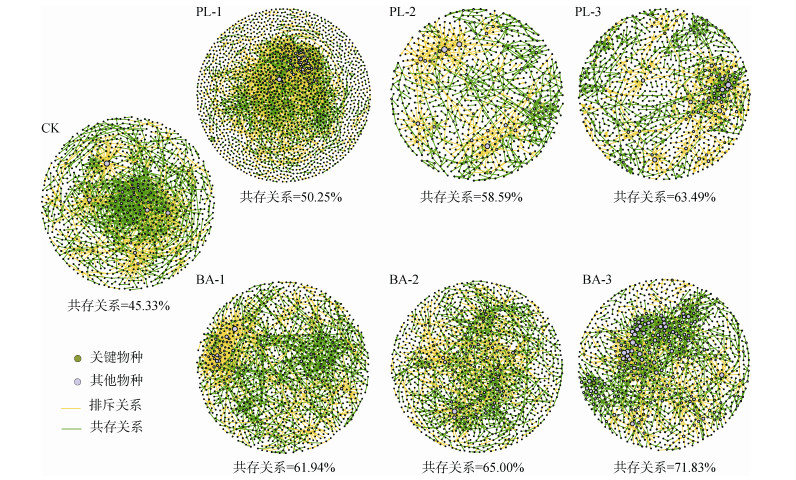
|
(在网络中,每一个点都代表不同菌群,灰绿色点代表了关键物种,灰蓝色点代表了非关键物种;点大小代表了该物种和其他物种之间的连接度,点越大,代表连接度越高;点与点之间的连接线代表了物种之间相互关系,绿色代表共存关系,橙色代表排斥关系) 图 3 土壤关键物种的共现网络 Fig. 3 Co-occurrence networks of soil keystone taxa |
|
|
表 5 土壤菌群共现网络的特征值 Table 5 Eigenvalues of co-occurrence networks of soil indigenous flora |
基于共现网络分析方法,本研究在对照组、外源降解基因DNA添加和外源含降解基因菌群添加处理组中分别筛选到24种、24 ~ 68种、38 ~ 32种关键物种(图 4)。在不同处理中,这些关键物种相对丰度占比由对照组中0.87% 上升为1.77% ~ 2.41%(外源降解基因DNA处理组)和1.86% ~ 4.24%(外源含降解基因菌群处理组)。其中,在对照组中,这些关键物种属于Proteobacteria(0.39%)、Chloroflexi(0.15%)、Verrucomicrobia(0.12%)、Gemmatimonadetes(0.11%)、Acidobacteria(0.046%)、匿杆菌门(Latescibacteria,0.040%)、Actinobacteria(0.0037%)和泉古菌门(Crenarchaeota,0.0071%);在外源添加降解基因DNA片段处理组中,除了上述7个细菌门和1个古菌门外,这些关键物种还隶属于硝化螺旋菌门(Nitrospirae,0 ~ 0.32%)、Bacteroidetes(0 ~ 0.14%),棒状杆菌门(Rokubacteria,0 ~ 0.039%),迷踪菌门(Elusimicrobia,0 ~ 0.014%)和蓝细菌(Cyanobacteria,0 ~ 0.0061%);而与对照组相比,在外源添加含降解基因菌群处理组中,关键物种还隶属于Rokubacteria(0.011% ~ 0.53%)、Nitrospirae(0.0085% ~ 0.40%)、Bacteroidetes (0 ~ 0.035%)、髌骨细菌门(Patescibacteria,0 ~ 0.016%)和厚壁菌门(Firmicutes,0 ~ 0.0071%)这5个细菌门。同时,与整体共现网络特征值相比,关键物种的平均连接度、平均紧密中心性等特征值显著上升(P < 0.01),平均中介中心度等特征值显著下降(P < 0.05,表 6),网络中心未发生显著变化。该结果说明,与共现网络中其他非关键物种相比,关键物种与其他物种之间连接度更高;去除这些非关键物种,共现网络中心未发生改变,因而关键物种在网络中承担着枢纽作用。

|
(图柱上方数字代表各处理组与对照组之间的相关系数,*表示相关性达P < 0.05显著水平) 图 4 土壤关键物种的相对丰度堆积图 Fig. 4 Histogram of relative abundance of soil keystone taxa |
|
|
表 6 土壤关键菌群共现网络的特征值 Table 6 Eigenvalues of co-occurrence networks of soil keystone taxa |
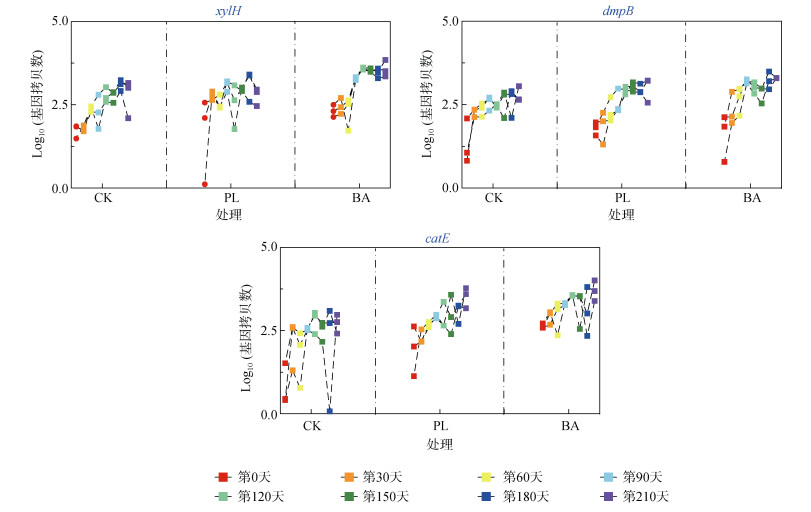
关注土壤中降解基因动态变化也有助于洞悉外源添加降解基因对于土著菌群降解功能的潜在影响。本研究对所有处理组中降解基因210 d内动态变化进行了定量,其中,xylH、dmpB和catE 3种降解基因都能专属作用于苯环类物质降解,尤其是氯苯物质降解。在对照组中,与第0天相比,在第210天时这3种降解基因都有显著上调,xylH、dmpB和catE分别增长了59.48%、67.54% 和51.53%;在外源添加降解基因DNA片段处理中,第210天相对于第0天时,xylH、dmpB和catE分别增长了73.55%、108.13% 和82.66%;而在外源添加含降解基因菌群处理中,相对于第0天时,3种基因在第210天时分别增长了171.66%、110.76% 和126.46%(P < 0.05)。同时,在外源添加降解基因处理中,降解基因增长率比对照组上升了1.24 ~ 1.60个数量级;外源添加降解菌群处理组中,降解基因增长率则比对照组上升了1.64 ~ 2.89个数量级。该结果说明,外源添加降解基因或降解菌群之后,土著菌群中潜在降解基因含量均显著上升,特别是添加含降解基因的菌群效果更显著(P < 0.05)。
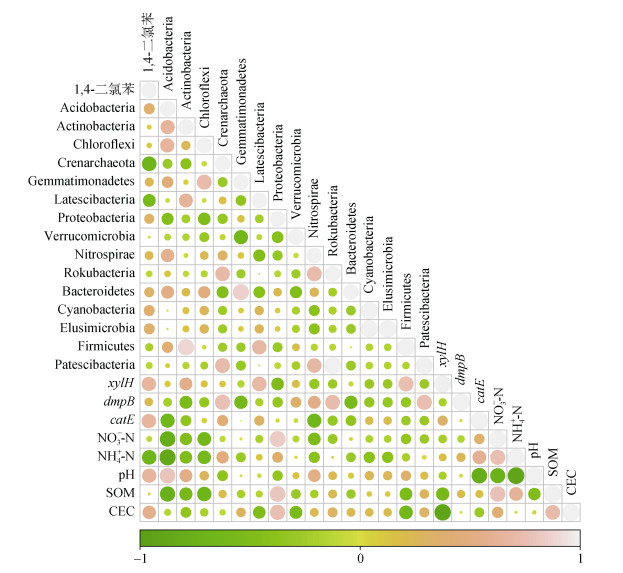
2.6 土壤性质、关键菌群与降解基因的交互作用为了深入分析土壤菌群降解活性的影响因素,选择有机氯农药、土壤理化性质、关键菌群及降解基因分析它们之间相互作用关系(图 6)。其中,1,4-二氯苯降解率除与Crenarchaeota、Latescibacteria等关键物种之间呈显著负相关关系外,与Proteobacteria、Bacteroidetes、Acidobacteria、Elusimicrobia和Cyanobacteria等细菌门之间都呈显著正相关关系,与3种降解基因拷贝数也呈显著正相关关系;在土壤环境因子中,土壤NO3–-N和NH4+-N都与农药降解率之间呈负相关关系,土壤pH、有机质含量和CEC等都与1,4-二氯苯的降解率呈显著正相关关系。该结果说明,在外源添加降解基因DNA片段或菌群处理中,土著菌群降解1,4-二氯苯能力与土壤关键菌群增长和降解基因增长显著正相关。
3 讨论 3.1 外源添加降解基因提升土著菌群降解活性外源添加降解基因及降解菌群210 d后,土壤菌群多样性指数(Simpson指数)和丰富度指数(Chao1)变化并不显著,说明外源添加降解基因及其载体并不会造成土著菌群发生显著波动,这表明外源降解基因添加量(菌群105 ~ 106 CFU/μl和DNA片段102 ~ 103 copies/μl)并不会挤占原有土著菌群的生长空间,这也与土壤环境的稳定性和缓冲能力有关(图 1和表 1)[26-27]。同时,降解基因和降解菌群添加组中,1,4-二氯苯降解效率都显著上升,分别提升了1.74倍~ 2.07倍和2.32倍~ 2.41倍,且降解菌群的添加组中,1,4-二氯苯的降解效率最高,达到44.74%,这说明,外源添加降解基因与降解菌群对土著菌群降解活性的提升效果并不一致,根本原因是这两者在土壤环境中归趋并不相同(图 2)。研究表明,微生物经过结合、吸收、整合等过程后才可能表达外源基因片段,而未被成功表达的基因片段会很快进入土壤环境,被土壤颗粒吸附或被土壤DNA酶降解[28]。在直接添加降解菌群处理组中,微生物已经成功表达了基因片段,并且,菌群细胞外也附着了大量胞外聚合物,这些胞外聚合物中含有可观的降解基因片段,因而,在添加降解菌群处理组中,土著菌群的降解活性提升更显著[29]。此外,共现网络分析结果也显示,外源添加降解基因和降解菌群处理组中,土壤菌群共现网络共存关系比例都显著增加,分别增加了10.85% ~ 40.06% 和36.64% ~ 58.46%,并且外源添加降解菌群处理组中,共现网络共存关系比例上升幅度更大(图 3)。前人研究证实,共存关系比例较高时,有助于提高体系代谢效率,体系稳定性随之下降;互斥关系高时,共现网络稳定,竞争有利于体系维持稳定[30]。因此,与对照组相比,外源添加降解基因和降解菌群后,土壤体系中微生物代谢活性上升,与污染物降解率结果相一致。
3.2 外源添加降解基因促进关键物种应对农药胁迫关键物种在生态系统中的功能一直广受关注,它们驱动着土壤养分循环、植物生长、生物地球化学循环等过程,与生态系统和人体健康的维护息息相关[22-24]。关键物种在环境中丰度较低,在本研究中仅占比0.87% ~ 4.24%,但它们个体或者群体都会对微生物组结构和功能产生巨大影响。利用高通量测序和共现网络分析方法,联合高平均连接度、高紧密中心性和低中介中心性这3个指标共同筛选关键物种,这种方法具有85% 的准确性[20-22]。在本研究中,外源添加降解基因和降解菌群后,1,4-二氯苯污染土壤中变形菌门(Proteobacteria)、酸杆菌门(Acidobacteria)、绿弯菌门(Chloroflexi)、拟杆菌门(Bacteroidetes)和疣微菌门(Verrucomicrobia)等优势菌门的环境比例上升(图 2),这5种菌门被证实与土壤中养分循环和农药降解过程相关[31]。这些优势菌门中也包含了一些关键物种,与优势菌门利用其环境丰度发挥作用不同,关键物种更依赖于其在土壤菌群网络中的高连接度和与其他微生物之间紧密的互作关系(图 3和图 4)[20-22]。当土壤中污染信号传递时,关键物种可以利用这种高连接度网络将信息传递给微生物群落中其他成员,进而影响微生物群落对污染物质的胁迫响应过程。此外,外源添加降解基因和菌群后,关键物种在环境中占比也显著上升(P < 0.01),尤其是添加catE降解基因和菌群处理组中,关键物种上升幅度最大。这可能的原因:①catE比其他两种降解基因在土壤环境中的原始绝对定量更高(图 5);②有研究表明,与其他两种基因只在生长稳定期发挥作用不同,单功能过氧化氢酶catE在早期生长阶段也能发挥其生理作用,并且它的作用在生长稳定期持续上升[32]。这也表明外源添加降解基因增加了关键物种之间潜在互作关系,加强了微生物群落间有机氯农药降解功能传递(图 4)。结合相关关系分析,关键物种与1,4-二氯苯降解率和降解基因环境丰度呈现显著正相关(图 6),表明关键物种降解能力提升与有机氯农药降解程度显著正相关,因而,提升关键物种降解能力能有效调控有机氯农药污染土壤中微生物群落的降解效率。
|
图 5 土壤降解基因的绝对丰度折线图 Fig. 5 Line chart of absolute abundance of soil degradation genes |
|
(图中圆圈大小代表相关系数大小) 图 6 土壤理化性质、关键种群及降解基因间的相关关系矩阵 Fig. 6 Correlation matrix among soil physicochemical properties, keystone taxa and degradation genes |
外源添加降解基因及降解菌群后,土壤中1,4-二氯苯降解效率显著提升,且外源添加降解菌群后降解效率提升最快;同时,优势菌门尤其是与有机氯农药降解相关的菌群环境丰度显著上升,菌群之间共存关系被显著促进;外源添加降解基因或降解菌群后,土壤中关键物种及降解基因环境丰度显著上升,且添加降解菌群后其上升幅度更显著。本研究深入阐释了外源添加降解基因及菌群后土著菌群提升降解潜能抵御农药污染胁迫响应机制,提出操控关键物种降解效率可为缩减农药降解进程提供借鉴,从而为土壤污染生物治理提供新科学思路。
| [1] |
赵玲, 滕应, 骆永明. 我国有机氯农药场地污染现状与修复技术研究进展[J]. 土壤, 2018, 50(3): 435-445 DOI:10.13758/j.cnki.tr.2018.03.001 (  0) 0) |
| [2] |
Kuang L H, Hou Y Z, Huang F Q, et al. Pesticide residues in breast milk and the associated risk assessment: A review focused on China[J]. Science of the Total Environment, 2020, 727: 138412 DOI:10.1016/j.scitotenv.2020.138412 (  0) 0) |
| [3] |
Hu G J, Liu H, Chen C, et al. An integrated geospatial correlation analysis and human health risk assessment approach for investigating abandoned industrial sites[J]. Journal of Environmental Management, 2021, 293: 112891 DOI:10.1016/j.jenvman.2021.112891 (  0) 0) |
| [4] |
Pant R, Pandey P, Kotoky R. Rhizosphere mediated biodegradation of 1, 4-dichlorobenzene by plant growth promoting rhizobacteria of Jatropha curcas[J]. Ecological Engineering, 2016, 94: 50-56 DOI:10.1016/j.ecoleng.2016.05.079 (  0) 0) |
| [5] |
Tananaki C, Zotou A, Thrasyvoulou A. Determination of 1, 2-dibromoethane, 1, 4-dichlorobenzene and naphthalene residues in honey by gas chromatography-mass spectrometry using purge and trap thermal desorption extraction[J]. Journal of Chromatography A, 2005, 1083(1/2): 146-152 (  0) 0) |
| [6] |
高寒, 陈娟, 王沛芳, 等. 农药污染土壤的生物强化修复技术研究进展[J]. 土壤, 2019, 51(3): 425-433 DOI:10.13758/j.cnki.tr.2019.03.002 (  0) 0) |
| [7] |
Barba S, Villaseñor J, Rodrigo M A, et al. Biostimulation versus bioaugmentation for the electro-bioremediation of 2, 4-dichlorophenoxyacetic acid polluted soils[J]. Journal of Environmental Management, 2021, 277: 111424 DOI:10.1016/j.jenvman.2020.111424 (  0) 0) |
| [8] |
Raimondo E E, Saez J M, Aparicio J D, et al. Coupling of bioaugmentation and biostimulation to improve lindane removal from different soil types[J]. Chemosphere, 2020, 238: 124512 DOI:10.1016/j.chemosphere.2019.124512 (  0) 0) |
| [9] |
Sun G D, Du Y, Yin J X, et al. Response of microbial communities to different organochlorine pesticides (OCPs) contamination levels in contaminated soils[J]. Chemosphere, 2019, 215: 461-469 DOI:10.1016/j.chemosphere.2018.09.160 (  0) 0) |
| [10] |
Wu M L, Wu J L, Zhang X H, et al. Effect of bioaugmentation and biostimulation on hydrocarbon degradation and microbial community composition in petroleum-contaminated loessal soil[J]. Chemosphere, 2019, 237: 124456 DOI:10.1016/j.chemosphere.2019.124456 (  0) 0) |
| [11] |
Borowski S, Matusiak K, Powałowski S, et al. A novel microbial-mineral preparation for the removal of offensive odors from poultry manure[J]. International Biodeterioration & Biodegradation, 2017, 119: 299-308 (  0) 0) |
| [12] |
Cycoń M, Mrozik A, Piotrowska-Seget Z. Bioaugmentation as a strategy for the remediation of pesticide-polluted soil: A review[J]. Chemosphere, 2017, 172: 52-71 DOI:10.1016/j.chemosphere.2016.12.129 (  0) 0) |
| [13] |
Liao Q H, Liu H, Lu C, et al. Root exudates enhance the PAH degradation and degrading gene abundance in soils[J]. Science of the Total Environment, 2021, 764: 144436 DOI:10.1016/j.scitotenv.2020.144436 (  0) 0) |
| [14] |
Bagi A, Knapik K, Baussant T. Abundance and diversity of n-alkane and PAH-degrading bacteria and their functional genes - Potential for use in detection of marine oil pollution[J]. Science of the Total Environment, 2022, 810: 152238 DOI:10.1016/j.scitotenv.2021.152238 (  0) 0) |
| [15] |
Dong P Y, Wang H, Fang T T, et al. Assessment of extracellular antibiotic resistance genes (eARGs) in typical environmental samples and the transforming ability of eARG[J]. Environment International, 2019, 125: 90-96 DOI:10.1016/j.envint.2019.01.050 (  0) 0) |
| [16] |
Nihemaiti M, Yoon Y, He H, et al. Degradation and deactivation of a plasmid-encoded extracellular antibiotic resistance gene during separate and combined exposures to UV254 and radicals[J]. Water Research, 2020, 182: 115921 DOI:10.1016/j.watres.2020.115921 (  0) 0) |
| [17] |
Meziti A, Tsementzi D, Ar Kormas K, et al. Anthropogenic effects on bacterial diversity and function along a river-to-estuary gradient in Northwest Greece revealed by metagenomics[J]. Environmental Microbiology, 2016, 18(12): 4640-4652 DOI:10.1111/1462-2920.13303 (  0) 0) |
| [18] |
Shannon P, Markiel A, Ozier O, et al. Cytoscape: a software environment for integrated models of biomolecular interaction networks[J]. Genome Research, 2003, 13(11): 2498-2504 DOI:10.1101/gr.1239303 (  0) 0) |
| [19] |
Li B B, Roley S S, Duncan D S, et al. Long-term excess nitrogen fertilizer increases sensitivity of soil microbial community to seasonal change revealed by ecological network and metagenome analyses[J]. Soil Biology and Biochemistry, 2021, 160: 108349 DOI:10.1016/j.soilbio.2021.108349 (  0) 0) |
| [20] |
Banerjee S, Schlaeppi K, van der Heijden M G A. Keystone taxa as drivers of microbiome structure and functioning[J]. Nature Reviews Microbiology, 2018, 16(9): 567-576 DOI:10.1038/s41579-018-0024-1 (  0) 0) |
| [21] |
Fan X Y, Fu Y, Nie Y X, et al. Keystone taxa-mediated bacteriome response shapes the resilience of the paddy ecosystem to fungicide triadimefon contamination[J]. Journal of Hazardous Materials, 2021, 417: 126061 DOI:10.1016/j.jhazmat.2021.126061 (  0) 0) |
| [22] |
Banerjee S, Walder F, Büchi L, et al. Agricultural intensification reduces microbial network complexity and the abundance of keystone taxa in roots[J]. The ISME Journal, 2019, 13(7): 1722-1736 DOI:10.1038/s41396-019-0383-2 (  0) 0) |
| [23] |
邱丽丽, 李丹丹, 张佳宝, 等. 基于共现网络的关键微生物对秸秆还田土壤小麦产量的影响[J]. 土壤学报, 2022(待发表).
(  0) 0) |
| [24] |
Bastian M, Heymann S, Jacomy M. Gephi: An open source software for exploring and manipulating networks[J]. Third Int. AAAI Conf. Weblogs Soc. Media, 2009, 361-362 (  0) 0) |
| [25] |
Wasmund K, Pelikan C, Schintlmeister A, et al. Genomic insights into diverse bacterial taxa that degrade extracellular DNA in marine sediments[J]. Nature Microbiology, 2021, 6(7): 885-898 DOI:10.1038/s41564-021-00917-9 (  0) 0) |
| [26] |
Picariello E, Baldantoni D, Muniategui-Lorenzo S, et al. A synthetic quality index to evaluate the functional stability of soil microbial communities after perturbations[J]. Ecological Indicators, 2021, 128: 107844 DOI:10.1016/j.ecolind.2021.107844 (  0) 0) |
| [27] |
Ran Y G, Liu Y, Wu S J, et al. A higher river sinuosity increased riparian soil structural stability on the downstream of a dammed river[J]. Science of the Total Environment, 2022, 802: 149886 DOI:10.1016/j.scitotenv.2021.149886 (  0) 0) |
| [28] |
Levy-Booth D J, Campbell R G, Gulden R H, et al. Cycling of extracellular DNA in the soil environment[J]. Soil Biology and Biochemistry, 2007, 39(12): 2977-2991 DOI:10.1016/j.soilbio.2007.06.020 (  0) 0) |
| [29] |
张铭, 蔡鹏, 吴一超, 等. 细菌胞外聚合物: 基于土壤生态功能的视角[J]. 土壤学报, 2022, 59(2): 308-323 (  0) 0) |
| [30] |
Coyte K Z, Schluter J, Foster K R. The ecology of the microbiome: Networks, competition, and stability[J]. Science, 2015, 350(6261): 663-666 DOI:10.1126/science.aad2602 (  0) 0) |
| [31] |
Xu Y F, Teng Y, Wang X M, et al. Exploring bacterial community structure and function associated with polychlorinated biphenyl biodegradation in two hydrogen-amended soils[J]. Science of the Total Environment, 2020, 745: 140839 DOI:10.1016/j.scitotenv.2020.140839 (  0) 0) |
| [32] |
Prapagdee B, Eiamphungporn W, Saenkham P, et al. Analysis of growth phase regulated KatA and CatE and their physiological roles in determining hydrogen peroxide resistance in Agrobacterium tumefaciens[J]. FEMS Microbiology Letters, 2004, 237(2): 219-226 (  0) 0) |
2. University of Chinese Academy of Sciences, Beijing 100049, China;
3. Jiangsu Environmental Engineering Technology Co., Ltd., Nanjing 210019, China;
4. Nanjing Institute of Environmental Sciences, Ministry of Ecology and Environment, Nanjing 210042, China
 2023, Vol. 55
2023, Vol. 55


