2. 安徽省宣城市宣州区农业农村局, 安徽宣城 242099;
3. 安徽中烟工业有限责任公司蚌埠卷烟厂, 安徽蚌埠 233010
微生物对于维持土壤生态系统功能和稳定性至关重要,土壤中多样化的微生物群落是决定农业生产力的要素之一[1-2]。环境条件对土壤微生物群落的组成具有决定性影响,包括不同的农业管理方式和土地利用类型等因素[3]。了解相似土地利用类型、农业管理土壤微生物群落之间的差异,有助于预测农业生态系统的动态和功能,并对保持微生物多样性至关重要。
大量研究表明,土壤细菌群落在相似的土地管理措施下,往往会变得更均质,群落间差异较小[4]。造成这种情况的原因可能是相似的土地管理降低了土壤环境异质性,从而降低了群落间的差异[5]。但微生物群落通常呈现倾斜的丰度分布,包括少数具有广泛分布的丰富分类群和众多分布有限的中等、稀有分类群,且不同细菌类群具有不同特性[6]。有研究表明,与丰富分类群相比,稀有分类群具有较窄的生态位宽度和较低的竞争能力,更容易受到外部因素的影响,而丰富分类群往往具有较宽的生态位宽度和较高的竞争能力,受外部因素的影响较小[7-9]。然而,目前仅了解相似的土地管理会造成整体的细菌群落均质化,但不同细菌亚群的结构及功能的差异尚不清楚。
本研究针对茶园、菜园、葡萄园和桃园4种园地土壤,利用16S rRNA基因扩增子Illumina Miseq测序技术,比较分析几种园地土壤中不同丰度细菌类群的结构和功能差异,这将有助于揭示土壤细菌亚群对不同土地利用方式的生态响应,以及阐明不同细菌类群对土地利用的敏感性和适应性。
1 材料与方法 1.1 样品采集不同园地土壤采样地点位于安徽省宣城市,该地区属于典型的亚热带湿润季风气候,年平均温度为17.8 ℃,年平均降雨量为1 700.7 mm。土壤样品于2021年10月采集,分别来自宣城市新丰农业有限公司菜园(VS)、宣城市绿品果园家庭农场葡萄园(GS)、宣城市绿丰家庭农场茶园(TS)和宣城市四季园果树种植家庭农场桃园(PS),上述园地均有5 □ 8年的种植历史。分别在50 m2范围内,采用五点取样法,采集0 □ 20 cm土层土壤样品,充分混匀后得到1个土样。每个类型园地分别设置3个重复,共计12个样品。采集的新鲜样品立即储存在无菌袋中,并在低温条件下运回实验室,用于DNA的提取。不同类型园地土壤基本理化性质见表 1。
|
|
表 1 土壤基本理化性质 Table 1 Soil basic physicochemical properties |
准确称取0.5 g新鲜土壤,使用FastDNATM Spin Kit(MP Biomedicals,美国)按照制造商的说明提取土壤中的总DNA。提取后,使用PowerClean DNA纯化试剂盒(PowerClean,美国)对DNA进行纯化,并使用NanoDrop ND-1000分光光度计(Thermo Scientific,美国)测量DNA的浓度和质量。经检测合格后,选择细菌16S rRNA基因的V4区域进行扩增,使用以下引物:515F(5'-GTGCCAGCMGCCGCGGT AA-3')和806R(5'-GGACTACHVGGGTWTCTAAT- 3')[10]。PCR条件如下:94 ℃预变性3 min;然后进行24个循环,循环条件为95 ℃变性5 s,57 ℃退火90 s,72 ℃延伸10 s;最后72 ℃延伸5 min。将PCR产物使用GeneJETTM Gel Extraction Kit (Thermo Scientific,美国)进行纯化,然后使用GENEWIZ公司的Illumina Miseq平台对纯化的产物进行测序。
1.3 高通量数据分析使用QIIME流程(版本1.9.0)[11]对原始测序序列进行处理。首先,通过Cutadapt(V1.9.1)进行质量过滤,剔除长度小于150 bp且平均碱基质量得分低于20的低质量测序reads,以获取高质量的序列。采用UCHIME方法对嵌合序列进行检测和删除。然后,利用QIIME中的UCLUST功能对序列进行聚类,设定相似性阈值为≥97%,生成可操作分类单元(OTUs)[12]。最后,利用Mothur算法对OTUs进行注释分析。在注释过程中,使用Silva数据库(https://www.arb-silva.de/)检索每个样本中的代表性分类单元的信息,并计算每个样本中的OTU数量。
1.4 统计和生物信息学分析将细菌群落根据相对丰度划分为丰富、中等和稀有类群。相对丰度大于0.1% 的OTUs被定义为“丰富类群”,相对丰度小于0.01% 的OTUs被定义为“稀有类群”,相对丰度在0.1% ~ 0.01% 的OTUs被定义为“中等类群”。
采用R studio中的不同库/包(运行R版本3.6.1)进行分析。为了评估细菌亚群的α多样性,使用R程序中的“Vegan”包[13]计算Chao1丰富度指数和Shannon多样性指数,并使用非参数的Wilcoxon秩和检验进行方差分析[14];基于Bray-Curtis距离进行主坐标(PCoA)分析,揭示细菌亚群的β多样性组成差异[15];通过Kruskal-Wallis整体检验,对群落组成差异进行评估,并使用Wilcoxon秩和检验进行组间两两比较[16];聚类树的构建使用了基于Bray-Curtis距离矩阵的UPGMA层级聚类方法[17];使用FAPROTAX(http://www.loucalab.com/archive/FAPROTAX/)预测不同土地利用下土壤细菌的群落功能[18]。
2 结果 2.1 不同园地土壤细菌亚群多样性如图 1所示,在未将细菌细分为细菌亚群时,除桃园和茶园之间的Shannon多样性指数具有显著差异外,其他类型园地之间的Chao1丰富度指数和Shannon多样性指数没有显著差异。
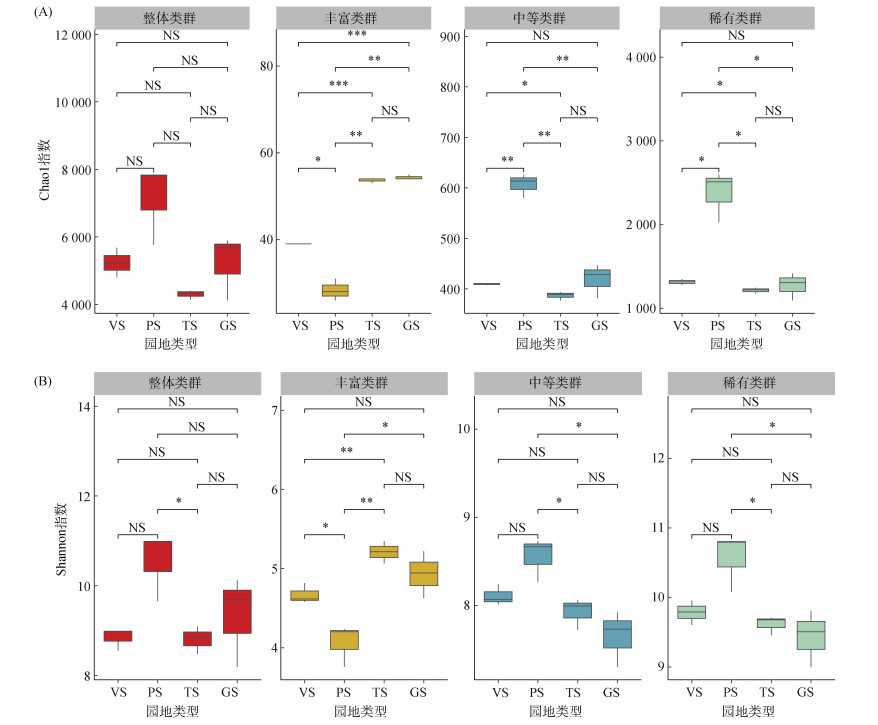
|
(图中***、**、*分别表示显著性差异达P < 0.001、P < 0.01、P < 0.05水平,NS表示无显著差异;下同) 图 1 不同园地土壤微生物α多样性Chao1指数(A)和Shannon指数(B) Fig. 1 Microbial alpha diversity indices Chao1 (A) and Shannon (B) in different garden soils |
将细菌分为不同亚群后,不同类型园地中的丰富、中等和稀有类群的α-多样性存在显著差异。对于丰富类群,茶园和葡萄园中的多样性较高,而桃园和菜园中的多样性较低。对于中等和稀有类群,桃园中的多样性较高,而菜园、茶园和葡萄园中的多样性较低。菜园土壤中的3个细菌亚群的多样性均较低。
2.2 不同园地土壤细菌亚群结构基于Bray-Curtis相似性矩阵的主坐标分析(PCoA)显示,不同园地类型下的土壤细菌亚群结构存在差异(图 2)。ANOSIM分析显示,4种不同园地样本之间群落组成变化显著(丰富类群,r=0.754,P=0.001;中等类群,r=0.706,P=0.001;稀有类群,r=0.695,P=0.001)。

|
图 2 不同园地土壤微生物丰富(A、D)、中等(B、E)和稀有(C、F)亚群OTU水平PCoA和亚群组成 Fig. 2 OTU level PCoA and compositions of microbial abundant (A, D), medium(B, E) and rare sub-communities in different garden soils |
尽管微生物主要的门种类和丰度在丰富、中等和稀有类群中存在差异,但在门水平上主要包括变形菌门(Proteobacteria)、酸杆菌门(Acidobacteriota)和绿弯菌门(Chloroflexi)。此外,利用UPGMA层级聚类构建聚类树探究细菌群落的组成关系发现,在丰富类群中,葡萄园和桃园的土壤细菌群落组成相较于菜园和茶园更为相似;而对于中等和稀有类群,葡萄园和菜园的土壤细菌群落组成相较于桃园和茶园更为相似。
2.3 不同园地土壤细菌亚群组成差异在门水平上,对4种园地土壤样本进行Kruskal- Wallis检验及Wilcoxon秩和检验,筛选丰度存在显著差异的微生物,结果(图 3)表明,不同细菌亚群的主要门类在4种园地土壤中表现出不同趋势。在丰富类群中,排名前10位的细菌门中有8个门在4种园地土壤中具有显著性差异,但拟杆菌门(Bacteroidetes)和硝化螺旋菌门(Nitrospirae)无显著差异;在中等类群中,有6个门具有显著性差异,但酸杆菌门(Acidobacteria)、放线菌门(Actinobacteria)、Patescibacteria和棒状杆菌门(Rokubacteria)无显著差异;在稀有类群中,有6个门具有显著性差异,但变形菌门(Proteobacteria)、酸杆菌门、放线菌门和Patescibacteria无显著差异。这些结果表明细菌亚群在不同园地具有不同的群落结构,且中等和稀有类群相对于丰富类群变化较小。

|
图 3 不同园地土壤微生物丰富(A)、中等(B)和稀有(C)亚群前10位门水平差异 Fig. 3 Variations in top 10 phylum levels of microbial abundant (A), medium(B) and rare (C) sub-communities in different garden soils |
本研究还观察到,细菌亚群中门的丰度变化可能与园地类型相关。如,绿弯菌门(Chloroflexi)和酸杆菌门在茶园的3个细菌亚群均具有较高丰度,而变形菌门具有相对较低的丰度;放线菌门在桃园的3个细菌亚群均具有较低丰度,而变形菌门具有相对较高的丰度。
2.4 不同园地土壤细菌亚群功能分异利用FAPROTAX对不同土地利用类型下土壤细菌亚群功能进行预测,发现3个细菌亚群功能在不同类型园地存在显著差异(图 4)。选择前10个功能进行比较,发现3个亚群中的好氧化能异养(aerobic_chemoheterotrophy)、化能异养(chemoheterotrophy)、硝化作用(nitrification)和好氧氨氧化(aerobic_ammonia_ oxidation)这4个功能均占据较高的比例。不同园地类型下,稀有和中等类群功能差异比丰富类群更为显著。

|
图 4 不同园地土壤微生物丰富(A、D、G)、中等(B、E、H)和稀有亚群(C、F、I)的功能变化 Fig. 4 Functional changes of microbial abundant (A, D, G), medium (B, E, H) and rare sub-communities (C, F, I) in different garden soils |
在不同类型园地中,功能预测还显示普遍性的一致结果,即稀有和中等类群的功能多样性往往高于丰富类群;丰富类群主要参与碳(C)、氮(N)和硫(S)的循环过程,而中等和稀有类群参与的循环更加多样化,包括碳、氮、硫、铁(Fe)、锰(Mn)和氢(H)的循环。
3 讨论 3.1 不同园地土壤细菌亚群多样性指数变化揭示不同园地土壤细菌多样性,对于农业可持续发展和生态环境保护具有重要意义[19]。本研究结果显示,在未进行区分微生物亚群时,不同类型园地土壤微生物多样性整体呈现无显著性差异。不同园地长期的人为趋同影响可能是导致土壤微生物多样性之间基本无差异的原因。在长期类似的农业管理实践下,不同土地利用类型的土壤可能具有一些共同的环境特征,如土壤pH、湿度、有机质含量、养分状况等,从而降低了环境异质性[20]。这些共同的环境特征可能为微生物提供了相似的生存条件,导致土壤微生物群落趋于稳定状态,形成了接近生态平衡的状态,在这种情况下,微生物多样性之间的差异可能较小,因为各种微生物群落已经适应了特定土地利用方式带来的环境变化[21-22]。
然而,将微生物群落区分为不同的亚群后,不同亚群在不同的土地利用下具有显著性差异。虽然长期的趋同影响可以降低环境异质性,使细菌群落趋于均质化方向发展,但特定的园地土壤仍然具有特定的生态位[7-8]。造成微生物亚群多样性在不同园地的变化可能与土壤类型和植被类型相关。不同土地利用类型通常伴随着不同的植被类型,而不同植被类型为微生物提供了不同的根际环境和有机物输入,植物根系分泌物和残留物可以影响土壤微生物亚群的组成及多样性[23-25]。
此外,不同园地的土壤管理行为也会直接或间接地影响微生物类群。例如,耕作、施用化肥、农药和有机肥料等农业管理行为会改变土壤中的养分水平和化学性质,从而影响微生物的群落结构[26-27]。施用肥料除了直接影响土壤化学成分变化,引起土壤微生物活性、土壤微生物群落结构改变外,还能通过改变土壤的物理性状,影响地上植被的生长状况,从而间接地影响土壤微生物群落结构。据报道,大量施肥通常会促使微生物群落均一化,进而导致微生物多样性降低[28];耕作会改变土壤结构,继而破坏原有的土壤环境和微生物生境,减少微生物生活的空间,降低微生物多样性[29];农药使用会抑制微生物的生长和繁殖,对某些微生物具有选择性杀灭作用,导致微生物群落的多样性下降[30]。具体而言,菜园通常需要频繁地耕作、施用化肥和农药,以增加产量及控制病虫害和杂草,而这些管理行为会改变土壤结构和微生物生境,导致微生物多样性的下降。
3.2 不同园地土壤细菌亚群的组成分异不同土地利用方式下,土壤微生物群落的组成通常不一致,这主要归因于土壤物理化学性质的变化、植被类型和根系分泌物的影响、土壤管理行为以及生态位的竞争和选择等因素的综合作用[31-32]。
本研究通过UPGMA层级聚类构建聚类树,探究细菌群落的组成关系,结果表明,中等和稀有类群在不同园地类型下保持一致。同时,对于主要细菌亚群的主要门类进行分析,发现丰富类群的细菌门变化大于中等和稀有类群。不同类型园地之间的相似管理可能会降低土壤间的异质性,使得中等和稀有类群更容易在不同园地类型之间共存,从而降低了不同园地细菌亚群的差异性。而丰度较高的类群往往在土壤微生物组成中占据主导地位,这些类群具备更高的资源利用能力、生长速率和适应性,使得它们能够在土壤环境中更好地竞争和繁殖[8]。丰度较高的类群通常具有较高的群落弹性和重建能力,当外界环境发生变化时,这些类群可以通过重新适应和重建其种群来恢复其丰度和功能,能够更快地适应新的环境条件并恢复其群落结构[33-34]。因此,即使在相似的土壤环境中,丰富类群可能仍然能够保持较高的差异。同时,不同园地植被类型和根系分泌物也会对土壤微生物群落产生直接影响,植被类型的差异会导致根系分泌物的化学成分和数量变化,从而影响土壤中细菌类群的分布和丰度[35]。特定的植被类型和根系分泌物可以为某些类群提供更有利的生存条件,促进它们的生长和繁殖,从而导致其在土壤微生物组成中的丰度较高[36]。这也同样会导致丰度较高的细菌类群在不同土地利用类型中差异较大。
此外,特定类群的增加可能与土壤类型和植物种类相关,本研究发现绿弯菌门(Chloroflexi)和酸杆菌门(Acidobacteria),与其他土壤相比,在茶园中具有较高丰度,这可能与茶树本身的特性密切相关。茶树原产中国云贵高原原始森林地区的酸性土壤,长期的系统发育使其逐渐形成了喜酸的遗传特性,并且只有在酸性土壤中才能正常生长[37]。因此,茶园土壤通常具有一定的酸性,酸杆菌门在茶树土壤中表现出较好的适应性,并能够利用有机质作为能源和营养源快速生长[38]。另外,一些研究表明,绿弯菌门还与茶树的共生关系有关,茶园土壤中的绿弯菌门可以与茶树根系形成共生关系,这种共生关系可以提高茶树的养分吸收效率,增强其抗病能力和适应环境的能力[39]。
3.3 不同园地土壤细菌亚群功能分析微生物群落的功能对于土壤的养分循环、生物保护、土壤结构形成以及生态系统稳定性具有重要的意义。理解和保护微生物群落的功能对于土壤的肥力、健康和可持续管理至关重要[35, 40]。
本研究通过FAPROTAX预测分析发现,在不同园地类型下,土壤细菌稀有和中等类群的功能差异大于丰富类群。这意味着在不同土地利用类型下,丰富类群的功能变化相对较小,而稀有和中等类群在功能上表现出更大的差异。然而,与功能差异不同的是,本研究结果显示微生物群落的组成差异中丰富类群的变化大于中等和稀有类群。这与本研究预期的结果相反,因为微生物的组成变化一般会导致功能发生变化,但更大的组成差异,并未导致更大的功能差异。丰度较高的微生物类群通常包含了多个物种和亚群,这些物种可能在功能上存在重叠和冗余,这可能与细菌群落的功能冗余有关[41]。在土壤生态系统中,丰富类群扮演主要功能的角色,当环境条件发生变化时,具有相似功能的微生物群体可以替代受到影响的群体,维持生态系统的功能稳定性和适应性[42]。相比之下,稀有和中等丰度的微生物亚群可能占据了较为特定的生态位空间,并发挥特定的功能,它们可能在土壤中承担一些特殊的生物化学过程,如降解、稀有物质的转化、抗生素产生等[43]。不同园地土壤微生物的生态位空间可能会发生较大的变化和分隔,导致稀有和中等丰度亚群的功能差异更加显著[44]。
4 结论本研究对4种园地土壤进行细菌群落差异研究,发现整体细菌群落多样性相似,这表明不同类型园地对微生物多样性的整体影响较小,可能是因为相似的土地管理措施使得土壤环境在某种程度上保持了相对稳定的状态。但不同丰度的细菌类群之间存在显著差异。在微生物群落组成方面,中等和稀有细菌类群的差异性相较于丰富类群较小。然而,由于细菌群落功能的冗余性存在,丰富类群相较于中等和稀有类群功能差异较小。表明尽管丰富亚群在组成上存在较大差异,但不同的丰富亚群可能在维持土壤生态系统功能方面可能具有一定的替代性。这些结果将有助于更好地理解土地利用对土壤微生物群落的影响,并为可持续土地管理和农业环境保护提供科学依据。
| [1] |
Wagg C, Bender S F, Widmer F, et al. Soil biodiversity and soil community composition determine ecosystem multifunctionality[J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(14): 5266-5270 (  0) 0) |
| [2] |
Allan E, Manning P, Alt F, et al. Land use intensification alters ecosystem multifunctionality via loss of biodiversity and changes to functional composition[J]. Ecology Letters, 2015, 18(8): 834-843 DOI:10.1111/ele.12469 (  0) 0) |
| [3] |
Liu H, Han H M, Zhang R L, et al. Biogeographic patterns of fungal sub-communities under different land-use types in subtropical China[J]. Journal of Fungi, 2023, 9(6): 646 DOI:10.3390/jof9060646 (  0) 0) |
| [4] |
Banerjee S, Walder F, Büchi L, et al. Agricultural intensification reduces microbial network complexity and the abundance of keystone taxa in roots[J]. The ISME Journal, 2019, 13(7): 1722-1736 DOI:10.1038/s41396-019-0383-2 (  0) 0) |
| [5] |
Stover H J, Henry H A L. Soil homogenization and microedges: Perspectives on soil-based drivers of plant diversity and ecosystem processes[J]. Ecosphere, 2018, 9(6): e02289 DOI:10.1002/ecs2.2289 (  0) 0) |
| [6] |
Pedrós-Alió C. The rare bacterial biosphere[J]. Annual Review of Marine Science, 2012, 4: 449-466 DOI:10.1146/annurev-marine-120710-100948 (  0) 0) |
| [7] |
Li P, Xue Y, Shi J L, et al. The response of dominant and rare taxa for fungal diversity within different root environments to the cultivation of Bt and conventional cotton varieties[J]. Microbiome, 2018, 6(1): 184 DOI:10.1186/s40168-018-0570-9 (  0) 0) |
| [8] |
Jiao S, Lu Y H. Abundant fungi adapt to broader environmental gradients than rare fungi in agricultural fields[J]. Global Change Biology, 2020, 26(8): 4506-4520 DOI:10.1111/gcb.15130 (  0) 0) |
| [9] |
Zhang C Z, Liu H, Liu S L, et al. Response of fungal sub-communities in a maize-wheat rotation field subjected to long-term conservation tillage management[J]. Frontiers in Microbiology, 2022, 13: 829152 DOI:10.3389/fmicb.2022.829152 (  0) 0) |
| [10] |
Kozich J J, Westcott S L, Baxter N T, et al. Development of a dual-index sequencing strategy and curation pipeline for analyzing amplicon sequence data on the MiSeq Illumina sequencing platform[J]. Applied and Environmental Microbiology, 2013, 79(17): 5112-5120 DOI:10.1128/AEM.01043-13 (  0) 0) |
| [11] |
Caporaso J G, Lauber C L, Walters W A, et al. Ultra-high- throughput microbial community analysis on the Illumina HiSeq and MiSeq platforms[J]. The ISME Journal, 2012, 6(8): 1621-1624 DOI:10.1038/ismej.2012.8 (  0) 0) |
| [12] |
Edgar R C, Haas B J, Clemente J C, et al. UCHIME improves sensitivity and speed of chimera detection[J]. Bioinformatics, 2011, 27(16): 2194-2200 DOI:10.1093/bioinformatics/btr381 (  0) 0) |
| [13] |
Oksanen J, et al. Vegan: the community ecology package. The Comprehensive R Archive Network. https://cran.r-project.org/web/packages/vegan/vegan.pdf.
(  0) 0) |
| [14] |
Cuzick J. A wilcoxon-type test for trend[J]. Statistics in Medicine, 1985, 4(1): 87-90 DOI:10.1002/sim.4780040112 (  0) 0) |
| [15] |
Anderson M J, Willis T J. Canonical analysis of principal coordinates: A useful method of constrained ordination for ecology[J]. Ecology, 2003, 84(2): 511-525 DOI:10.1890/0012-9658(2003)084[0511:CAOPCA]2.0.CO;2 (  0) 0) |
| [16] |
McKight P E, Najab J. Kruskal-wallis test[C]//The corsini encyclopedia of psychology. John Wiley & Sons, Inc, 2010.
(  0) 0) |
| [17] |
Gronau I, Moran S. Optimal implementations of UPGMA and other common clustering algorithms[J]. Information Processing Letters, 2007, 104(6): 205-210 DOI:10.1016/j.ipl.2007.07.002 (  0) 0) |
| [18] |
Louca S, Parfrey L W, Doebeli M. Decoupling function and taxonomy in the global ocean microbiome[J]. Science, 2016, 353(6305): 1272-1277 DOI:10.1126/science.aaf4507 (  0) 0) |
| [19] |
Sabir M S, Shahzadi F, Ali F, et al. Comparative effect of fertilization practices on soil microbial diversity and activity: An overview[J]. Current Microbiology, 2021, 78(10): 3644-3655 DOI:10.1007/s00284-021-02634-2 (  0) 0) |
| [20] |
Shen W S, Hu M C, Qian D, et al. Microbial deterioration and restoration in greenhouse-based intensive vegetable production systems[J]. Plant and Soil, 2021, 463(1): 1-18 (  0) 0) |
| [21] |
Gossner M M, Lewinsohn T M, Kahl T, et al. Land-use intensification causes multitrophic homogenization of grassland communities[J]. Nature, 2016, 540(7632): 266-269 DOI:10.1038/nature20575 (  0) 0) |
| [22] |
de Graaff M A, Hornslein N, Throop H L, et al. Effects of agricultural intensification on soil biodiversity and implications for ecosystem functioning: A meta-analysis[J]. Advances in Agronomy, 2019, 155: 1-44 (  0) 0) |
| [23] |
Baudoin E, Benizri E, Guckert A. Impact of artificial root exudates on the bacterial community structure in bulk soil and maize rhizosphere[J]. Soil Biology and Biochemistry, 2003, 35(9): 1183-1192 DOI:10.1016/S0038-0717(03)00179-2 (  0) 0) |
| [24] |
Doornbos R F, van Loon L C, Bakker P A H M. Impact of root exudates and plant defense signaling on bacterial communities in the rhizosphere. A review[J]. Agronomy for Sustainable Development, 2012, 32(1): 227-243 DOI:10.1007/s13593-011-0028-y (  0) 0) |
| [25] |
Dhungana I, Kantar M B, Nguyen N H. Root exudate composition from different plant species influences the growth of rhizosphere bacteria[J]. Rhizosphere, 2023, 25: 100645 DOI:10.1016/j.rhisph.2022.100645 (  0) 0) |
| [26] |
Gajda A M, Czyż E A, Dexter A R, et al. Effects of different soil management practices on soil properties and microbial diversity[J]. International Agrophysics, 2018, 32(1): 81-91 DOI:10.1515/intag-2016-0089 (  0) 0) |
| [27] |
Kraut-Cohen J, Zolti A, Shaltiel-Harpaz L, et al. Effects of tillage practices on soil microbiome and agricultural parameters[J]. The Science of the Total Environment, 2020, 705: 135791 DOI:10.1016/j.scitotenv.2019.135791 (  0) 0) |
| [28] |
Chen R R, Zhong L H, Jing Z W, et al. Fertilization decreases compositional variation of paddy bacterial community across geographical gradient[J]. Soil Biology and Biochemistry, 2017, 114: 181-188 DOI:10.1016/j.soilbio.2017.07.013 (  0) 0) |
| [29] |
Wang Y, Li C Y, Tu C, et al. Long-term no-tillage and organic input management enhanced the diversity and stability of soil microbial community[J]. Science of the Total Environment, 2017, 609: 341-347 DOI:10.1016/j.scitotenv.2017.07.053 (  0) 0) |
| [30] |
Devi Y B, Meetei T T, Kumari N. Impact of pesticides on soil microbial diversity and enzymes: A review[J]. International Journal of Current Microbiology and Applied Sciences, 2018, 7(6): 952-958 DOI:10.20546/ijcmas.2018.706.113 (  0) 0) |
| [31] |
Jangid K, Williams M A, Franzluebbers A J, et al. Land-use history has a stronger impact on soil microbial community composition than aboveground vegetation and soil properties[J]. Soil Biology and Biochemistry, 2011, 43(10): 2184-2193 DOI:10.1016/j.soilbio.2011.06.022 (  0) 0) |
| [32] |
Schmid M W, van Moorsel S J, Hahl T, et al. Effects of plant community history, soil legacy and plant diversity on soil microbial communities[J]. Journal of Ecology, 2021, 109(8): 3007-3023 DOI:10.1111/1365-2745.13714 (  0) 0) |
| [33] |
Tilman D, Downing J A. Biodiversity and stability in grasslands[J]. Nature, 1994, 367(6461): 363-365 DOI:10.1038/367363a0 (  0) 0) |
| [34] |
Hooper D U, Adair E C, Cardinale B J, et al. A global synthesis reveals biodiversity loss as a major driver of ecosystem change[J]. Nature, 2012, 486(7401): 105-108 DOI:10.1038/nature11118 (  0) 0) |
| [35] |
Philippot L, Raaijmakers J M, Lemanceau P, et al. Going back to the roots: The microbial ecology of the rhizosphere[J]. Nature Reviews Microbiology, 2013, 11(11): 789-799 DOI:10.1038/nrmicro3109 (  0) 0) |
| [36] |
Bulgarelli D, Schlaeppi K, Spaepen S, et al. Structure and functions of the bacterial microbiota of plants[J]. Annual Review of Plant Biology, 2013, 64: 807-838 DOI:10.1146/annurev-arplant-050312-120106 (  0) 0) |
| [37] |
刘美雅, 伊晓云, 石元值, 等. 茶园土壤性状及茶树营养元素吸收、转运机制研究进展[J]. 茶叶科学, 2015, 35(2): 110-120 DOI:10.13305/j.cnki.jts.2015.02.002 (  0) 0) |
| [38] |
Fu H P, Li H A, Yin P, et al. Integrated application of rapeseed cake and green manure enhances soil nutrients and microbial communities in tea garden soil[J]. Sustainability, 2021, 13(5): 2967 DOI:10.3390/su13052967 (  0) 0) |
| [39] |
Vik U, Logares R, Blaalid R, et al. Different bacterial communities in ectomycorrhizae and surrounding soil[J]. Scientific Reports, 2013, 3: 3471 DOI:10.1038/srep03471 (  0) 0) |
| [40] |
Bardgett R D, van der Putten W H. Belowground biodiversity and ecosystem functioning[J]. Nature, 2014, 515(7528): 505-511 DOI:10.1038/nature13855 (  0) 0) |
| [41] |
Allison S D, Martiny J B H. Resistance, resilience, and redundancy in microbial communities[J]. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105(supplement_1): 11512-11519 (  0) 0) |
| [42] |
Louca S, Polz M F, Mazel F, et al. Function and functional redundancy in microbial systems[J]. Nature Ecology & Evolution, 2018, 2(6): 936-943 (  0) 0) |
| [43] |
Jousset A, Bienhold C, Chatzinotas A, et al. Where less may be more: How the rare biosphere pulls ecosystems strings[J]. The ISME Journal, 2017, 11(4): 853-862 DOI:10.1038/ismej.2016.174 (  0) 0) |
| [44] |
Lynch M D J, Neufeld J D. Ecology and exploration of the rare biosphere[J]. Nature Reviews Microbiology, 2015, 13(4): 217-229 DOI:10.1038/nrmicro3400 (  0) 0) |
2. Agricultural and Rural Bureau of Xuanzhou District, Xuancheng, Anhui 242099, China;
3. Bengbu Cigarette Factory, China Tobacco Anhui Industrial Co. Ltd., Bengbu, Anhui 233010, China
 2023, Vol. 55
2023, Vol. 55


