2. 江西省农业科学院土壤肥料与资源环境研究所, 南昌 330200;
3. 土壤与农业可持续发展重点实验室(中国科学院), 南京 211135;
4. 中国科学院东北地理与农业生态研究所, 长春 130102
土壤中存在大量磷素,然而80% 以上以难溶性有机磷形式存在,无法直接被植物利用。因此,磷缺乏也成为制约农业发展的重要因素。解磷菌是土壤生态系统的重要组成,其自身细胞对磷素的需求驱动了土壤难溶性磷的活化和形态转化[1],从而促进土壤磷循环,提高磷素的生物有效性。掌握环境中解磷菌资源的多样性和富集培养特性,有助于筛选和利用高效解磷菌,充分发挥其生理生态功能,缓解农田土壤缺磷现状。
解磷菌活化磷素的一个重要途径是通过分泌多种胞外酶(如碱性磷酸酶(ALP)等)降解植酸盐、磷脂、核酸、磷蛋白等含磷有机化合物,释放可溶性无机磷酸盐[2-3]。ALP仅由微生物产生,主要由phoA、phoX和phoD这3种同源基因编码,其中携带phoD基因的细菌在水生和陆地环境中无处不在,而携带phoA和phoX的细菌主要出现在水生栖息地[4-5]。因此,通常将phoD作为土壤有机磷转化关键微生物的分子标靶[6]。携带phoD基因的细菌群落通过ALP调控土壤有机磷矿化,已经在农田[7]、森林[6]和沉积物[8]等生境中得到证实。研究phoD基因相关细菌群落的可培养特性,有助于深入揭示与有机磷活化相关的土壤解磷微生物种类、多样性以及筛选富集策略。
平板培养法是一种经典的微生物分离筛选技术,通常使用不同营养成分的琼脂培养基对土壤中可培养微生物进行分离培养,然后根据微生物的菌落形态及其菌落数,计算微生物的数量及鉴定菌落的系统发育类型。然而,由于菌落观察和筛选具有主观性和随机性,会遗漏很多肉眼看不清的微小菌落,很难实现特定生态功能菌群的培养分离,特别是在数量上占弱势的一部分功能菌群[9-10],因此,基于平板培养法的大量研究多属于定性的描述过程,缺乏定量评估。不依赖于培养的高通量测序技术在近十年间迅猛发展,能够直接从样本中进行无偏倚的微生物DNA分析,更准确地反映微生物群落多样性特征[11-12]。然而,仅通过分子生物学手段不能直接获得菌株,在一定程度上限制了对菌株生理功能和实际的开发应用,不利于进一步获得能够执行或调控特定生理生态功能的微生物种质资源。若将高通量测序技术与传统平板培养法相结合,不仅有利于明确环境中的微生物资源特征,同时又可进一步解析可培养微生物类群参与的生态过程,为实现功能微生物的定向调控和综合利用奠定基础[13]。针对土壤解磷菌,利用特定培养基进行培养,对其特征功能基因phoD进行测序分析,则能更加全面、准确地评估土壤中可培养解磷菌种类和比例以及在不同培养基中富集差异。
基于以上,本研究以phoD基因为分子标靶,利用常规细菌培养基和两种解磷菌选择性培养基,通过传统平板培养法结合高通量测序分析技术,探究土壤可培养解磷菌多样性和群落组成,定量评价不同培养基对土壤可培养解磷菌群落的影响,以期为发掘和利用土壤解磷菌资源提供理论借鉴。
1 材料与方法 1.1 供试土壤供试土壤为河流冲积物母质发育的水稻土,取自江西新余市。采集耕层土壤,清除石块和植物残体等,过2 mm筛,混合均匀,4 ℃保存。土壤pH 5.68,有机碳20.59 g/kg,全氮2.17 g/kg,有效磷8.33 mg/kg。
1.2 培养基配制常规细菌培养基(BPM):牛肉膏3 g,蛋白胨10 g,氯化钠5 g,琼脂18 g,加蒸馏水定容至1 L。无机解磷菌培养基(IPM):葡萄糖10.0 g,硫酸铵0.5 g,氯化钠0.3 g,硫酸镁0.3 g,硫酸亚铁0.03 g,硫酸锰0.03 g,碳酸钙5.0 g,氯化钾0.3 g,磷酸三钙5.0 g,琼脂18 g,加蒸馏水定容至1 L。有机解磷菌培养基(OPM):用0.3 g卵磷脂代替磷酸三钙,其余成分和用量与IPM相同。最后,调节培养基pH至7.0 ~ 7.5,115 ℃灭菌30 min。待固体培养基稍冷却后制备无菌平板。
1.3 富集培养方法通过传统平板培养法进行土壤微生物富集培养,为避免遗漏代时较长的微生物,本研究进行连续3次传代后,收集所有可培养微生物菌落进行群落多样性分析。具体步骤如下:称取土壤1.0 g于100 mL无菌水,加入5 g灭菌玻璃珠,以200 r/min振荡30 min,制成土壤悬液;吸取100 μL土壤悬液,均匀涂布在培养基表面,在30 ℃避光倒置培养5 d,再将平板取出,吸取10 mL无菌水将培养基表面菌落分多次洗脱至50 mL无菌离心管中;然后,再吸取100 μL菌液使用平板涂布法接种传代培养,培养5 d后,以同样方式冲脱菌落和传代,直至收集到第三代菌落富集液。所有培养操作步骤均为无菌操作,设置4次生物学重复。
1.4 微生物DNA提取称取0.5 g土壤3份,采用FastDNA® Spin Kit for Soil (MP Bio)试剂盒提取土壤本底微生物DNA。吸取1 mL菌落富集液,采用E.Z.N.A.® Bacterial DNA Kit (Omega)试剂盒提取可培养微生物DNA。DNA保存于–20 ℃,用于后续分子分析。
1.5 IIIumina高通量测序分析由上海派森诺生物科技股份有限公司扩增土壤本底和菌落富集液中可培养微生物phoD基因(引物ALPS-F730/ALPS-1101[14])并建库,在Illumina测序系统上对其双端测序。根据barcode划分样本,去除barcode序列。通过Vsearch软件进行序列去噪,在置信度80% 和相似度95% 进行OTU(operational taxonomic units)聚类,计算样本α和β多样性,评估处理间多样性差异。再根据NCBI本地BLAST数据库(nt库)进行物种注释,在微生物门和属分类水平下比较各样品中微生物群落组成和相对丰度。相对丰度是以样本中各分类水平各类型微生物phoD序列数除以phoD序列总数来表示。测序结果分析主要在派森诺基因云平台(https://www.genescloud.cn)进行。
1.6 可培养解磷菌比例及富集倍数的计算提取土壤DNA,高通量测序微生物phoD基因,基于OTU聚类在各微生物分类水平进行物种注释,获得土壤本底DNA中解磷菌的系统发育信息和分类单元总数;根据前述分析方法,获得菌落富集物DNA中可培养解磷菌的分类信息和分类单元数。可培养解磷菌的比例=菌落富集物DNA中可培养解磷菌分类单元数/土壤本底DNA中解磷菌分类单元数。
基于phoD基因系统发育分类,在属水平下,分别获得某一类可培养解磷菌的相对丰度和土壤中相应解磷菌属的相对丰度。可培养解磷菌富集倍数=某一类可培养解磷菌的相对丰度/土壤中相应解磷菌属的相对丰度
1.7 数据分析采用Excel和SPSS Statistics 19软件进行基础数据分析。采用单因素方差分析(one-way ANOVA)和Duncan法进行方差分析和多重比较,差异显著性水平为P < 0.05。门、属水平下优势解磷菌类群的判断依据为相对丰度 > 1%。通过α多样性分析和基于Bray-Curtis距离算法的主成分聚类分析(Principal coordinates analysis,PCoA)确定OTU水平的解磷菌群落多样性和结构差异,通过ANOSIM法进行组间差异显著性分析。利用韦恩图(Venn diagram)探究不同样品之间共有和特有解磷菌类型。通过LEfSe(LDA effect size)分析和Wilcoxon检验得到不同处理样品之间门和属水平上存在差异的解磷菌类群。采用Origin Pro 2021制图。
2 结果与分析 2.1 土壤本底和可培养解磷菌phoD基因多样性针对样本DNA中phoD基因进行高通量测序,共获得962 176条高质量序列,平均64 145条/样。在95% 相似度水平对phoD序列进行OTU聚类,评估样本多样性。
α多样性以Shannon、Simpson、Chao1和Goods coverage指数进行评估。结果表明:原位土壤解磷菌多样性显著高于土壤可培养解磷菌,土壤本底解磷菌的Shannon和Simpson指数平均值分别为6.43和0.96,而可培养解磷菌Shannon和Simpson指数仅分别为0.11 ~ 3.11和0.02 ~ 0.83(图 1A、1B);针对3种不同成分培养基,可培养解磷菌Shannon和Simpson指数在IPM中最低,而在BPM和OPM中无显著差异。土壤本底解磷菌丰富度Chao 1指数为1 135,而可培养解磷菌Chao 1指数仅为51 ~ 106(图 1C),且在3种培养基中无显著差异。此外,Goods coverage指数表明本次测序结果能够较大程度覆盖样品中解磷菌种群,覆盖度可达98.3% ~ 99.9%。
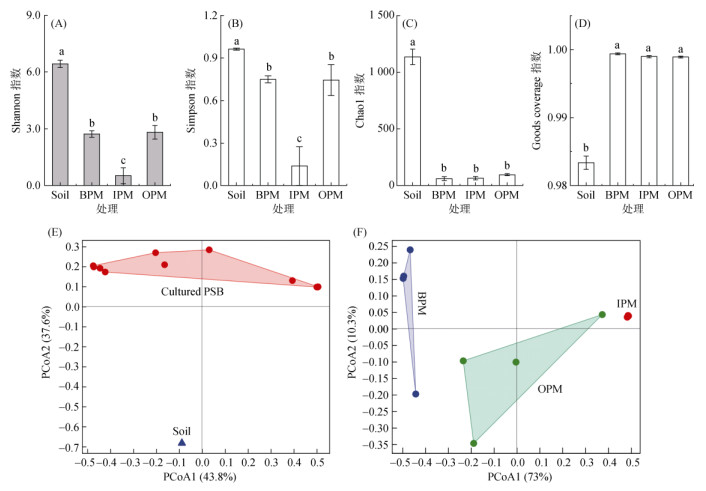
|
(Soil表示供试土壤;culturable PSB表示可培养解磷菌;BPM、IPM和OPM分别代表常规细菌培养基、无机磷细菌培养基和有机磷细菌培养基;小写字母不同表示差异达P < 0.05显著水平。下图同) 图 1 基于phoD基因的土壤和培养基中解磷菌群落多样性(OTU水平) Fig. 1 Diversity comparison of phoD-based phosphate-solubilizing bacteria community in soil and media at OTU level |
基于OTU水平的phoD基因PCoA主成分聚类分析(β多样性)和组间差异分析(ANOSIM)发现,土壤本底与可培养解磷菌群落结构发生了显著分异(P < 0.05,表 1),第1主成分上的贡献率达43.8%(图 1E)。针对可培养解磷菌开展主成分分析(图 1F)发现,3种培养基中可培养解磷菌群落分别聚集成簇,且彼此间均差异显著(P < 0.05,表 1),第1主成分上的贡献达73%。
|
|
表 1 不同处理间解磷菌群落相似性分析(ANOSIM) Table 1 Similarities analysis (ANOSIM) of PSB community structure between different groups |
基于phoD基因系统发育分类统计显示,在微生物门水平,土壤本底和可培养解磷菌种类数目差异较小;在微生物属水平,解磷菌种类数目在背景土壤中显著高于菌落富集物(图 2A)。3种培养基中,土壤可培养解磷菌的比例在门水平为52.5% ~ 82.5%,在属水平为17.3% ~ 38.1%(图 2B)。
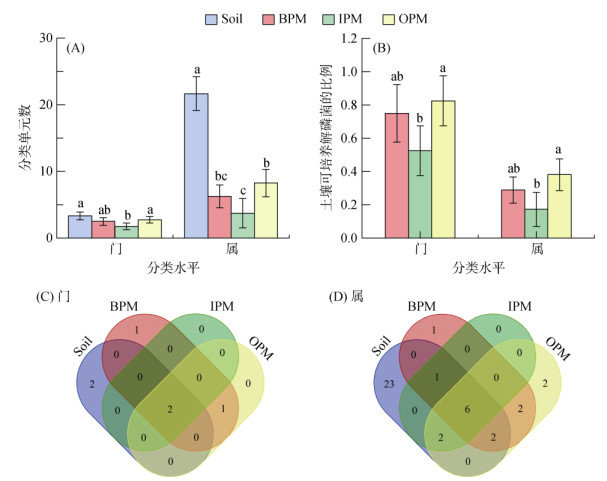
|
图 2 基于phoD基因的土壤本底和可培养解磷菌在微生物门和属水平的多样性以及共现规律 Fig. 2 phoD gene-based PSB diversity and co-occurrence in soil and enrichments from media at phylum and genus levels |
针对背景土壤和可培养解磷菌,开展共有和特有物种共现规律的韦恩图分析。在微生物门水平(图 2C),共检测到土壤本底土著解磷菌4门,3种培养基共获得可培养解磷菌4门;土壤本底与可培养解磷菌之间的共有门仅为2个。在属水平(图 2D),共检测到34属土壤本底土著解磷菌,3种培养基共获得可培养解磷菌14属;土壤本底与3种培养基中可培养解磷菌之间的共有属仅6个。
2.3 不同培养基中可培养解磷菌群落组成由图 3可知,在门水平(图 3A),土壤本底优势有机解磷菌为Proteobacteria(52.7%)和Actinobacteria (19.8%)。不同菌落富集物中,Proteobacteria均为最优势可培养解磷菌,占比95.8% ~ 99.9%。在属水平(图 3B),土壤本底与可培养解磷菌群落组成差异更大。首先,土壤本底与不同培养基可培养解磷菌共有属6个,分别为Pseudomonas、Bradyrhizobium、Cupriavidus、Sinorhizobium、Xanthomonas和Actinoplanes,但相对丰度在样本间差异较大;土壤本底解磷菌群落中Bradyrhizobium相对丰度最高(9.12%),其次为Amycolatopsis(2.95%);BPM主要富集了Pseudomonas(42.6%)、Bradyrhizobium(18.4%)、Sinorhizobium(1.50%)等;IPM富集优势解磷菌主要为Cupriavidus(99.7%);而OPM富集优势解磷菌,除Cupriavidus(36.8%)外,还包括Bradyrhizobium (7.54%)、Pseudomonas(7.62%)和Sinorhizobium(3.79%)等,其余属相对丰度均低于1%。另外,属水平未分类(unclassfied)phoD基因序列在土壤样本中占比高达59.6%,在菌落富集物中占比最高可达40.7%。
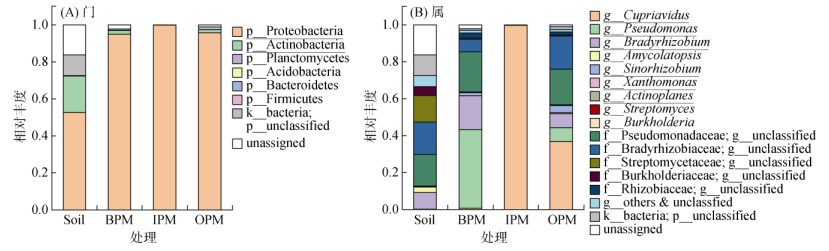
|
(图中下划线微生物分类单元为土壤和可培养解磷菌的共有微生物门或共有微生物属) 图 3 土壤和可培养解磷菌在微生物门(A)和属(B)水平的群落组成 Fig. 3 Community structures of PSB in soil and enrichments from different media at phylum (A) and genus (B) levels |
通过LEfSe差异分析(图 4A)表明,不同培养基富集了具有显著差异的解磷菌类群。图中不同颜色代表不同培养基来源,每个圆圈代表 1个解磷菌类群。在属水平,与其他两种培养基进行比较,BPM中显著差异的可培养解磷菌属具体表现在Bradyrhizobium、Pseudomonas和Xanthomonas;IPM中为Cupriavidus;而OPM具体为Burkholderia。以上5属微生物在不同培养基中具有富集优势(图 4B ~ 4E),Cupriavidus在IPM和OPM中分别富集1 672倍和616倍,显著高于BPM;Pseudomonas和Xanthomonas在BPM中分别富集588倍和54倍,显著高于IPM和OPM;Bradyrhizobium在BPM和OPM中分别富集2.0倍和0.8倍,显著高于IPM;Burkholderia仅在OPM中有富集,在土壤和其他两种培养基中均未检测到。
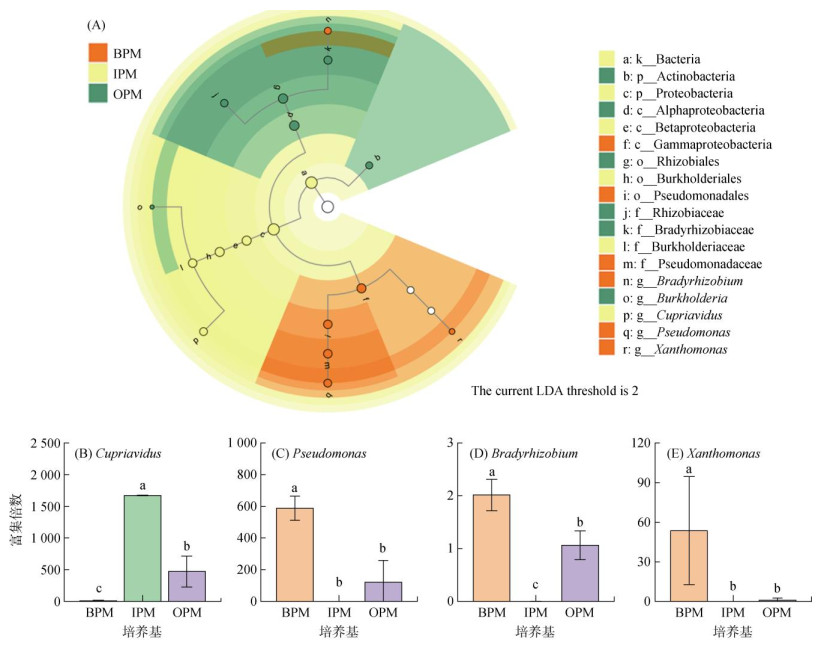
|
(图 4A为基于LEfSe分析的解磷菌群落差异,节点大小对应于该分类单元的平均相对丰度;空心节点代表组间差异不显著的分类单元,而其他颜色的节点则表明这些分类单元体现出显著的组间差异,且在该色所代表分组样本中丰度较高;字母则标识了组间存在显著差异的分类单元的名称。图 4B ~ 4E为显著差异的解磷菌属在不同培养基中的富集倍数比较) 图 4 不同培养基中土壤可培养解磷菌群落的差异比较 Fig. 4 Differences of soil culturable phosphate-solubilizing bacteria communities in different media |
可培养解磷菌群落多样性远低于原位土壤(图 1),造成这种差异的原因可能包括:(1)人工配置的培养基通常营养丰富,无法模拟微生物生长的真实土壤环境,无法满足所有目的微生物的营养需求及培养条件,导致很多原位土壤中本就存在的寡营养型微生物无法生长[15];而适当降低培养基营养物质浓度,可能提高可培养细菌的数量和种类[10, 16];(2)在利用营养丰富的培养基进行培养时,一些微生物快速生长,可能产生超出自身调节能力的过量超氧化物和自由基等“活性氧物质”,损伤细胞甚至加速死亡,降低微生物可培养性[17-19];(3)琼脂作为常用固体培养基凝固剂,对一些微生物具有一定程度的毒害作用,从而降低了可培养率。例如:琼脂培养基上的菌落数仅为结冷胶培养基上的1/10[20]。培养基配制时,琼脂和磷酸盐混合灭菌还可能产生H2O2,抑制了微生物生长[21]。
目前对解磷机理的认识仍不全面,限制了解磷菌的应用[22-23]。解磷菌分为能够溶解有机磷的有机磷细菌和溶解无机磷的无机磷细菌[8],但实际上难以明确区分。本研究证实一部分具有分解有机磷能力的细菌属可以在IPM上进行生长,同时具有降解难溶有机和无机磷化合物的能力,如Cupriavidus、Pseudomonas、Bradyrhizobium、Sinorhizobium、Xanthomonas和Actinoplanes(图 3B),尤其Cupriavidus在OPM和IPM上均表现出明显生长富集优势(图 4B)。除与磷源活化有关外[24-26],Cupriavidus还能够通过抗性基因表达抵抗金属毒性[27],帮助其适应高铝毒的酸性土壤[28]和其他污染土壤[29]。因此,Cupriavidus在开发高效、多功能解磷菌生物肥料方面具有良好应用前景。
培养基中磷源、碳源、氮源、生长因子等因素不同,影响解磷菌的解磷能力以及不同细胞的生长代谢。OPM与BPM为微生物提供了大量有机磷底物,有利于各种有机磷细菌生长,其上富集的解磷菌phoD基因多样性远远高于IPM(图 1A、1B)。经过3代富集培养,OPM和BPM分别富集含phoD基因解磷菌14属和12属,其中10属为共有解磷菌,Bradyrhizobium、Pseudomonas和Sinorhizobium相对丰度较高,表现出更强的有机磷活化和生长繁殖能力。此外,BPM含营养成分较OPM更为丰富,除磷脂外,还存在含磷蛋白、核酸、生长因子等,为平板表面其他“非”解磷菌的生长及菌落形成提供了条件[9-10],从而可能改变微生物菌落之间的群体感应过程[30],进而影响可培养解磷菌的生长繁殖状况和群落特征(图 1F)。
由于解磷机制复杂多样,解磷菌还存在多种溶磷基因[2],这些基因不一定同时包含于相同种、属菌株中。因此,针对phoD基因进行测序,仅能获得平板上包含phoD基因的可培养解磷菌菌落信息,可能遗漏某些具有其他解磷途径的解磷菌,后续有必要继续在分子水平对解磷菌溶磷机制的多样性进行深入探讨。
随着分子生物学技术手段的发展,利用高通量测序技术测定解磷菌多样性的研究逐渐增多,同时越来越多未分类解磷菌属和种被发现。出现这种现象主要是由于目前的可培养技术限制了微生物的分离鉴定,导致解磷微生物数据库不够完善,能够鉴定的物种仍十分有限。然而,相同种、属或同一菌株在不同环境条件下的解磷能力及机理也可能存在差别[31]。未来仍需要通过改进现有技术或发展新技术[15]以定向分离或鉴定更多的解磷菌属和种,为全面解析环境解磷微生物多样性和生理机制提供参考。
4 结论培养基成分不同,强烈影响不同类型解磷菌的解磷能力及细胞的生长代谢。以phoD基因为标靶,土壤可培养解磷菌α多样性远远低于原位土壤,群落结构也与土壤本底显著不同。BPM和OPM富集可培养解磷菌多样性高于IPM。不同培养基检测具有显著富集差异的可培养解磷菌共5属,Cupriavidus在IPM和OPM中显著富集,Pseudomonas、Bradyrhizobium和Xanthomonas在BPM和OPM中富集显著,Burkholderia仅在OPM中被检测。
| [1] |
Sarmah R, Sarma A K. Phosphate solubilizing microorganisms: A review[J]. Communications in Soil Science and Plant Analysis, 2023, 54(10): 1306-1315 DOI:10.1080/00103624.2022.2142238 (  0) 0) |
| [2] |
Rawat P, Das S, Shankhdhar D, et al. Phosphate-solubilizing microorganisms: Mechanism and their role in phosphate solubilization and uptake[J]. Journal of Soil Science and Plant Nutrition, 2021, 21(1): 49-68 DOI:10.1007/s42729-020-00342-7 (  0) 0) |
| [3] |
陶冬雪, 高英志. 土壤解磷微生物促进植物磷素吸收策略研究进展[J]. 生态学报, 2023, 43(11): 4390-4399 (  0) 0) |
| [4] |
Hu Y J, Xia Y H, Sun Q, et al. Effects of long-term fertilization on phoD-harboring bacterial community in Karst soils[J]. Science of the Total Environment, 2018, 628: 53-63 (  0) 0) |
| [5] |
Neal A L, Rossmann M, Brearley C, et al. Land-use influences phosphatase gene microdiversity in soils[J]. Environmental Microbiology, 2017, 19(7): 2740-2753 DOI:10.1111/1462-2920.13778 (  0) 0) |
| [6] |
Wan W J, He D L, Li X, et al. Adaptation of phoD-harboring bacteria to broader environmental gradients at high elevations than at low elevations in the Shennongjia primeval forest[J]. Geoderma, 2021, 401: 115210 DOI:10.1016/j.geoderma.2021.115210 (  0) 0) |
| [7] |
Wan W J, Liu S, Li X, et al. Dispersal limitation driving phoD-harboring bacterial community assembly: A potential indicator for ecosystem multifunctionality in long-term fertilized soils[J]. Science of the Total Environment, 2021, 754: 141960 DOI:10.1016/j.scitotenv.2020.141960 (  0) 0) |
| [8] |
马凯, 王效昌, 谢嘉慧, 等. 沉积物解磷菌的研究进展: 分布、解磷能力及功能基因[J]. 农业资源与环境学报, 2023, 40(2): 280-290 (  0) 0) |
| [9] |
李凯凯, 曹伟伟, 文昌丽, 等. 基于高通量测序的稀释平板计数细菌群落变化研究[J]. 微生物学报, 2022, 62(11): 4447-4464 (  0) 0) |
| [10] |
文昌丽, 曹伟伟, 唐雪莲, 等. 基于高通量测序的连续传代富集土壤可培养菌菌群变化规律研究[J]. 生态与农村环境学报, 2023, 39(1): 123-135 (  0) 0) |
| [11] |
Quail M A, Kozarewa I, Smith F, et al. A large genome center's improvements to the Illumina sequencing system[J]. Nature Methods, 2008, 5(12): 1005-1010 DOI:10.1038/nmeth.1270 (  0) 0) |
| [12] |
夏围围, 贾仲君. 高通量测序和DGGE分析土壤微生物群落的技术评价[J]. 微生物学报, 2014, 54(12): 1489-1499 (  0) 0) |
| [13] |
喻江, 王淳, 龙永, 等. 鄱阳湖湿地细菌群落多样性和可培养细菌功能基因丰度[J]. 环境科学, 2024, 45(4): 2223-2232 (  0) 0) |
| [14] |
Fraser T D, Lynch D H, Bent E, et al. Soil bacterial phoD gene abundance and expression in response to applied phosphorus and long-term management[J]. Soil Biology and Biochemistry, 2015, 88: 137-147 DOI:10.1016/j.soilbio.2015.04.014 (  0) 0) |
| [15] |
熊盈盈, 莫祯妮, 邱树毅, 等. 未培养环境微生物培养方法的研究进展[J]. 微生物学通报, 2021, 48(5): 1765-1779 (  0) 0) |
| [16] |
Aagot N, Nybroe O, Nielsen P, et al. An altered Pseudomonas diversity is recovered from soil by using nutrient-poor Pseudomonas-selective soil extract media[J]. Applied and Environmental Microbiology, 2001, 67(11): 5233-5239 (  0) 0) |
| [17] |
Apel K, Hirt H. Reactive oxygen species: Metabolism, oxidative stress, and signal transduction[J]. Annual Review of Plant Biology, 2004, 55: 373-399 (  0) 0) |
| [18] |
Bruns A, Nübel U, Cypionka H, et al. Effect of signal compounds and incubation conditions on the culturability of freshwater bacterioplankton[J]. Applied and Environ mental Microbiology, 2003, 69(4): 1980-1989 (  0) 0) |
| [19] |
岳秀娟, 余利岩, 张月琴. 自然界中处于VBNC状态微生物的研究进展[J]. 微生物学通报, 2004, 31(2): 108-111 (  0) 0) |
| [20] |
Tamaki H, Sekiguchi Y, Hanada S, et al. Comparative analysis of bacterial diversity in freshwater sediment of a shallow eutrophic lake by molecular and improved cultivation-based techniques[J]. Applied and Environmental Microbiology, 2005, 71(4): 2162-2169 (  0) 0) |
| [21] |
Tanaka T, Kawasaki K, Daimon S, et al. A hidden pitfall in the preparation of agar media undermines microorganism cultivability[J]. Applied and Environmental Microbiology, 2014, 80(24): 7659-7666 (  0) 0) |
| [22] |
陈哲, 吴敏娜, 秦红灵, 等. 土壤微生物溶磷分子机理研究进展[J]. 土壤学报, 2009, 46(5): 925-931 (  0) 0) |
| [23] |
姜焕焕, 李嘉钦, 陈刚, 等. 解磷微生物及其在盐碱土中的应用研究进展[J]. 土壤, 2021, 53(6): 1125-1131 DOI:10.13758/j.cnki.tr.2021.06.004 (  0) 0) |
| [24] |
Qian Y C, Shi J Y, Chen Y X, et al. Characterization of phosphate solubilizing bacteria in sediments from a shallow eutrophic lake and a wetland: Isolation, molecular identification and phosphorus release ability determination[J]. Molecules, 2010, 15(11): 8518-8533 (  0) 0) |
| [25] |
Wan W J, Qin Y, Wu H Q, et al. Isolation and characterization of phosphorus solubilizing bacteria with multiple phosphorus sources utilizing capability and their potential for lead immobilization in soil[J]. Frontiers in Microbiology, 2020, 11: 752 (  0) 0) |
| [26] |
Yu X, Liu X, Zhu T H, et al. Isolation and characterization of phosphate-solubilizing bacteria from walnut and their effect on growth and phosphorus mobilization[J]. Biology and Fertility of Soils, 2011, 47(4): 437-446 (  0) 0) |
| [27] |
Janssen P J, Van Houdt R, Moors H, et al. The complete genome sequence of Cupriavidus metallidurans strain CH34, a master survivalist in harsh and anthropogenic environments[J]. PLoS One, 2010, 5(5): e10433 (  0) 0) |
| [28] |
Zheng M M, Wang C, Li W X, et al. Soil nutrients drive function and composition of phoC-harboring bacterial community in acidic soils of Southern China[J]. Frontiers in Microbiology, 2019, 10: 2654 (  0) 0) |
| [29] |
Minari G D, Saran L M, Lima Constancio M T, et al. Bioremediation potential of new cadmium, chromium, and nickel-resistant bacteria isolated from tropical agricultural soil[J]. Ecotoxicology and Environmental Safety, 2020, 204: 111038 (  0) 0) |
| [30] |
Mukherjee S, Bassler B L. Bacterial quorum sensing in complex and dynamically changing environments[J]. Nature Reviews Microbiology, 2019, 17(6): 371-382 (  0) 0) |
| [31] |
石晶晶, 张林, 江飞焰, 等. AM真菌菌丝际细菌具有固氮解磷双重功能[J]. 土壤学报, 2021, 58(5): 1289-1298 (  0) 0) |
2. Soil and Fertilizer & Resources and Environment Institute, Jiangxi Academy of Agricultural Sciences, Nanchang 330200, China;
3. Key Laboratory of Soil and Sustainable Agriculture, Chinese Academy of Sciences, Nanjing 211135, China;
4. Northeast Institute of Geography and Agroecology, Chinese Academy of Sciences, Changchun 130102, China
 2024, Vol. 56
2024, Vol. 56

