2. 中国科学院大学, 北京 100049
土壤微生物是土壤的重要组成部分,对养分循环、有机质分解、污染物降解起着重要作用[1]。人类活动正不断地塑造着土地利用方式,影响着土壤环境[2-3]和微生物群落特征[4-5]。不同土地利用方式下,土壤pH、养分和重金属含量会发生改变,并对微生物生物量、碳氮循环以及功能多样性产生影响[6-7]。研究不同土地利用方式对微生物群落的影响,对合理利用土地、充分发挥土地的生态效益具有指导意义。
不同土地利用方式会改变植被类型、植物覆盖程度、土壤理化性质,进而影响细菌的群落结构。Nacke等[8]发现,林地土壤中木质素含量丰富,参与木质素降解的细菌群落相对活跃,而草地能够接受更高的太阳辐射,使得与光合作用相关的细菌群落相对活跃。将耕地转变为林地会导致根瘤菌增加,拟杆菌和硝化螺旋菌减少[9]。当湿地转化为农田时,土壤细菌的多样性随之下降,拟杆菌门更偏好稻田生态系统,而酸杆菌门则偏好林地生态系统[10]。土地利用方式是微生物群落组成和生物量的关键驱动因素,农业管理(耕作、灌溉等)对土壤微生物群落的影响比海拔、降水的作用更为显著[11]。农业管理是重要的人为活动之一,施肥、耕作、灌溉等人为干扰会影响土壤的细菌群落结构[11],将农用地与自然土地进行比较有利于衡量农业活动对细菌群落的影响。
土地利用方式不仅会影响细菌群落结构,还会影响其构建过程及共现网络特征[7]。构建过程可分为确定性过程和随机过程,其中确定性过程包括同质选择和异质选择,随机过程包括生态漂变、同质扩散和扩散限制[12]。共现网络表征微生物间的相关性,揭示微生物间可能的交互作用或环境偏好[13]。对旱作、水旱轮作和长期淹水的土壤细菌群落进行分析发现,随着淹水程度的增加,细菌群落更易受随机过程的影响,且细菌共现网络的复杂度也逐渐增加[14-15]。Yu等[16]对嫩滩地、农田和休耕地土壤细菌群落构建和共现网络分析发现,农田细菌群落的确定性过程高于嫩滩地和休耕地,而嫩滩地具有更加稳定的共现网络。Xu等[17]对内蒙平原草地和农田的研究发现,草地的随机过程高于农田。但对华北地区农田–草地生态交错带的土壤细菌群落研究发现,农田和草地细菌群落构建十分相似,随土地利用持续时间的增加,确定性过程的影响逐渐增加,共现网络也变得更加复杂[18]。可见,研究土地利用方式对群落构建和共现网络的影响有利于深入理解群落结构变化的生态机制。
稻田和林地是两种重要的土地利用形式,两者在空间分布上往往相互嵌套,在利用形式上也相互转换。长期稻田和林地利用方式下,细菌群落结构和构建过程有怎样的差异,目前还不清楚。由此,根据全国第二次土壤普查结果,本研究选取我国东部和西南部共14个典型水稻土样点,以稻田附近的林地作为对照,对稻田和林地土壤进行采样和16s rDNA测序,获取土壤细菌群落信息,以探究稻田和林地利用方式下土壤细菌群落结构和潜在生态功能的差异,以及群落构建和共现网络的不同,为土地资源的合理管理和利用、实现农业生态系统的可持续发展提供科学依据。
1 材料与方法 1.1 样品采集和气候数据获取根据全国第二次土壤普查数据,于2015年10月水稻收割后从我国东部(江苏、安徽、上海)和西南部(贵州、云南)14个典型水稻土样点和邻近的林地采集土壤0 ~ 20 cm表层样品(表 1)。每个样点按“S”形采集6个样并混匀。样品采集后放入冰盒尽快运回实验室,剔除根系和小石块等杂质,分为两部分:一部分保存在–80 ℃用于土壤总DNA提取;另一部分风干、研磨后用于土壤基本理化性质测定。每个采样点的年均温(AMT)和年均降水量(AMP)从Worldclim网站(https://worldclim.org/)获取,选用的数据为1970—2000年的平均值,见表 1。
|
|
表 1 采样点信息 Table 1 Information of sampling sites |
依据鲁如坤[19]主编的《土壤农业化学分析方法》进行土壤基本理化性质的测定。测定的土壤理化指标包括土壤pH、有机质、全磷、全钾、有效磷、速效钾。土壤pH采用土水质量比1∶2.5电位法测定;有机质采用重铬酸钾氧化–外加热法测定;全磷采用硫酸–高氯酸消煮,钼蓝比色法测定;全钾采用氢氟酸-高氯酸消煮,火焰光度计法测定;有效磷采用碳酸氢钠–乙酸铵浸提,钼蓝比色法测定;速效钾采用乙酸铵浸提,火焰光度计法测定。
1.3 土壤总DNA提取、PCR扩增及Illumina Miseq测序称取0.5 g新鲜土壤样品,采用FastDNA Spin Kit for Soil提取试剂盒(MP Biomedicals,Santa Ana,CA,USA)提取土壤微生物总DNA,利用NanoDrop ND-2000(Thermo Fisher,USA)分析提取DNA的浓度和质量,并通过1% 的琼脂糖凝胶电泳检测其完整性。利用细菌16S rRNA基因的通用引物515F (GTG CCAGCMGCCGCGG) 与907R (CCGTCAATTCM TTTRAGTTT) 扩增其V4 ~ V5区基因片段,正向引物515F的3′端加入已知碱基序列的分类标签(Barcode)以区分不同样品。PCR反应体系为20 uL,其中含Premix 10 μL、上游引物0.4 μL、下游引物0.4 μL、DNA模板1 μL,加无菌水补足20 μL。PCR扩增的反应条件为:95℃ 5 min,30 × (95℃ 30 s,57℃ 30 s,72℃ 45 s),72℃ 10 min。PCR产物用1% 的琼脂糖凝胶电泳检进行纯化,使用AxyPrep DNA Gel Extraction Kit回收试剂盒回收扩增的目的片段。将纯化后的PCR产物等摩尔混匀,使用Illumina Miseq进行测序分析,该测序平台由南京土壤研究所分析测试中心提供。
1.4 生物信息分析16S rRNA测序基因序列使用QIIME 1.9.0 (Quantitative Insights Into Microbial Ecology,http://qiime.sourceforge.net/)进行分析,完成序列质量控制、长度修剪、去除嵌合体、操作分类单元(OTU)聚类、物种信息注释、多样性计算和系统发育树构建[20]。去除质量低于20,长度小于200 bp的序列,使用UCLUST算法按照97% 相似性将序列划分为不同的OTU[21]。物种注释选择Green genes database(http://greengenes.lbl.gov/)数据库。为了进行样品间多样性比较,避免因样本数据测序深度不同造成偏差,序列统一抽平到5 000条进行物种多样性分析。利用FAPROTAX软件对潜在生态功能进行预测[22]。
1.5 数据分析利用STAMP软件对物种组成和潜在功能进行差异分析[23]。利用R软件中“vegan”包进行数量生态学分析[24]。基于Bray-Curtis距离的非度量多维尺度(NMDS)分析对细菌群落β多样性进行降维,并利用置换多元方差分析(ADONIS)检验组间差异。在将林地细菌群落与稻田进行比较时,为排除树木种类不同对微生物的影响,本研究还根据树种类型将其分为针叶林组(松树和香柏)和阔叶林组(白杨和香樟),并将不同树种林地与对应的稻田进行比较。利用基于Bray-Curtis距离的冗余分析(db-RDA)并结合向前筛选变量的方法确定环境变量和细菌群落组成之间的相关性。利用t检验法对稻田和林地细菌群落属性进行显著性分析。根据Stegen等[25]的方法计算βNTI值(β nearest taxon index)和RCbray距离(Raup crick index based on Bray-Curtis distance)。βNTI < –2或βNTI > 2表示确定性过程,其中βNTI < –2表示同质选择,βNTI > 2表示异质选择;–2 < βNTI < 2表示随机过程,其中RCbray < 0.95表示均质扩散,–0.95 < RCbray < 0.95表示生态漂变,RCbray > 0.95表示扩散限制[12]。在构建共现网络时,仅保留所在样品数超过一半的OTU,并计算OTU间的Spearman相关性,仅保留相关系数大于0.6且显著性小于0.01的相关系数,再利用“igraph”包构建网络[26],并用Gephi 0.9.2软件进行网络的可视化[27]。利用Iyer等[28]的方法对共现网络的稳定性进行分析。为进一步解析环境异质选择对共现网络连接数的影响,随机从14个样本中选择10个样本构建共现网络,并计算βNTI矩阵的平均值。分析不同异质选择强度下,共现网络连接数的变化情况,并进行相关性分析。
2 结果与分析 2.1 稻田和林地土壤性质对稻田与周边林地土壤的理化性质进行显著性检验发现(图 1),稻田有效磷含量(9.62 ~ 57.38 mg/kg)显著高于林地(3.7 ~ 41.4 mg/kg)(P < 0.01),而全钾含量(9.98 ~ 21.46 g/kg)则显著低于林地(11.88 ~ 30.30 g/kg)(P < 0.01)。在土壤有机质、pH、全磷和有效钾方面,稻田与林地差异不显著(P > 0.05,图 1)。其中,稻田有机质(SOM)含量为13.29 ~ 94.14 g/kg,林地为13.3 ~ 77.20 g/kg;稻田的pH变化范围为5.06 ~ 8.0,林地为4.76 ~ 8.32;稻田全磷(TP)含量的变化范围为0.27 ~ 1.05 g/kg,林地为0.31 ~ 1.36 g/kg;稻田速效钾(AK)含量的变化范围为66.5 ~ 191.18 mg/kg,林地为88.8 ~ 451.0 mg/kg (表 2)。
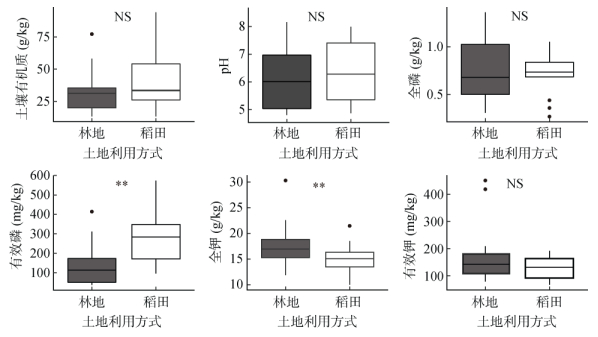
|
(图中NS表示稻田和林地间差异不显著(P > 0.05);*、**、***分别表示稻田和林地间差异在P < 0.05、P < 0.01、P < 0.001水平显著;下同) 图 1 稻田和林地土壤性质比较 Fig. 1 Comparisons of soil properties between paddy fields and woodlands |
|
|
表 2 土壤样品的化学性质 Table 2 Chemical properties of soil samples |
稻田土壤中,变形菌门(相对丰度35.44%)是最丰富的细菌门类,其中α变形菌纲相对丰度8.36%,β变形菌纲10.75%,γ变形菌纲2.89%,δ变形菌纲12.42%;其次是绿弯菌门(19.60%)、酸杆菌门(18.61%)、放线菌门(6.06%)、硝化螺旋菌门(3.67%)、厚壁菌门(3.08%)、浮霉菌门(2.59%)和拟杆菌门(2.12%),这些门类的序列占比所有细菌序列的97%(图 2A)。林地土壤中,变形菌门(相对丰度33.83%)是最丰富的细菌门类,其中α变形菌纲相对丰度14.89%,β变形菌纲7.51%,γ变形菌纲7.24%,δ变形菌纲4.21%;其次是酸杆菌门(31.15%)、放线菌门(8.33%)、绿弯菌门(8.07%)、浮霉菌门(4.51%)、拟杆菌门(2.85%)、厚壁菌门(2.79%)和硝化螺旋菌门(2.14%),这些门类的序列占比所有细菌序列的94%(图 2B)。另外,稻田土壤中绿弯菌门、硝化螺旋菌门和δ变形菌纲的相对丰度显著高于林地(P < 0.001),而酸杆菌门、浮霉菌门、放线菌门和α变形菌纲的相对丰度则显著低于林地(P < 0.001)(图 2C)。
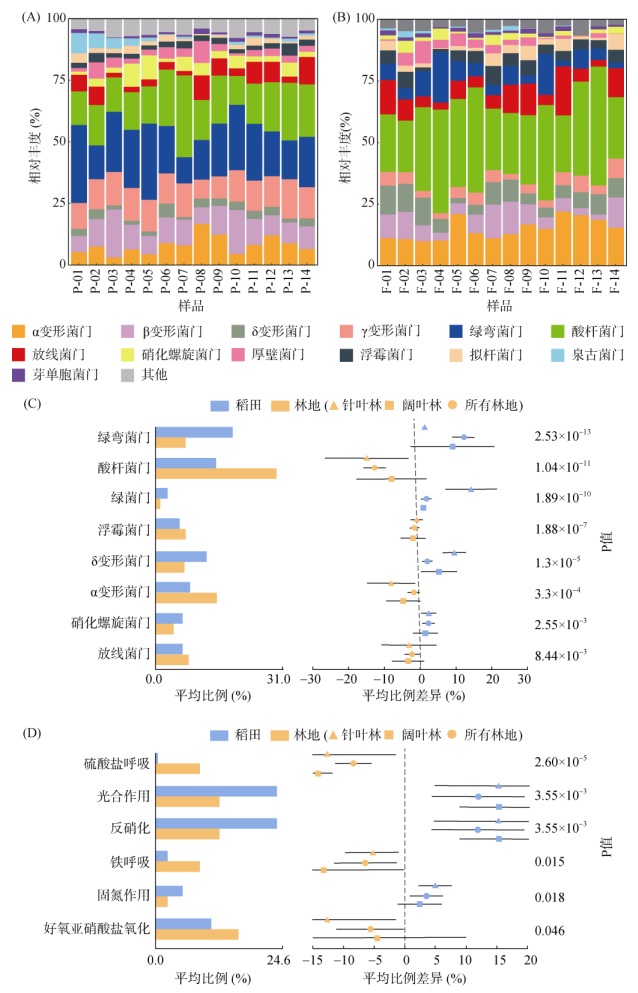
|
图 2 稻田(A)和林地(B)细菌门水平物种组成,及稻田和林地细菌门丰度组间差异(C)和潜在生态功能的组间差异(D) Fig. 2 Relative abundances of bacteria community compositions at phylum level in paddy (A) and woodland soils (B), and statistical comparison between bacteria phylum abundance (C) and potential ecological functions (D) in paddy and woodland soils |
两种土地利用方式下土壤细菌潜在功能也存在显著差异,林地富集了更多铁呼吸、硫酸盐呼吸和硝化作用的功能基因,而稻田富集了更多光合作用、反硝化作用以及生物固氮作用的功能基因(图 2D)。不同的树种类型(针叶林和阔叶林)下,林地与稻田的微生物群落组成和功能差异具有相似的变化特征,但针叶林与稻田差异更为显著(P < 0.001)(图 2C、2D)。
2.3 稻田和林地土壤细菌群落α多样性在土壤细菌群落α多样性上,稻田的物种丰富度(Observed OTUs)显著高于林地(P < 0.01),而Pielou’s均匀度指数和Shannon多样性指数与林地差异不显著(P > 0.05)(图 3A)。pH对林地和稻田土壤细菌群落α多样性影响显著(P < 0.01),无论是稻田还是林地,物种丰富度、Pielou’s均匀度和Shannon多样性指数均与pH呈显著的抛物线关系(P < 0.01)(图 3B)。进一步分析发现,当林地土壤pH < 6时,相应稻田土壤pH显著高于林地(P < 0.01),此时稻田细菌的物种丰富度也显著高于林地;但当林地土壤pH > 6时,稻田和林地的pH差异不显著,物种丰富度的差异也不显著(P > 0.05)(图 3C)。
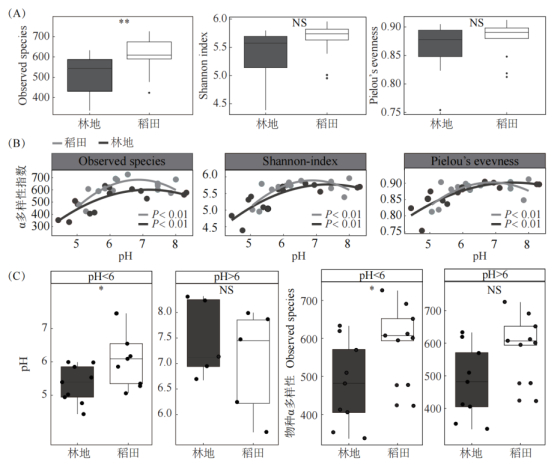
|
图 3 稻田和林地土壤细菌α多样性(A)及其与pH的关系(B),及不同林地pH划分下相应稻田和林地pH和细菌丰富度比较(C) Fig. 3 α-diversityof soil bacterial in paddy field and woodland (A) and its relationship with soil pH, and comparation of soil pH and bacterial richness between paired paddy and woodland soils under different pH of woodland soils(C) |
基于Bray-Curtis距离的NMDS分析并结合ADONIS检验发现(图 4A),稻田和林地细菌群落差异显著(P=0.002),阔叶林和针叶林与稻田细菌群落均有显著差异。阔叶林与针叶林细菌群落差异不显著,但本研究发现阔叶林与针叶林在NMDS2轴上有显著差异。与稻田相比,林地细菌群落的变异更大(图 4B)。采用冗余分析对细菌群落结构与环境因子进行相关性分析发现,pH对稻田和林地细菌群落结构均影响显著。此外,稻田细菌群落还与年均降水量、土壤有机质显著相关,而林地细菌群落则与海拔显著相关(图 4C、4D)。
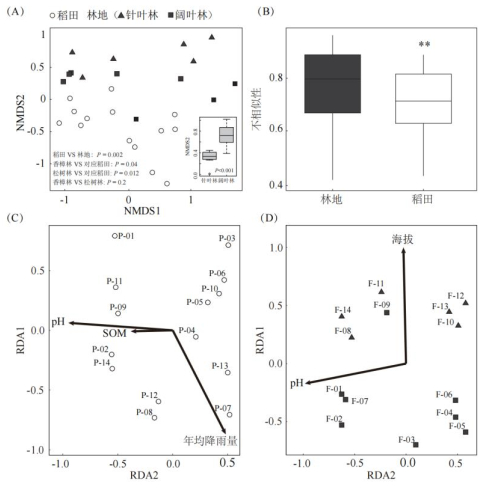
|
图 4 稻田和林地细菌群落基于Bray-Curtis距离的NMDS分析(A)、β多样性比较(B)及与环境变量的RDA分析(C、D) Fig. 4 NMDS analysis of bacterial communities based on Bray-Curtis distance in paddy and woodland soils (A), comparation of β-diversity (B) and RDA analysis with environmental factors(C, D) |
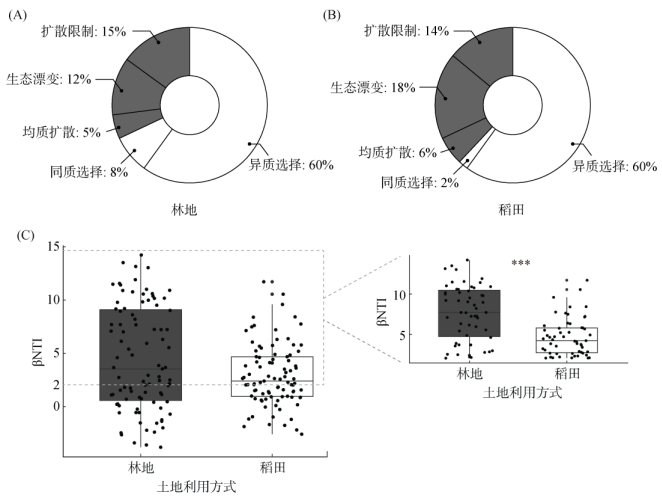
稻田和林地土壤细菌群落均主要受确定性过程中异质选择的影响(图 5A、5B)。在稻田中,确定性过程贡献62%,其中异质选择占60%,同质选择占2%;随机过程贡献38%,其中扩散限制占14%,生态漂变占18%,均质扩散占6%(图 5A)。在林地中,确定性过程贡献68%,其中异质选择占60%,同质选择占8%;随机过程贡献32%,其中扩散限制占15%,生态漂变占12%,均质扩散占5%(图 5B)。尽管稻田和林地异质选择所占比例相同,但林地异质选择的强度显著高于稻田(P < 0.001)(图 5C)。
|
图 5 稻田(A)和林地(B)土壤细菌群落构建及βNTI值比较(C) Fig. 5 Portions of different soil bacteria assembly processes in paddy fields (A) and woodlands (B) and comparisons of their βNTI values(C) |
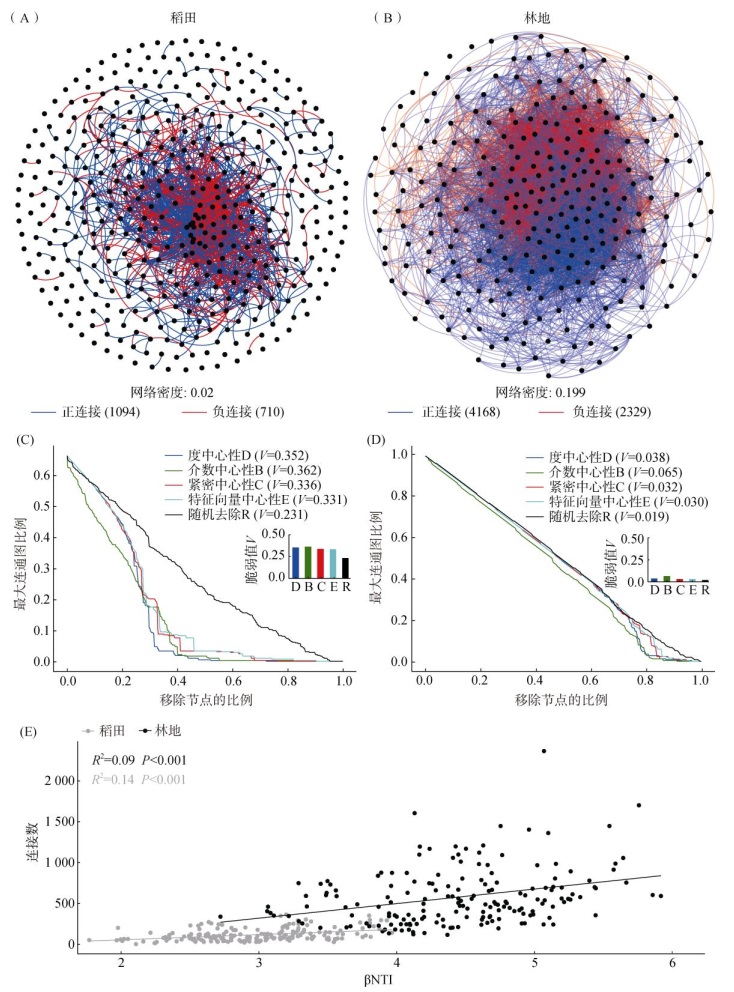
稻田土壤细菌共现网络有连接1 804条,其中正连接1 094条,占60.6%,负连接710条,占39.4%,网络密度为0.02(图 6A)。林地土壤细菌共现网络有连接6 497条,其中正连接4 168条,占64%,负连接2 329条,占36%,网络密度0.199 (图 6B)。林地细菌共现网络稳定性高于稻田(图 6C、6D)。为分析环境异质选择对共现网络的影响,本研究随机从14个样点中选择10个样点计算βNTI值,并构建共现网络,结果发现,共现网络的连接数会随群落βNTI值的增加而增加,在βNTI值相同的情况下林地共现网络的连接数高于稻田(图 6E)。
|
图 6 稻田和林地土壤细菌共现网络(A、B)及其稳定性分析(C、D),及共现网络连接数与βNTI值的关系(E) Fig. 6 Bacteria co-occurrence networks(A, B) and their stabilities(C, D) in paddy soils and woodland soils, and relationship between edge numbers in co-occurrence network and βNTI values(E) |
本研究结果表明,不同土地利用方式会引起细菌多样性的变化,稻田的细菌丰富度显著高于林地(图 3)。刘文静等[14]研究发现,与旱地相比,稻田水旱轮作的耕作管理方式增加了细菌的α多样性。一方面水稻种植期间有干湿交替阶段,能产生更多的生态位,容纳更多的微生物共存;另一方面,稻田淹水使得土壤pH趋于中性,有利于微生物共存。pH与细菌α多样性呈显著的抛物线关系,这与前人到的研究结果一致[29]。由于pH对细菌α多样性的重要影响,本研究进一步分析发现,当林地pH < 6时,稻田的pH和细菌丰富度显著高于林地(图 3C);当林地pH > 6时,稻田和林地的pH和α多样性差异均不显著。这说明本研究中稻田细菌丰富度高于林地主要是由于稻田淹水使土壤pH趋于中性,而非干湿交替造成的环境异质性。
土地利用方式对细菌群落组成和潜在功能产生了显著影响(图 2)。与周围林地相比,稻田绿弯菌门、绿菌门和硝化螺旋菌门的相对丰度显著增加,而酸杆菌门、浮霉菌门和放线菌门的相对丰度则显著减少(图 2C)。酸杆菌门[30]、浮霉菌门[31]和放线菌门[32]在降解林地枯枝落叶上发挥了重要的作用,林地生态系统中枯枝落叶等凋落物要多于稻田,更有利于酸杆菌门、浮霉菌门和放线菌门的生长。绿弯菌和绿菌可以进行光合作用固碳[33],硝化螺旋菌可以进行硝化和反硝化作用[34]。稻田的淹水管理和氮肥施用有利于绿弯菌门、绿菌门和硝化螺旋菌门的生长,因此稻田也富集了更多的光合作用和反硝化作用功能基因(图 2D)。此外,稻田的厌氧环境还有利于生物固氮[35],从而富集了更多的固氮功能基因(图 2D)。固氮可以将空气中的氮气转化为微生物可能利用的氮,而反硝化作用则促进了氮的损失[35]。与林地相比,稻田可能会有更快的氮循环速率。
不同土地利用方式不仅显著改变了细菌群落结构(图 4A),也影响了细菌群落对环境因子的响应(图 4C、4D)。pH和有机质对细菌群落结构的重要作用已被广泛报道[29, 36-37],本研究中,pH对稻田和林地细菌均具有显著影响,这与前人的研究结果一致。本研究中稻田有机质含量较林地具有更高的变异程度(图 1),因而有机质对对稻田细菌的影响显著,而对林地细菌的影响不显著。本研究中,稻田和林地有着相同的海拔和和年均降水量,但稻田细菌群落对年均降水量变化更为敏感,而林地细菌群落则对海拔变化更为敏感。降水量增加会加剧稻田盐基离子的淋湿,影响土壤pH和养分状况[38-39],进而影响细菌群落结构;而林地的树木冠层及枯枝落叶层可以减缓降雨对土壤的淋溶作用,且本研究也观察到林地全钾含量显著高于稻田,因此降雨对林地细菌影响不显著。林地细菌群落对海拔变化更加敏感,海拔会影响土壤理化性质、温度及植被类型,进而影响细菌群落结构[40]。本研究中,海拔可能会通过影响树种进而影响细菌群落,在低海拔地区,林地以阔叶林(香樟和白杨树)为主;而在高海拔地区,则以针叶林(松树和香柏树)为主(表 1)。不同树种产生的根际分泌物和凋落物具有不同的化学属性,塑造了不同的细菌群落结构[40]。
不同土地利用方式下细菌群落构建也存在差异(图 5),主要体现在同质选择和生态漂变方面。稻田的生态漂变(18%)高于林地(12%),而同质选择(2%)则弱于林地(8%)(图 5)。尽管稻田和林地的异质选择(βNTI > 2)均为60%,但林地异质选择的强度显著高于稻田(图 5C),林地具有更强的群落变异(图 4B)。稻田和林地群落构建的差异可能与管理方式有关,土壤水分和肥力状况会影响细菌群落构建。淹水增加了土壤细菌群落的随机过程[14]。确定性过程发生在低土壤营养条件下,而随机性过程发生在较高的土壤营养条件下[41]。稻田的肥料施用和淹水环境为细菌群落提供了良好的环境,减弱了环境选择的压力,有利于生态漂变的发生,从而维持更高的物种丰富度(图 3A)。
不同土地利用方式会影响细菌共现网络特征,林地细菌共现网络的连接数是稻田的3.6倍(图 6)。这与前人的研究结果相似,与人为干扰强烈的葡萄园相比,林地细菌的共线网络具有更多的连接数[7]。共现网络由环境异质选择和微生物的交互作用共同塑造[13],共现网络的正(负)连接表示两种微生物在样点的分布上具有强的正(负)相关性,这可能是由于他们有着相同(或相反)的环境偏好,或者彼此之间存在互利(互斥)的交互作用,而随机过程则不会导致两种微生物的强相关关系[42]。由于本研究采样尺度较大,土壤性质、气候特征均具有较大变异,环境异质选择会导致相同或相反环境偏好的微生物显著相关,影响共现网络对微生物交互作用的推断。本研究在林地共现网络中观察到了更多的连接数和更高的网络密度(图 6A、6B),可能是因为林地更高的异质选择强度所致(图 5C)。
由于环境异质选择对共现网络的作用,有学者指出共现网络并不能反映微生物间的交互作用[43]。为解析环境选择对共现网络的影响,Goberna等[44]将网络中不同微生物在各个样点中的状态(正连接:有–有vs无–无;负连接:有–无vs无–有)作为自变量,样点的理化性质(降维为主成分1和主成分2)作为因变量,进行多元方差分析,不同状态(正连接下:有–有vs无–无;负连接下:有–无vs无–有)下,若样点理化性质差异显著,则归类为环境选择,不显著则归类为潜在微生物交互,结果发现,在共现网络的连接中,约一半连接来源于环境选择,且环境选择导致的正相关微生物间会有更小的遗传发育距离。Stegen等[25]研究表明细菌遗传距离越相近,其环境偏好也越相似,因此,βNTI值越大意味着环境异质选择的强度也就越大,共现网络的连接数也会越多。因此在同等βNTI条件下比较共现网络的拓扑特征可以减少环境选择对共现网络的影响。考虑到微生物受多种理化性质的影响,Goberna等[44]的方法需要测定较多的理化性质。因此,本研究选择在同等βNTI条件下比较林地和稻田共现网络的拓扑特征,以推断潜在微生物交互作用的强弱。本研究利用随机抽样的方法来产生一定的βNTI变异,从而研究不同βNTI条件下共现网络的连接边数,发现共现网络连接数会随环境异质选择强度的增加而增加,这与前人的结论一致[13]。当稻田和林地具有相同的异质选择强度时,林地共现网络的连接数高于稻田,这意味着林地细菌会具有更加强烈的细菌交互作用,这可能与林地土壤更低的养分状况有关。低养分环境会塑造更加复杂的细菌共现网络,细菌共现网络的连接数会随土壤发育程度的增加逐渐降低[45];而根际细菌共现网络的连接数也低于非根际土[46]。本研究中,林地有效磷的含量显著低于稻田(图 1),一方面,林地细菌需要相互合作来降解有机质以获取必需的矿质养分;另一方面,林地相对贫乏的养分状况加剧了细菌间的竞争,而稻田施肥则可能弱化了细菌间的合作与竞争效应,因此在林地中观察到了更多的正连接和负连接。
4 结论稻田淹水使土壤pH趋于中性,进而提高了细菌丰富度。稻田与林地细菌群落结构和潜在功能具有显著差异,稻田富集了更多生物固氮和反硝化功能基因,可能具有更快的氮周转速率。稻田和林地细菌群落构建均由异质选择主导,但林地异质选择的强度要高于稻田。异质选择强度越高,共现网络的连接数就越多。在相同的异质选择强度下,林地共现网络的连接数要高于稻田,表明林地细菌群落可能存在更加紧密的生物交互作用,以适应养分相对贫乏的环境。
| [1] |
孔亚丽, 秦华, 朱春权, 等. 土壤微生物影响土壤健康的作用机制研究进展[J]. 土壤学报, 2024, 61(2): 331-347 (  0) 0) |
| [2] |
李珊, 杨越超, 姚媛媛, 等. 不同土地利用方式对山东滨海盐碱土理化性质的影响[J]. 土壤学报, 2022, 59(4): 1012-1024 (  0) 0) |
| [3] |
文慧颖, 吴华勇, 董岳, 等. 不同土地利用方式下南亚热带赤红壤酸化特征[J]. 土壤, 2023, 55(2): 372-378 DOI:10.13758/j.cnki.tr.2023.02.018 (  0) 0) |
| [4] |
胡芳, 王芳, 韩晓增, 等. 不同土地利用方式下典型黑土区土壤微生物群落演替规律[J]. 土壤学报, 2022, 59(5): 1238-1247 (  0) 0) |
| [5] |
薛壮壮, 冯童禹, 王超, 等. 土地利用方式对酸性红壤丛枝菌根真菌群落的影响[J]. 土壤, 2022, 54(4): 733-739 DOI:10.13758/j.cnki.tr.2022.04.010 (  0) 0) |
| [6] |
Zhao D, Li F, Yang Q, et al. The influence of different types of urban land use on soil microbial biomass and functional diversity in Beijing, China[J]. Soil Use and Management, 2013, 29(2): 230-239 DOI:10.1111/sum.12034 (  0) 0) |
| [7] |
Xue P P, Minasny B, McBratney A B. Land-use affects soil microbial co-occurrence networks and their putative functions[J]. Applied Soil Ecology, 2022, 169: 104184 DOI:10.1016/j.apsoil.2021.104184 (  0) 0) |
| [8] |
Nacke H, Fischer C, Thürmer A, et al. Land use type significantly affects microbial gene transcription in soil[J]. Microbial Ecology, 2014, 67(4): 919-930 DOI:10.1007/s00248-014-0377-6 (  0) 0) |
| [9] |
Szoboszlay M, Dohrmann A B, Poeplau C, et al. Impact of land-use change and soil organic carbon quality on microbial diversity in soils across Europe[J]. FEMS Microbiology Ecology, 2017, 93(12): fix146 (  0) 0) |
| [10] |
张拓, 徐飞, 怀宝东, 等. 松花江下游沿江湿地土地利用变化对土壤细菌群落多样性的影响[J]. 环境科学, 2020, 41(9): 4273-4283 (  0) 0) |
| [11] |
Drenovsky R E, Steenwerth K L, Jackson L E, et al. Land use and climatic factors structure regional patterns in soil microbial communities[J]. Global Ecology and Biogeography, 2010, 19(1): 27-39 DOI:10.1111/j.1466-8238.2009.00486.x (  0) 0) |
| [12] |
Ning D L, Yuan M T, Wu L W, et al. A quantitative framework reveals ecological drivers of grassland microbial community assembly in response to warming[J]. Nature Communications, 2020, 11(1): 4717 DOI:10.1038/s41467-020-18560-z (  0) 0) |
| [13] |
Barberán A, Bates S T, Casamayor E O, et al. Using network analysis to explore co-occurrence patterns in soil microbial communities[J]. The ISME Journal, 2012, 6(2): 343-351 DOI:10.1038/ismej.2011.119 (  0) 0) |
| [14] |
刘文静, 张建伟, 邱崇文, 等. 水旱轮作对土壤微生物群落构建过程的影响机制[J]. 土壤, 2020, 52(4): 710-717 DOI:10.13758/j.cnki.tr.2020.04.009 (  0) 0) |
| [15] |
Liu W J, Graham E B, Dong Y, et al. Balanced stochastic versus deterministic assembly processes benefit diverse yet uneven ecosystem functions in representative agroecosystems[J]. Environmental Microbiology, 2021, 23(1): 391-404 DOI:10.1111/1462-2920.15326 (  0) 0) |
| [16] |
Yu Y Y, Shi Y, Li M M, et al. Land-use type strongly affects soil microbial community assembly process and inter-Kingdom co-occurrence pattern in a floodplain ecosystem[J]. Applied Soil Ecology, 2022, 179: 104574 DOI:10.1016/j.apsoil.2022.104574 (  0) 0) |
| [17] |
Xu A A, Liu J, Guo Z Y, et al. Distinct assembly processes and determinants of soil microbial communities between farmland and grassland in arid and semiarid areas[J]. Applied and Environmental Microbiology, 2021, 87(22): e0101021 DOI:10.1128/AEM.01010-21 (  0) 0) |
| [18] |
Xu A A, Guo Z Y, Pan K, et al. Increasing land-use durations enhance soil microbial deterministic processes and network complexity and stability in an ecotone[J]. Applied Soil Ecology, 2023, 181: 104630 DOI:10.1016/j.apsoil.2022.104630 (  0) 0) |
| [19] |
鲁如坤. 土壤农业化学分析方法[M].
中国农业科学技术出版社, 北京, 2000
(  0) 0) |
| [20] |
Caporaso J G, Kuczynski J, Stombaugh J, et al. QIIME allows analysis of high-throughput community sequencing data[J]. Nature Methods, 2010, 7(5): 335-336 DOI:10.1038/nmeth.f.303 (  0) 0) |
| [21] |
Edgar R C. Search and clustering orders of magnitude faster than BLAST[J]. Bioinformatics, 2010, 26(19): 2460-2461 DOI:10.1093/bioinformatics/btq461 (  0) 0) |
| [22] |
Louca S, Parfrey L W, Doebeli M. Decoupling function and taxonomy in the global ocean microbiome[J]. Science, 2016, 353(6305): 1272-1277 DOI:10.1126/science.aaf4507 (  0) 0) |
| [23] |
Parks D H, Tyson G W, Hugenholtz P, et al. STAMP: Statistical analysis of taxonomic and functional profiles[J]. Bioinformatics, 2014, 30(21): 3123-3124 DOI:10.1093/bioinformatics/btu494 (  0) 0) |
| [24] |
Dixon P. VEGAN, a package of R functions for community ecology[J]. Journal of Vegetation Science, 2003, 14(6): 927 DOI:10.1111/j.1654-1103.2003.tb02228.x (  0) 0) |
| [25] |
Stegen J C, Lin X J, Fredrickson J K, et al. Quantifying community assembly processes and identifying features that impose them[J]. The ISME Journal, 2013, 7(11): 2069-2079 DOI:10.1038/ismej.2013.93 (  0) 0) |
| [26] |
Csardi G, Nepusz T. The igraph software package for complex network research[J]. Complex Systems, 2006, 1695(5): 1-9 (  0) 0) |
| [27] |
Bastian M, Heymann S, Jacomy M. Gephi: An open source software for exploring and manipulating networks[J]. Proceedings of the International AAAI Conference on Web and Social Media, 2009, 3(1): 361-362 DOI:10.1609/icwsm.v3i1.13937 (  0) 0) |
| [28] |
Iyer S, Killingback T, Sundaram B, et al. Attack robustness and centrality of complex networks[J]. PLoS One, 2013, 8(4): e59613 DOI:10.1371/journal.pone.0059613 (  0) 0) |
| [29] |
Fierer N, Jackson R B. The diversity and biogeography of soil bacterial communities[J]. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(3): 626-631 (  0) 0) |
| [30] |
Rawat S R, Männistö M K, Bromberg Y, et al. Comparative genomic and physiological analysis provides insights into the role of Acidobacteria in organic carbon utilization in Arctic tundra soils[J]. FEMS Microbiology Ecology, 2012, 82(2): 341-355 DOI:10.1111/j.1574-6941.2012.01381.x (  0) 0) |
| [31] |
Dedysh S N, Ivanova A A. Planctomycetes in boreal and subarctic wetlands: Diversity patterns and potential ecological functions[J]. FEMS Microbiology Ecology, 2019, 95(2): fiy227 (  0) 0) |
| [32] |
Kirby R. Actinomycetes and lignin degradation[J]. Advances in Applied Microbiology, 2005, 58: 125-168 (  0) 0) |
| [33] |
Gupta R S. Molecular markers for photosynthetic bacteria and insights into the origin and spread of photosynthesis[M]//Advances in Botanical Research. Amsterdam: Elsevier, 2013: 37–66.
(  0) 0) |
| [34] |
Sheng R, Meng D L, Wu M N, et al. Effect of agricultural land use change on community composition of bacteria and ammonia oxidizers[J]. Journal of Soils and Sediments, 2013, 13(7): 1246-1256 DOI:10.1007/s11368-013-0713-3 (  0) 0) |
| [35] |
Jetten M S. The microbial nitrogen cycle[J]. Environmental Microbiology, 2008, 10(11): 2903-2909 DOI:10.1111/j.1462-2920.2008.01786.x (  0) 0) |
| [36] |
Fierer N. Embracing the unknown: Disentangling the complexities of the soil microbiome[J]. Nature Reviews Microbiology, 2017, 15(10): 579-590 DOI:10.1038/nrmicro.2017.87 (  0) 0) |
| [37] |
Rousk J, Bååth E, Brookes P C, et al. Soil bacterial and fungal communities across a pH gradient in an arable soil[J]. The ISME Journal, 2010, 4(10): 1340-1351 DOI:10.1038/ismej.2010.58 (  0) 0) |
| [38] |
Ma M Z, Gao Y, Song X W, et al. Migration and leaching characteristics of base cation: Indicating environmental effects on soil alkalinity in a Karst area[J]. Environmental Science and Pollution Research International, 2018, 25(21): 20899-20910 DOI:10.1007/s11356-018-2266-x (  0) 0) |
| [39] |
Wang Y Y, Li H Z, Xu Z X. Rainfall-induced nutrient losses from manure-fertilized farmland in an alluvial plain[J]. Environmental Monitoring and Assessment, 2016, 188(1): 8 DOI:10.1007/s10661-015-5008-2 (  0) 0) |
| [40] |
Shen C C, Xiong J B, Zhang H Y, et al. Soil pH drives the spatial distribution of bacterial communities along elevation on Changbai Mountain[J]. Soil Biology and Biochemistry, 2013, 57: 204-211 DOI:10.1016/j.soilbio.2012.07.013 (  0) 0) |
| [41] |
Feng Y Z, Guo Z Y, Zhong L H, et al. Balanced fertilization decreases environmental filtering on soil bacterial community assemblage in North China[J]. Frontiers in Microbiology, 2017, 8: 2376 DOI:10.3389/fmicb.2017.02376 (  0) 0) |
| [42] |
Liu C, Li C N, Jiang Y Q, et al. A guide for comparing microbial co-occurrence networks[J]. iMeta, 2023, 2(1): e71 DOI:10.1002/imt2.71 (  0) 0) |
| [43] |
Goberna M, Verdú M. Cautionary notes on the use of co-occurrence networks in soil ecology[J]. Soil Biology and Biochemistry, 2022, 166: 108534 DOI:10.1016/j.soilbio.2021.108534 (  0) 0) |
| [44] |
Goberna M, Montesinos-Navarro A, Valiente-Banuet A, et al. Incorporating phylogenetic metrics to microbial co-occurrence networks based on amplicon sequences to discern community assembly processes[J]. Molecular Ecology Resources, 2019, 19(6): 1552-1564 DOI:10.1111/1755-0998.13079 (  0) 0) |
| [45] |
Dong K, Yu Z, Kerfahi D, et al. Soil microbial co-occurrence networks become less connected with soil development in a high Arctic glacier foreland succession[J]. Science of the Total Environment, 2022, 813: 152565 DOI:10.1016/j.scitotenv.2021.152565 (  0) 0) |
| [46] |
Fan K K, Cardona C, Li Y T, et al. Rhizosphere-associated bacterial network structure and spatial distribution differ significantly from bulk soil in wheat crop fields[J]. Soil Biology and Biochemistry, 2017, 113: 275-284 DOI:10.1016/j.soilbio.2017.06.020 (  0) 0) |
2. University of Chinese Academy of Sciences, Beijing 100049, China
 2024, Vol. 56
2024, Vol. 56


