2. 安徽省农业废弃物肥料化利用与耕地质量提升工程研究中心, 安徽凤阳 233100
土壤是地球生态系统的基础,对维护植物生长和全球生物地球化学循环的平衡具有至关重要的作用[1]。在这一复杂系统中,土壤细菌是不可或缺的一部分,它们对保持土壤肥力和促进作物生产具有深远的影响[2]。虽然传统农业中化肥的大量使用极大地提高了作物产量,但也带来了如土壤结构破坏、生物多样性降低以及潜在的环境风险等副作用[3]。因此,寻找并实施可持续的土壤管理策略变得尤为迫切,其中使用有机肥料配施化肥已成为一项行之有效的重要举措[4]。相对于化肥,有机肥料的优势在于不仅能提供作物生长所需的丰富营养物质,同时也有助于改善土壤结构,提高土壤肥力,从而增强土壤微生物活性及其多样性,恢复或维护土壤生态系统的健康和平衡[5-6]。
土壤细菌亚群的多样性及其组成对土壤质量具有重要影响[7]。研究表明,有机肥中丰富的有机物为土壤细菌提供了优质的能量来源,特别是对在有机物分解和养分循环过程中扮演关键角色的细菌群体[8-9]。然而,不同的土壤细菌亚群可能对有机肥与化肥的响应各不相同,这影响着土壤的生态平衡及作物生长[10]。尽管现有研究已对相关问题进行了解答,但对有机肥部分替代化肥对土壤细菌亚群特性影响的认识仍存在不足。且现阶段多数研究集中于有机肥施用的短期效应,而对长期影响的研究则相对欠缺[11-12]。另外,从生态位角度对土壤细菌亚群变化的深入也探讨较为缺乏,忽视了土壤微生物在多个生态位中竞争与共存的复杂动态关系。此外,环境因素,如土壤pH和土壤养分,对土壤微生物多样性及活性起着关键作用[13-14],长期条件下这些因素如何具体作用于微生物的结构尚需进步研究。总之,研究有机肥对不同土壤细菌亚群特性的影响,对于改进肥料管理和提升农业生产的可持续性具有重大意义。
由此,本研究基于安徽省阜阳市临泉县农业科学研究所进行的长期田间试验,从7种不同施肥管理措施下采集土样并进行测定,同时使用16S rRNA基因扩增子测序技术进行分析,研究了不同施肥管理措施对土壤理化性质变化的影响,以及不同丰度细菌类群的特征及其主要驱动因素,探究了不同丰度细菌类群与施肥管理之间的潜在联系,以从微生物学视角为土壤施肥改良提供建议。
1 材料与方法 1.1 试验地概况本研究依托安徽省阜阳市临泉县农业科学研究所进行的长期田间试验,该试验始于2010年。试验区属于大陆性暖温带半湿润季风气候区,气候温和,雨量适中,日照充足,四季分明,平均最低气温12 ℃,平均最高气温22 ℃,年平均气温15 ℃。土壤类型为砂姜黑土,耕作方式为小麦‒玉米轮作。试验前土壤的基本理化性质为:黏粒质量分数9.97%,粉粒质量分数47.88%,砂粒质量分数42.15%,土壤pH 5.72,有机碳含量7.33 g/kg,碱解氮含量80.1 mg/kg,有效磷含量16 mg/kg,速效钾含量116.70 mg/kg,全磷含量0.36 g/kg。
1.2 供试材料供试作物:小麦品种为丰德存麦12号,播种量为225 kg/hm2;玉米品种为豫中禾968,播种量为75 000粒/hm2。
供试肥料:尿素(N 460 g/kg,安徽吴源化工集团有限公司)、过磷酸钙(P2O5 160 g/kg,云南省个旧市丰收磷化工有限公司)、硫酸钾(K2O 500 g/kg,山东青上化工有限公司)购置于市场;猪粪有机肥(N 16.4 g/kg、P2O5 36.0 g/kg、K2O 11.0 g/kg、有机质≥450 g/kg)由阜阳义丰肥业有限公司提供,肥料登记证号为皖农肥(2017)准字5496;秸秆腐熟剂由南京农业大学提供。
1.3 试验设计试验采用随机区组设计,共设置7个处理,每个处理4次重复。7个处理分别为空白对照(CK)、化肥(T1)、化肥+秸秆还田(T2)、70% 化肥+3 000 kg/hm2有机肥(T3)、70% 化肥+3 000 kg/hm2有机肥+秸秆还田(T4)、50% 化肥+6 000 kg/hm2有机肥(T5)、50% 化肥+6 000 kg/hm2有机肥+秸秆还田(T6),其中秸秆还田为全量秸秆还田。各处理施肥量如表 1所示。
|
|
表 1 不同处理的施肥量 Table 1 The application rates of fertilizers under different treatments |
于2023年6月小麦成熟期采用9点取样法在每个小区随机选取9个点,用土钻采集0 ~ 20 cm土样。土样混合均匀后去除石块、作物根茎等杂质,一部分置于–20 ℃冰箱保存用于土壤DNA提取,一部分风干、研磨、过筛后用于测定土壤理化性质。
1.5 土壤DNA提取和16S rRNA基因测序每个样品称取0.3 g,根据E.Z.N.A. soil试剂盒(Omega Bio-tek,Norcross,GA,U.S.)的说明书进行总DNA提取,DNA的浓度和纯度利用NanoDrop 2000进行检测,DNA提取的质量用1% 的琼脂糖凝胶电泳进行验证。用338F (5’-ACTCCTACGGGAGG CAGCAG-3’) 和806R (5’-GGACTACHVGGGTW TCTAAT-3’) 引物对V3 ~ V4可变区进行PCR扩增。PCR扩增体系(20 µL)为:4 µL的5×FastPfu Buffer,2 μL的2.5 mmol/L dNTPs,正向引物(5 μmol/L)0.8 μL,反向引物(5 μmol/L)0.8 μL,0.4 μL的FastPfu Polymerase,0.2 μL的BSA,10 ng的DNA模板,用ddH2O补足至20 μL。PCR反应在ABIGeneAmp 9700 PCR仪上进行,反应条件为:95 ℃预变性3 min;95 ℃变性30 s,55 ℃退火30 s,72℃延伸45 s,27个循环;72 ℃总延伸10 min,最后10℃保存。扩增产物由上海美吉生物医药科技有限公司在Miseq PE300平台上进行高通量测序。
1.6 土壤化学性质测定根据土壤相关指标的测定方法[15],进行土壤pH、有机质(SOM)、全氮(TN)、全磷(TP)、全钾(TK)、铵态氮(NH4+-N)、有效磷(AP)和速效钾(AK)含量的测定。
1.7 高通量数据分析原始测序序列经过QIIME流程(版本1.9.1)进行处理[16]。首先,应用Cutadapt(V1.9.1)软件进行序列的质量控制,剔除长度小于150 bp且平均碱基质量分数低于20的序列,确保序列数据的高质量[17];接着,采用UCHIME算法识别并剔除嵌合序列[18];随后,在QIIME平台中,利用UCLUST工具按照≥97% 的相似度标准对序列进行聚类,形成可操作分类单元(OTU);最终,使用Mothur工具对各OTU进行注释和分析,通过Silva数据库(https://www.arb-silva.de/)检索每个OTU的分类信息,并对各样本中OTU的丰度进行估算[19]。
1.8 统计和生物信息学分析根据以往研究,本研究将细菌群落按相对丰度划分为丰富和稀有细菌亚群。在所有样品中,相对丰度大于等于总序列0.1% 的OTU被定义为“丰富细菌亚群”,小于总序列0.01%的OTU被定义为“稀有细菌亚群”[20-21]。
本研究所有的统计分析,均通过R studio中的不同库/包(运行R版本3.6.1)进行。为评估细菌亚群的α多样性,运用R语言中的“Vegan”包,计算Chao1和Shannon指数确定群落的丰富度和多样性,并采用方差分析及(邓肯多重范围检验)对各组间差异进行显著性检验[22]。此外,基于Bray-Curtis距离,进行主坐标分析(PCoA),以揭示细菌亚群落β多样性的组成差异,并通过“Vegan”包中的“Adonis”功能进行了非参数多元方差分析(PERMANOVA),以进一步评估这些差异的统计学意义[23-24]。群落组成差异是基于Kruskal-Wallis整体检验,并通过Wilcoxon秩和检验对各组之间进行成对比较[25]。群落构建的聚类树依据Bray-Curtis距离矩阵,采用UPGMA层次聚类法完成[26]。物种生态位宽度的计算,本研究采用Wu等[27]的研究方法,用Levins生态位宽度指数表示。Mantel检验用于探讨土壤环境变量与细菌群落组成之间的相关性[28]。通过冗余分析(RDA),研究单个与整体土壤环境变量对群落结构差异的贡献,以评估土壤环境变量对细菌群落组成的影响[29]。
2 结果与分析 2.1 不同施肥处理下土壤化学性质变化如表 2所示,相较对照,不同施肥处理下土壤化学性质发生了显著变化。对于全量养分(SOM、TN、TK、TP),不同施肥处理土壤中其含量整体呈上升的趋势,其中,SOM和TN在T6处理中最高,TP在T4处理中达到最高,TK则在T1处理中上升最为显著。对于速效养分(NH4+-N、AP、AK),不同施肥处理同样呈现整体上升的趋势,且均在T6处理中达到最高。然而,在不同施肥处理土壤pH整体呈下降趋势,尤其在T6处理中下降程度最为显著。值得注意的是,秸秆还田(T2、T4、T6)处理显著提高了土壤中SOM的含量,而化肥减量施用(T3、T4、T5、T6)处理则有效地提高了土壤中速效养分的含量。这表明在土壤管理中,通过秸秆还田和调整化肥施用量,可以调节土壤中的养分水平。
|
|
表 2 不同施肥处理土壤化学性质 Table 2 Soil chemical properties under different fertilization treatments |
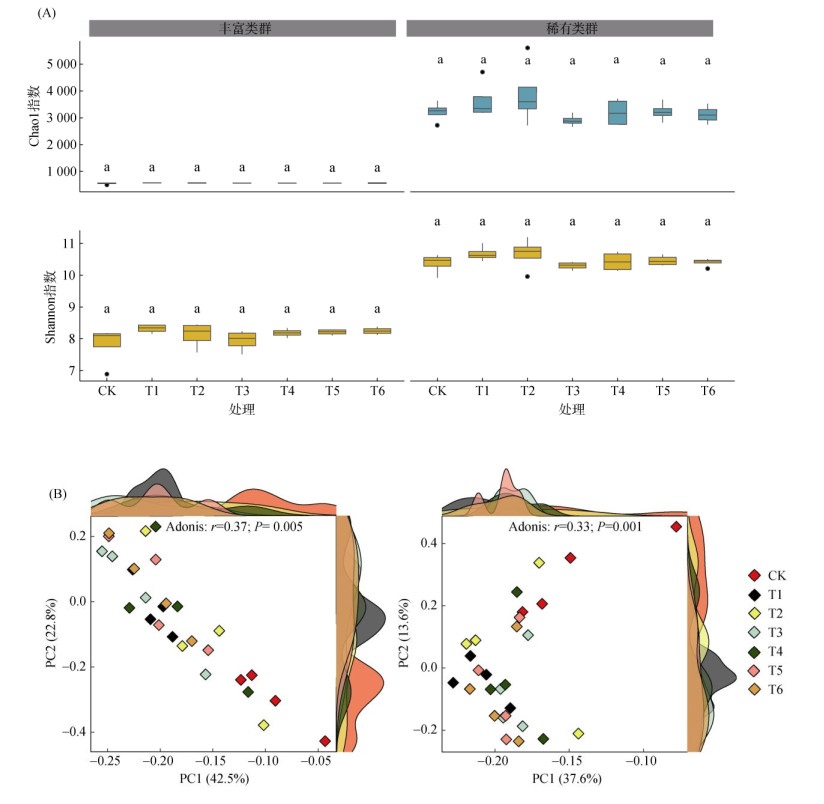
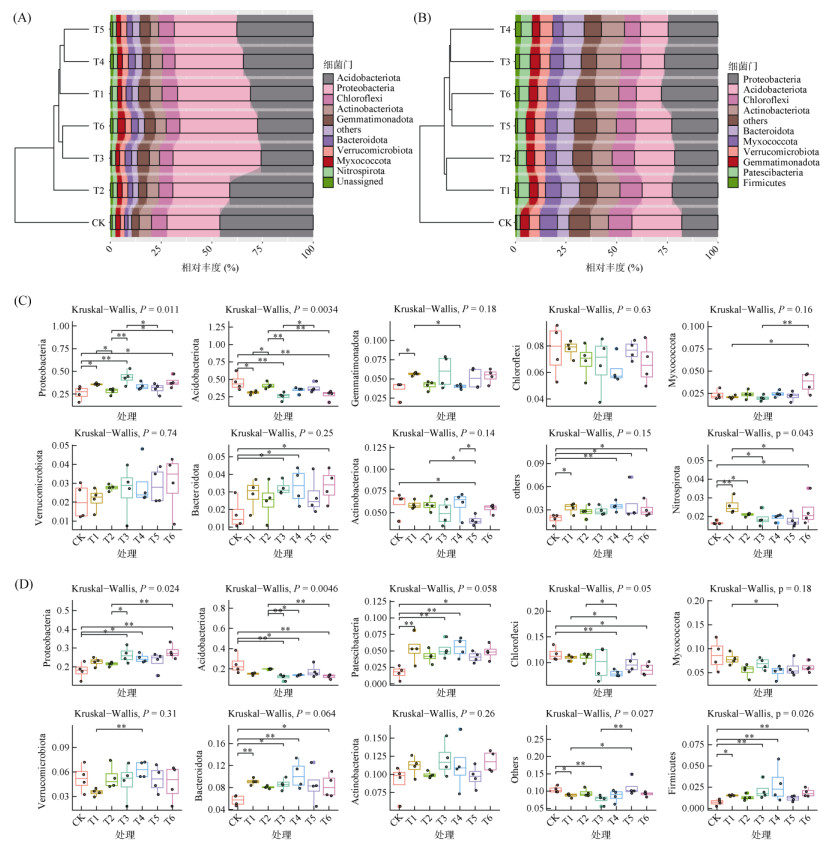
不同处理下土壤细菌亚群的Chao1和Shannon指数均无显著性差异,而丰富和稀有细菌亚群的α多样性存在显著性差异,稀有亚群的Chao1和Shannon指数均高于丰富亚群(图 1A)。基于Bray-Curtis相似性矩阵的PCoA分析显示,不同处理下的土壤细菌亚群组成存在显著差异(丰富类群,r=0.37,P=0.005;稀有类群,r=0.33,P=0.001) (图 1B)。此外,利用UPGMA层级聚类构建聚类树探究细菌群落的组成关系发现,不管是在丰富类群还是在稀有类群中,对照组较处理组均具有较大差异,且施用有机肥后细菌群落组成的变化较未施用处理也具有较大差异(图 2A、2B)。
|
(图A中小写字母表示同一细菌亚群不同处理间差异显著,P < 0.05,n = 4) 图 1 不同施肥处理下土壤丰富和稀有细菌亚群多样性(A)和OTU水平PCoA (B) Fig. 1 Diversity of soil abundant and rare sub-communities under different fertilization treatments (A) and PCoA at the OTU level (B) |
|
(图C、D中*、**分别表示处理间差异在P < 0.05、P < 0.01水平显著,纵坐标表示未百分数的各菌门的相对丰度) 图 2 不同施肥处理下丰富和稀有细菌亚群组成(A、B)和丰度前10位细菌门(C、D)水平差异 Fig. 2 Composition of soil abundant and rare bacterial sub-communities under different fertilization treatments (A, B) and differences at the level of the top 10 phyla (C, D) |
不同施肥处理土壤细菌在丰富和稀有类群中的主要门类和丰度存在差异,但在门水平上主要包括变形菌门(Proteobacteria)、酸杆菌门(Acidobacteriota)和绿弯菌门(Chloroflexi)。基于Kruskal-Wallis检验及Wilcoxon秩和检验结果(图 2C、2D),不同细菌亚群的主要门类在不同处理中表现出不同的趋势。在丰富类群中,变形菌门和酸杆菌门显示出显著的变化。变形菌门在施肥处理后整体呈上升趋势,其中T1、T3、T6处理显著提高了变形菌门在土壤中的丰度,尤其是T3处理中丰度最高。相反,酸杆菌门丰度呈下降趋势,T1、T3、T6处理显著降低了酸杆菌门在土壤中的丰度,尤其是T3处理中丰度最低。此外,硝化螺菌门(Nitrospirota)、疣微菌门(Verrucomicrobiota)、拟杆菌门(Bacteroidota)丰度尽管整体上没有显著差异,但在施肥处理后呈现丰度上升的趋势。在稀有类群中,与丰富类群的变化一致,变形菌门丰度呈上升趋势,而酸杆菌门丰度呈下降趋势。一些类群虽然没有显著差异,但也发生了一致性的变化,如髌骨菌门(Patescibacteria)、拟杆菌门(Bacteroidota)、浮霉菌门(Planctomycetota)整体丰度呈上升趋势,而绿弯菌门(Chloroflexi)与粘细菌门(Myxococcota)呈下降趋势。这些结果表明,在不同的施肥处理条件下细菌亚群的群落结构发生了不同的变化。
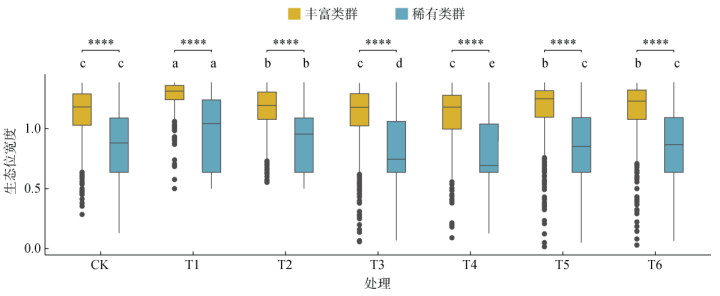
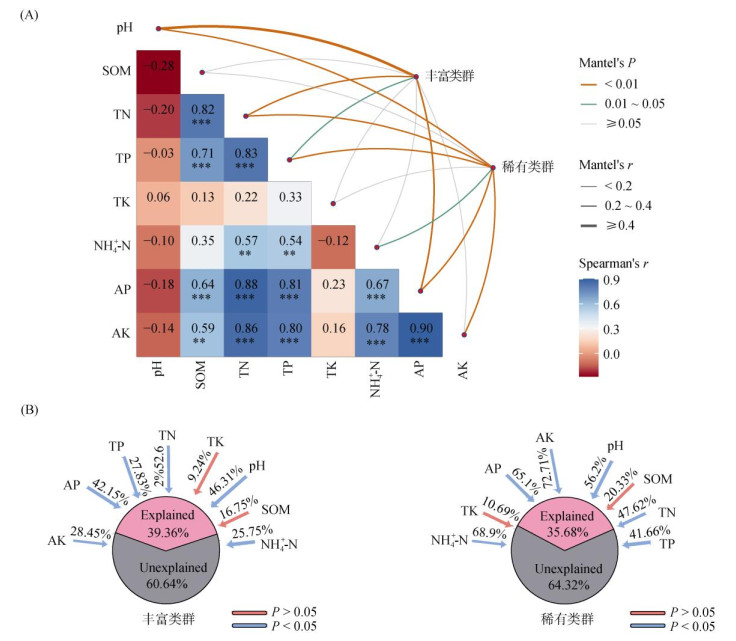
2.4 细菌亚群环境适应性及与环境因子的相关性基于Levins生态位宽度指数的分析表明,不同施肥处理显著改变了细菌亚群的环境适应性,且丰富类群的环境适应性要大于稀有类群(图 3)。通过Mantel检验与RDA分析,本研究探讨了环境因素对群落结构变化的影响,结果如图 4A所示。Mantel检验结果显示,4个环境因子显著影响了丰富类群的群落结构,其中pH (r= 0.46,P= 0.001)的影响最大;对于稀有类群,6个环境因子显著影响群落结构,同样pH对于群落结构(r= 0.39,P=0.001)的影响最大。这表明pH可能是影响丰富和稀有类群的关键因素,RDA分析进一步验证了这一结果。RDA分析结果表明,所有环境因子对于丰富和稀有类群的变化分别解释了39.36% 和35.68% 的细菌群落变化(图 4B),丰富类群中,pH、AP、AK和TN是影响细菌群落结构的主要环境因子;稀有类群中,解释度较高的环境因子主要为AK、NH4+-N、AP和pH。此外,本研究发现相较于丰富类群,稀有类群更受速效养分的影响。
|
(图中****表示细菌亚群间差异在P < 0.000 1水平显著;不同小写字母表示同一细菌亚群不同处理间差异在P < 0.05水平显著 图 3 不同施肥处理下丰富和稀有细菌亚群生态位宽度 Fig. 3 Niche breadths of soil abundant and rare bacterial sub-communities under different fertilization treatments |
|
图 4 土壤理化因子对于土壤丰富和稀有细菌亚群的影响:Mantel检验(A)和冗余分析(B) Fig. 4 The impact of soil physicochemical factors on abundant and rare bacterial sub-communities: Mantel test(A) and redundancy analysis(B) |
本研究发现,长期的化肥施用以及秸秆还田会显著降低土壤pH,而有机肥部分替代化肥会缓解土壤酸化。在自然条件下,土壤在地质时间尺度上的酸化,主要是由于阳离子Ca2+、Mg2+、K+、Na+等的浸出作用、CO2的溶解和有机酸的作用导致[30]。据报道,长期施用化肥可能导致土壤中Ca2+、Mg2+等碱性离子的流失,同时化肥中的氮在土壤中发生硝化作用,NO3–的吸附也会使得土壤中的H+增加,从而引起土壤pH的降低[31-32];秸秆分解增加CO2的水平和土壤中的有机酸,这些弱酸可能加速土壤阳离子浸出,从而导致土壤酸化[30]。然而,有机肥中含有丰富的碱性离子,如Ca2+、Mg2+等,这些碱性离子可以中和土壤中的H+,缓解土壤酸化。同时,有机肥含有丰富的有机物质,这些有机物质在分解过程中释放有机酸,可以提高土壤的酸碱缓冲性[5]。另外,本研究还发现,在有机肥部分替代化肥后,土壤速效养分的含量显著上升,而且在有机肥和化肥相对均衡的情况下,速效养分的含量提升更为显著。这可能是因为有机肥相对于化肥更容易被微生物利用,从而加快了养分的周转速率,有机肥还会增加土壤活性碳和活性氮组分,增强与养分转化有关的微生物和酶的活性,从而提高土壤有效养分[33-34]。
3.2 有机肥部分替代化肥对细菌亚群组成的影响大于对α多样性的影响细菌群落的组成和多样性对土壤生态系统的健康至关重要,了解细菌群落的变化可以帮助评估土壤的整体健康状态[35]。本研究结果发现,长期条件下有机肥部分替代化肥对于细菌亚群的多样性无显著影响。不同的施肥措施对细菌多样性的影响可能在短期产生明显的效应,但在长期内可能不明显[11]。在长期的相同施肥措施下,提供的不同养分可能满足了空间内细菌群落的生长需求,细菌群落可能适应了新的条件,导致多样性的稳定[36]。
然而,微生物在对养分的选择上却表现出一定的偏好,这种偏好性意味着特定类型的养分能够显著促进特定微生物群落的繁殖和生长,这种相互作用的特异性使得微生物在不同环境条件下能够形成具有特定功能和生态角色的群落[37]。本研究发现,不管是丰富类群还是稀有类群,变形菌门(Proteobacteria)丰度在不同施肥处理中整体呈上升趋势,而酸杆菌门(Acidobacteriota)呈下降趋势。一般来说,酸杆菌门更能适应酸性环境[38],然而,本研究却观察到相反的结果,这可能与变形菌门的利用能力相关。变形菌门是土壤中一类分布广泛、适应性强的细菌,具有较高的生态位广度[39],特别是在有机质输入增加的情况下,变形菌门在土壤中化其他微生物具有更强的竞争能力[40]。然而,酸杆菌门对这些有机物质的利用能力较差,变形菌门可能与酸杆菌门进行资源竞争,导致其相对丰度降低。
3.3 有机肥部分替代化肥细菌亚群变化驱动因素深入了解细菌亚群落的动态变化有助于更好地理解土壤生态系统对外界干扰的响应机制。通过探究有机质、氮、磷含量以及pH等对细菌亚群的影响,可以为优化有机肥与化肥的配比、提高土壤质量、促进植物生长提供指导。本研究得出,在不同施肥策略中,土壤pH是影响微生物群落结构变化的关键因素,这与以往研究结果相吻合[14,41]。施用化肥和秸秆会改变土壤的酸碱度,进而作用于微生物群落的组成和功能[36]。由于不同微生物种类对pH的耐受程度不一,pH的波动可能直接决定了某些微生物的存活与繁殖。此外,土壤酶活性作为反映微生物代谢的指标,对土壤pH的变化极为敏感,这种变化不但直接影响酶的活性,还可能间接影响土壤营养的循环[42]。微生物通过各种代谢途径参与土壤营养的转化和流动,因此pH的波动能影响营养元素的形态和可用性,最终影响微生物的生理活性[43]。例如,在酸性条件下,一部分微生物可能增加有机酸的产出,而在碱性土壤中,另一部分微生物则可能偏向于生成碱性代谢物,这些变化反映了微生物对环境pH变化的适应性和代谢多样性。
本研究结果还发现,在长期施肥处理下,稀有细菌类群相较丰富类群更易受环境的影响,且速效养分对于稀有类群的影响更大。稀有类群可能源于生态漂移、空间异质性或随机事件,其相对丰度较低,生态位较窄,数量有限,一般占据了土壤生态系统中特定的生态位,因此对新的环境条件更为敏感[44],更易受速效养分的影响[45]。丰富类群通常占据较宽的生态位,拥有更多资源利用机会,相对较少受到环境过滤的影响[20]。因此,有机肥部分替代化肥对于稀有类群的影响要大于丰富类群。
4 结论有机肥部分替代化肥显著影响了土壤中全量养分和速效养分的含量。秸秆还田显著增加了土壤的有机质含量,减量使用化肥则对提高土壤中速效养分含量效果显著。长期使用化肥虽然导致了土壤pH的下降,但有机肥部分替代化肥可以缓和这一下降趋势,有助于维持土壤细菌群落的多样性。施肥措施的组合变化改变了土壤的理化环境,并影响了土壤细菌亚群的组成,变形菌门(Proteobacteria)的丰度在施肥处理后整体呈上升趋势,而酸杆菌门(Acidobacteriota)的丰度呈下降趋势。从环境适应性角度考虑,丰富和稀有类群细菌在不同施肥处理下表现出不一样的适应性和群落结构。土壤pH是影响微生物群落结构的主要环境驱动因素,稀有类群对环境变化更为敏感,有机肥对其影响超过了对丰富类群的影响。
| [1] |
Rahmati M, Or D, Amelung W, et al. Soil is a living archive of the Earth system[J]. Nature Reviews Earth & Environment, 2023, 4: 421-423 (  0) 0) |
| [2] |
Crowther T W, van den Hoogen J, Wan J, et al. The global soil community and its influence on biogeochemistry[J]. Science, 2019, 365(6455): eaav0550 DOI:10.1126/science.aav0550 (  0) 0) |
| [3] |
张卫信, 申智锋, 邵元虎, 等. 土壤生物与可持续农业研究进展[J]. 生态学报, 2020, 40(10): 3183-3206 (  0) 0) |
| [4] |
郭伟, 李丹丹, 徐基胜, 等. 秸秆与有机无机肥配施对不同质地潮土土壤质量和小麦产量的影响[J/OL]. 土壤学报. https://link.cnki.net/urlid/32.1119.p.20240304.1822.002.
(  0) 0) |
| [5] |
宁川川, 王建武, 蔡昆争. 有机肥对土壤肥力和土壤环境质量的影响研究进展[J]. 生态环境学报, 2016, 25(1): 175-181 (  0) 0) |
| [6] |
温烜琳, 马宜林, 周俊学, 等. 腐熟羊粪有机肥配施无机肥对植烟土壤微生物群落结构和多样性的影响[J]. 土壤, 2023, 55(4): 804-811 (  0) 0) |
| [7] |
Liu J, Wang C K, Guo Z Y, et al. Linking soil bacterial diversity to satellite-derived vegetation productivity: A case study in arid and semi-arid desert areas[J]. Environmental Microbiology, 2021, 23(10): 6137-6147 DOI:10.1111/1462-2920.15683 (  0) 0) |
| [8] |
Luo J P, Liao G C, Banerjee S, et al. Long-term organic fertilization promotes the resilience of soil multifunctionality driven by bacterial communities[J]. Soil Biology and Biochemistry, 2023, 177: 108922 DOI:10.1016/j.soilbio.2022.108922 (  0) 0) |
| [9] |
郭伟, 周云鹏, 陈美淇, 等. 秸秆与有机无机肥配施对潮土关键微生物及小麦产量的影响[J/OL]. 土壤学报. https://link.cnki.net/urlid/32.1119.p.20231013.0850.002.
(  0) 0) |
| [10] |
Liu X P, Wang H T, Wu Y J, et al. Manure application effects on subsoils: Abundant taxa initiate the diversity reduction of rare bacteria and community functional alterations[J]. Soil Biology and Biochemistry, 2022, 174: 108816 DOI:10.1016/j.soilbio.2022.108816 (  0) 0) |
| [11] |
Hu Z M, Ji L F, Wan Q, et al. Short-term effects of bio-organic fertilizer on soil fertility and bacterial community composition in tea plantation soils[J]. Agronomy, 2022, 12(9): 2168 DOI:10.3390/agronomy12092168 (  0) 0) |
| [12] |
刘魁, 田阳阳, 王正旭, 等. 施用有机肥对烤烟根际土壤细菌群落多样性的影响[J]. 云南农业大学学报(自然科学), 2021, 36(6): 977-984 (  0) 0) |
| [13] |
Philippot L, Chenu C, Kappler A, et al. The interplay between microbial communities and soil properties[J]. Nature Reviews Microbiology, 2024, 22(4): 226-239 DOI:10.1038/s41579-023-00980-5 (  0) 0) |
| [14] |
Malik A A, Puissant J, Buckeridge K M, et al. Land use driven change in soil pH affects microbial carbon cycling processes[J]. Nature Communications, 2018, 9(1): 3591 DOI:10.1038/s41467-018-05980-1 (  0) 0) |
| [15] |
中国科学院南京土壤研究所. 土壤理化分析[M].
上海科学技术出版社, 上海, 1978
(  0) 0) |
| [16] |
Caporaso J G, Lauber C L, Walters W A, et al. Ultra-high-throughput microbial community analysis on the Illumina HiSeq and MiSeq platforms[J]. The ISME Journal, 2012, 6(8): 1621-1624 DOI:10.1038/ismej.2012.8 (  0) 0) |
| [17] |
Martin M. Cutadapt removes adapter sequences from high-throughput sequencing reads[J]. EMBnet Journal, 2011, 17(1): 10 DOI:10.14806/ej.17.1.200 (  0) 0) |
| [18] |
Edgar R C, Haas B J, Clemente J C, et al. UCHIME improves sensitivity and speed of Chimera detection[J]. Bioinformatics, 2011, 27(16): 2194-2200 DOI:10.1093/bioinformatics/btr381 (  0) 0) |
| [19] |
Yang S Z, Liebner S, Alawi M, et al. Taxonomic database and cut-off value for processing mcrA gene 454 pyrosequencing data by MOTHUR[J]. Journal of Microbiological Methods, 2014, 103: 3-5 DOI:10.1016/j.mimet.2014.05.006 (  0) 0) |
| [20] |
Liu L M, Yang J, Yu Z, et al. The biogeography of abundant and rare bacterioplankton in the lakes and reservoirs of China[J]. The ISME Journal, 2015, 9(9): 2068-2077 DOI:10.1038/ismej.2015.29 (  0) 0) |
| [21] |
喻言, 刘浩, 匡崇婷, 等. 不同园地土壤细菌亚群的结构与功能分异[J]. 土壤, 2023, 55(5): 1035-1043 (  0) 0) |
| [22] |
Oksanen J, F. Blanchet G, Friendly M, et al. Vegan: Community ecology package[OL]. R Package Version2: 5-7 https://CRAN.R-project.org/package=vegan (2020).
(  0) 0) |
| [23] |
Anderson M J, Willis T J. Canonical analysis of principal coordinates: A useful method of constrained ordination for ecology[J]. Ecology, 2003, 84(2): 511-525 DOI:10.1890/0012-9658(2003)084[0511:CAOPCA]2.0.CO;2 (  0) 0) |
| [24] |
Anderson M J. Permutational multivariate analysis of variance (PERMANOVA)[J]. Wiley statsref: Statistics reference online, 2014, 1-15 (  0) 0) |
| [25] |
McKight P E, Najab J. Kruskal-wallis test[C]//The corsiniencyclopedia of psychology. John Wiley & Sons, Inc, 2010.
(  0) 0) |
| [26] |
Gronau I, Moran S. Optimal implementations of UPGMA and other common clustering algorithms[J]. Information Processing Letters, 2007, 104(6): 205-210 DOI:10.1016/j.ipl.2007.07.002 (  0) 0) |
| [27] |
Wu W X, Logares R, Huang B Q, et al. Abundant and rare picoeukaryotic sub-communities present contrasting patterns in the epipelagic waters of marginal seas in the northwestern Pacific Ocean[J]. Environmental Microbiology, 2017, 19(1): 287-300 DOI:10.1111/1462-2920.13606 (  0) 0) |
| [28] |
Smouse P E, Long J C, Sokal R R. Multiple regression and correlation extensions of the mantel test of matrix correspondence[J]. Systematic Biology, 1986, 35(4): 627-632 (  0) 0) |
| [29] |
McArdle B H, Anderson M J. Fitting multivariate models to community data: A comment on distance-based redundancy analysis[J]. Ecology, 2001, 82(1): 290 DOI:10.1890/0012-9658(2001)082[0290:FMMTCD]2.0.CO;2 (  0) 0) |
| [30] |
Liang F, Li B Z, Vogt R D, et al. Straw return exacerbates soil acidification in major Chinese croplands[J]. Resources, Conservation and Recycling, 2023, 198: 107176 DOI:10.1016/j.resconrec.2023.107176 (  0) 0) |
| [31] |
徐仁扣. 土壤酸化及其调控研究进展[J]. 土壤, 2015, 47(2): 238-244 (  0) 0) |
| [32] |
Hao T X, Zhu Q C, Zeng M F, et al. Impacts of nitrogen fertilizer type and application rate on soil acidification rate under a wheat-maize double cropping system[J]. Journal of Environmental Management, 2020, 270: 110888 DOI:10.1016/j.jenvman.2020.110888 (  0) 0) |
| [33] |
Lazcano C, Gómez-Brandón M, Revilla P, et al. Short-term effects of organic and inorganic fertilizers on soil microbial community structure and function[J]. Biology and Fertility of Soils, 2013, 49(6): 723-733 DOI:10.1007/s00374-012-0761-7 (  0) 0) |
| [34] |
张亚丽, 张娟, 沈其荣, 等. 秸秆生物有机肥的施用对土壤供氮能力的影响[J]. 应用生态学报, 2002, 13(12): 1575-1578 (  0) 0) |
| [35] |
Dang P F, Li C F, Lu C, et al. Effect of fertilizer management on the soil bacterial community in agroecosystems across the globe[J]. Agriculture, Ecosystems & Environment, 2022, 326: 107795 (  0) 0) |
| [36] |
Sun R B, Zhang X X, Guo X S, et al. Bacterial diversity in soils subjected to long-term chemical fertilization can be more stably maintained with the addition of livestock manure than wheat straw[J]. Soil Biology and Biochemistry, 2015, 88: 9-18 DOI:10.1016/j.soilbio.2015.05.007 (  0) 0) |
| [37] |
Liu H, Han H M, Zhang C Z, et al. Patterns of bacterial distance decay and community assembly in different land-use types as influenced by tillage management and soil layers[J]. Ecotoxicology and Environmental Safety, 2023, 266: 115595 DOI:10.1016/j.ecoenv.2023.115595 (  0) 0) |
| [38] |
Ramoneda J, Stallard-Olivera E, Hoffert M, et al. Building a genome-based understanding of bacterial pH preferences[J]. Science Advances, 2023, 9(17): eadf8998 DOI:10.1126/sciadv.adf8998 (  0) 0) |
| [39] |
Delgado-Baquerizo M, Oliverio A M, Brewer T E, et al. A global atlas of the dominant bacteria found in soil[J]. Science, 2018, 359(6373): 320-325 DOI:10.1126/science.aap9516 (  0) 0) |
| [40] |
张旭博, 徐梦, 史飞. 藏东南林芝地区典型农业土地利用方式对土壤微生物群落特征的影响[J]. 农业环境科学学报, 2020, 39(2): 331-342 (  0) 0) |
| [41] |
Zhalnina K, Dias R, de Quadros P D, et al. Soil pH determines microbial diversity and composition in the park grass experiment[J]. Microbial Ecology, 2015, 69(2): 395-406 DOI:10.1007/s00248-014-0530-2 (  0) 0) |
| [42] |
Xu Z W, Yu G R, Zhang X Y, et al. Soil enzyme activity and stoichiometry in forest ecosystems along the North-South Transect in Eastern China (NSTEC)[J]. Soil Biology and Biochemistry, 2017, 104: 152-163 DOI:10.1016/j.soilbio.2016.10.020 (  0) 0) |
| [43] |
Souza L F T, Billings S A. Temperature and pH mediate stoichiometric constraints of organically derived soil nutrients[J]. Global Change Biology, 2022, 28(4): 1630-1642 DOI:10.1111/gcb.15985 (  0) 0) |
| [44] |
Jia X, Dini-Andreote F, Falcão Salles J. Community assembly processes of the microbial rare biosphere[J]. Trends in Microbiology, 2018, 26(9): 738-747 DOI:10.1016/j.tim.2018.02.011 (  0) 0) |
| [45] |
Cui W L, Li R C, Fan Z, et al. Weak environmental adaptation of rare phylotypes sustaining soil multi-element cycles in response to decades-long fertilization[J]. Science of the Total Environment, 2023, 871: 162063 DOI:10.1016/j.scitotenv.2023.162063 (  0) 0) |
2. Anhui Agricultural Waste Fertilizer Utilization and Cultivated Land Quality Improvement Engineering Research Center, Fengyang, Anhui 233100, China
 2025, Vol. 57
2025, Vol. 57


