2. 福建师范大学地理科学学院, 福州 350117
硝化作用是指氨(NH3)在微生物作用下逐步氧化为亚硝酸盐(NO2–),最终生成硝酸盐(NO3–)的过程,是生态系统氮循环的关键步骤[1-2]。其中,氨氧化过程是硝化作用的限速步骤,主要由氨氧化微生物驱动完成[3]。氨氧化微生物包括氨氧化细菌(AOB)、氨氧化古菌(AOA)和完全氨氧化菌(comammox Nitrospira),3者均是完成氨氧化过程的重要微生物。近年来,3种氨氧化微生物在森林、农田、湿地等生态系统中的分布规律与功能活性备受关注。但在森林土壤中,3者的相对重要性和功能活性仍备受争议。Lehtovirta-Morley等[4]发现,酸性森林土壤中因AOA对低pH具有更强的适应性,在氨氧化过程中起主导作用。Hu等[5]通过比较中国300个不同pH的森林土壤样品发现,在森林土壤中完全氨氧化菌的氨单加氧酶α亚基基因(amoA)丰度高于AOA和AOB。因此,不同氨氧化微生物在森林土壤中的分布规律仍有待进一步研究。
硝态氮作为氨氧化过程的终产物和反硝化作用的重要底物,其动态变化直接影响N2O的释放通量,因此明确其驱动机制对森林土壤氮循环调控具有重要意义[6]。森林土壤硝态氮和氨氧化微生物丰度受多种环境因子调控,包括pH、温度、底物浓度以及土壤有机质含量等[7-9]。其中,pH是影响氨氧化微生物群落结构的关键因子,低pH通常抑制AOB的活性,但对AOA和完全氨氧化菌的影响相对较小[10]。此外,土壤含水率[11]、氮沉降输入[12]及植被类型[13]也可能通过改变土壤微环境,间接调控氨氧化微生物的丰度与功能,进而影响硝态氮含量。然而,亚热带森林土壤硝态氮含量的关键驱动因子目前尚不清楚,亟待研究加以阐明。
杉木(Cunninghamia lanceolata)是我国南方重要的速生用材树种,广泛分布于亚热带地区。杉木人工林长期集约化经营导致土壤酸化、肥力下降等问题,严重制约林地生产力。研究表明,杉木人工林土壤硝化速率较天然林下降约40%[14],这种变化与氨氧化微生物群落的适应性重构密切相关。王梦娟等[15]发现杉木人工林的大面积种植显著改变了土壤理化性质(如pH降低、有机质含量下降),进而影响微生物群落的组成与功能。杉木人工林土壤酸化问题可能对氨氧化微生物丰度和活性以及硝态氮含量产生影响,但区域尺度的杉木人工林土壤pH变化如何影响氨氧化微生物丰度特征和硝态氮含量尚不清楚。
基于以上科学问题,本研究以福建省9个典型林场的杉木人工林土壤为研究对象,通过实时荧光定量PCR技术,测定了土壤中氨氧化微生物(AOA、AOB、完全氨氧化菌)的amoA功能基因丰度。本研究假设:在长期酸化背景下,杉木人工林土壤中氨氧化微生物的群落结构发生重构,其中AOA可能由于对低pH环境的适应性而在氨氧化过程中占据主导地位;土壤pH作为关键环境因子,通过调控AOA丰度与活性,进而显著影响硝态氮的积累。本研究旨在阐明杉木人工林土壤氨氧化微生物丰度分布特征及其对硝态氮含量的潜在贡献,并揭示其关键驱动因子,为杉木人工林可持续经营与氮素调控提供理论依据和数据支撑。
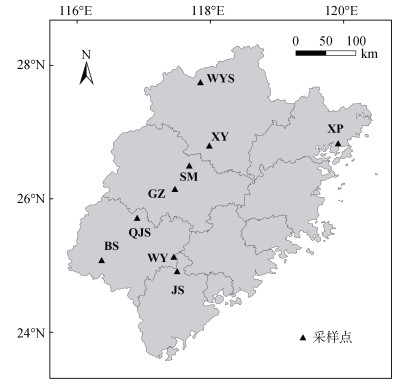
1 材料与方法 1.1 研究区概况研究区为位于中国东南沿海的福建省,其属亚热带季风气候,年均气温为17.5 ~ 21.5 ℃,年降水量为1 450 ~ 2 200 mm,区域内地形以低山丘陵为主,供试土壤类型以酸性红壤、黄红壤为主。本研究分别选取区内漳州市华安金山(JS)、龙岩市漳平五一(WY)、龙岩市上杭白砂(BS)、龙岩市连城邱家山(QJS)、三明市沙县官庄(GZ)、南平市延平峡阳(XY)、宁德市霞浦(XP)7个国有林场,以及南平市武夷山国家公园(WYS)和福建三明森林生态系统国家野外科学观测研究站(SM)进行杉木人工林土样采集。采样点位置如图 1所示。
|
图 1 杉木人工林土壤采样点 Fig. 1 Soil sampling sites of Cunninghamia lanceolata plantations |
于2022年夏季进行土壤样品采集。每个样点选择4块20 ~ 30年树龄的杉木人工林样地(30 m × 30 m),样地间距约200 m。在每个样地内随机选定9株生长状态较一致的代表性杉树,使用直径5 cm的土钻,在每株选定杉树距树干0.5 m处采集4钻深度为15 cm的土芯。每3株杉树的土样混合为1份复合样,因此每个林场共采集12份样本,总计108份。所有样品放置于无菌密封袋后运回实验室。将采集的土样过2 mm筛,去除肉眼可见的石块、细根和掉落物等杂质,充分混匀后分为两部分。一份风干后测定土壤基本理化性质;另一份放置于–80 ℃冰箱,用于后续分子生物学试验。土壤理化性质的测定参照鲁如坤[16]的方法进行。
1.3 土壤DNA提取和定量PCR称取0.5 g土壤,采用FastDNA SPIN Kit for Soil试剂盒(MP Biomedicals,USA),遵循操作说明书流程提取土壤总DNA。提取完成后,DNA样品保存于–80 ℃冰箱,以备后续试验使用。利用实时荧光定量PCR(qPCR)系统(CFX96 Touch™,Bio-Rad Laboratories,USA)测定AOA、AOB及完全氨氧化菌分支A(Clade A)的功能基因amoA丰度,引物序列分别为Arch-amoAF/Arch-amoAR、amoA-1F/amoA-2R[17]和CA377f/C576r[18]。由于完全氨氧化菌分支B(Clade B)在供试土壤样品中含量极低,后续研究不予以考虑。qPCR所用的反应体系均为20 μL,包含10 μL SYBR预混液、0.2 μL正向引物、0.2 μL反向引物、8.6 μL灭菌去离子水和1 μL DNA模板。每个样品3个重复,并设置3个阴性对照。采用含目标基因的质粒DNA进行10倍梯度稀释,建立标准曲线,具体方法参照先前研究[19]。标准曲线的扩增效率为82% ~ 89.4%,R2为0.996 ~ 0.999。
1.4 数据处理与分析采用SPSS 23.0软件进行数据分析,在数据分析之前对数据进行正态性检验和齐性检验,并对不满足正态分布的数据进行必要转换,以满足方差分析要求。不同林场氨氧化微生物的基因丰度差异利用单因素方差分析和多重比较进行检验。利用成对比较分析杉木人工林土壤中不同氨氧化微生物基因丰度差异。通过斯皮尔曼相关性探究土壤理化性质与氨氧化微生物基因丰度的相关关系。采用R.4.0.3软件“randomForest”包进行随机森林分析,再利用SPSS 23.0软件进行逐步回归分析,进一步探讨影响硝态氮含量的关键因子。利用Origin 2021作图。
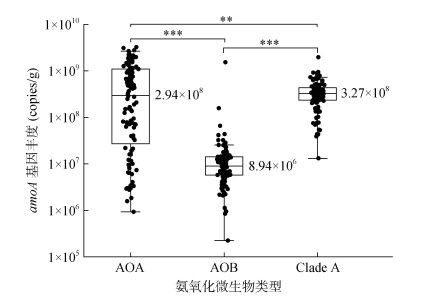
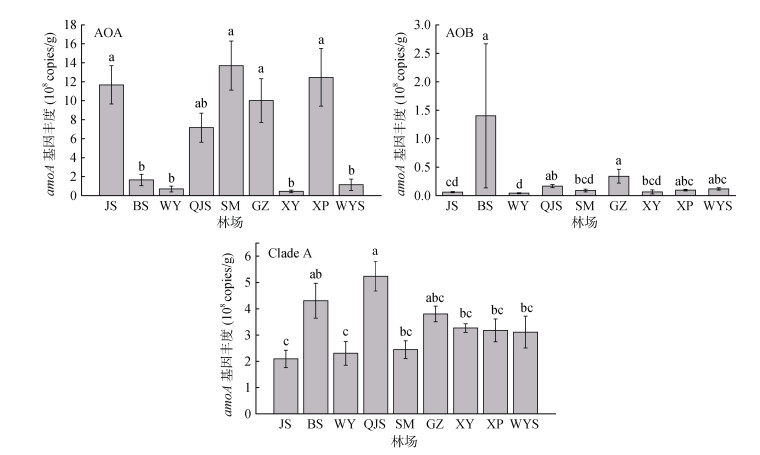
2 结果与分析 2.1 土壤氨氧化微生物的分布特征从图 2可以看出,供试土壤AOA、AOB和Clade A的amoA基因丰度存在显著差异。AOA的amoA基因丰度为9.29×105 ~ 3.23×109 copies/g,平均值为2.94×109 copies/g。AOB的amoA基因丰度为2.24×105 ~ 1.53×109 copies/g,平均值为8.94×106 copies/g。Clade A的amoA基因丰度为1.32×107 ~ 1.96×109 copies/g,平均值为3.27×108 copies/g。AOA和Clade A的amoA基因丰度极显著高于AOB,且Clade A的amoA基因丰度显著高于AOA。不同林场下AOA、AOB和Clade A的amoA基因丰度存在显著差异(图 3)。JS、SM、GZ、XP林场土壤AOA的amoA基因丰度显著高于BS、WY、XY、WYS,其中SM林场土壤AOA的amoA基因丰度最高,为1.37×109 copies/g。AOB的amoA基因丰度在BS林场最高,为1.40×108 copies/g,显著高于JS、WY、SM和XY林场。Clade A的amoA基因丰度在QJS林场最高,JS林场最低,分别为5.24×108 copies/g和2.09×108 copies/g。
|
(图中**、***分别表示差异达P < 0.01、P < 0.001显著水平,下同) 图 2 杉木人工林土壤不同类型氨氧化微生物功能基因丰度的差异 Fig. 2 Differences of genes abundances between different soil ammonia-oxidizing microorganisms in Cunninghamia lanceolata plantations |
|
(JS、WY、BS、QJS、SM、GZ、XY、WYS和XP分别为华安金山国有林场、漳平五一国有林场、上杭白砂国有林场、连城邱家山国有林场、福建三明森林生态系统国家野外科学观测研究站、沙县官庄国有林场、南平峡阳国有林场、武夷山国家森林公园和霞浦国有林场;柱图上方不同小写字母表示不同林场间差异显著(P < 0.05),下同) 图 3 不同林场下不同类型土壤氨氧化微生物功能基因丰度 Fig. 3 Genes abundances of different soil ammonia-oxidizing microorganisms in different Cunninghamia lanceolata plantations |
斯皮尔曼相关性分析(表 1)表明,AOA的amoA基因丰度与pH呈极显著负相关(P < 0.001),与碳氮比、铵态氮和有效磷含量呈极显著正相关(P < 0.001),与全氮含量(P < 0.05)和含水率(P < 0.01)呈显著正相关。AOB的amoA基因丰度仅与有效磷含量呈极显著正相关(P < 0.001),与其他土壤理化因子均无显著相关性。Clade A的amoA基因丰度与有效磷含量呈极显著正相关(P < 0.001),与含水率呈显著正相关(P < 0.05)。
|
|
表 1 土壤氨氧化微生物功能基因丰度与理化性质之间的斯皮尔曼相关性 Table 1 Spearman coefficients between soil ammonia-oxidizing genes abundance and physicochemical properties |
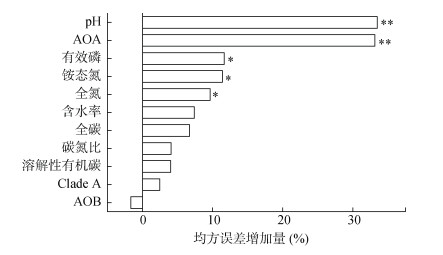
随机森林分析表明,土壤pH和AOA是影响硝态氮含量的关键环境因子(图 4)。有效磷、铵态氮和全氮对硝态氮含量也有显著影响。逐步回归分析表明,AOA、pH、全氮和AOB可以解释硝态氮含量84.70% 的变异(表 2)。其中,AOA是影响硝态氮含量最关键的正向因子(t=10.580,P < 0.001),而pH是最关键的负向影响因子(t= –6.403,P < 0.001)。这些结果表明,土壤pH和AOA在调控杉木人工林土壤硝酸盐含量过程中起着关键的作用。
|
(*、**分别表示在P < 0.05、P < 0.01水平影响显著) 图 4 土壤氨氧化微生物功能基因丰度及土壤理化性质与硝态氮含量的随机森林分析 Fig. 4 Random forest analysis of soil ammonia-oxidizing gene abundances and soil physiochemical properties with NO– 3-N contents |
|
|
表 2 土壤氨氧化微生物功能基因丰度和土壤理化性质对硝态氮含量的逐步回归分析(n=108) Table 2 Stepwise regression analysis of soil ammonia-oxidizing gene abundances and physicochemical properties to NO– 3-N contents |
从图 5可以看出,AOA功能基因丰度与土壤硝态氮含量呈显著正相关(P < 0.01),拟合的R2为0.728。AOB和Clade A功能基因丰度与土壤硝态氮含量无显著相关性(P > 0.05)。
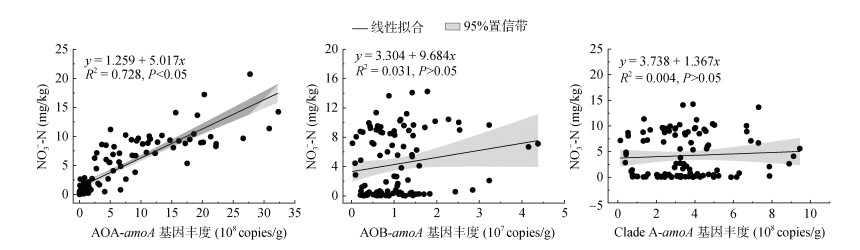
|
图 5 土壤氨氧化微生物功能基因丰度与硝态氮含量的关系 Fig. 5 Relationships between soil ammonia-oxidizing gene abundances and NO– 3-N contents |
土壤AOA、AOB和完全氨氧化菌Clade A功能基因amoA丰度已有大量研究,且大多数研究指出土壤AOA、AOB和Clade A存在着明显的生态位分异[20]。本研究结果显示,亚热带森林土壤中,Clade A的amoA丰度最高,AOB的amoA丰度最低,这与Li等[21]在澳大利亚森林土壤上的研究结果一致,表明Clade A和AOA在酸性森林土壤中占优势。
Clade A是亚热带森林土壤中丰度最高的氨氧化微生物,显著高于AOA,这可能是Clade A和AOA两者对底物的亲和力不同导致的。已有研究表明,Clade A在农田[22]、森林土壤[23]、海洋[24]等环境中均被检出,并且其基因丰度均高于AOA。Clade A在寡营养环境中的基因丰度显著高于AOA,核心机制是其氨单加氧酶对底物具有更高的亲和力。Kits等[25]通过酶动力学试验证实,Clade A表观半饱和常数Km(NH3)约为63 nmol/L,低于AOA;Palomo等[26]发现,Clade A的氨单加氧酶具有独特的跨膜螺旋和底物通道,增强了铵的跨膜运输能力。因此,在营养贫瘠的环境中,完全氨氧化菌Clade A可能拥有更强的竞争力。此外,Clade A的基因组中含有碱性磷酸酶基因phoD,在低磷条件下可以高度表达,并且该基因在AOA中未被检出。亚热带森林土壤通常具有高度风化和低养分有效性的特征[27],尤其磷素有效性特别低,因此,Clade A可能在亚热带森林土壤中具有更强的竞争优势。Koch等[28]进一步指出,Clade A具有多样的底物代谢途径,除氨氮以外,还可利用尿素、氰酸盐等作为氮源,在环境中具有强适应性。
本研究中,亚热带森林土壤AOA的amoA基因丰度显著高于AOB,这与大多数研究结果一致[29-30],这可能与土壤pH有较大关系。前人研究表明,AOB更适合在pH为中性或碱性条件下生存[31]。相反,AOA在低pH的环境下更占优势,在供试土壤中其功能基因丰度与pH呈显著负相关(表 1)。当土壤pH较低时,铵态氮形成NH3过程受阻,从而形成对AOA生长有利的环境。而AOA和AOB对NH3亲和力不同,在低NH3环境中,AOA对NH3的亲和力高于AOB,因此AOA往往更具竞争力。斯皮尔曼相关性分析发现,AOA的amoA基因丰度与土壤全氮、含水率和碳氮比呈显著正相关,这表明AOA的amoA基因丰度与土壤碳源、氮源和水分含量有密切联系。
3.2 杉木人工林硝态氮含量的关键驱动因子本研究表明,土壤pH、AOA、有效磷、铵态氮、全氮均对硝态氮含量有显著影响。随机森林模型分析与逐步回归分析进一步揭示,土壤pH和AOA是硝态氮含量的核心驱动因子。其中,pH对硝态氮含量具有显著负影响,而AOA则表现为显著正效应。因此,AOA是福建省杉木人工林土壤硝化作用的关键驱动者。这与众多前人的研究结果类似[32-33]。本研究中,AOA的amoA基因丰度显著高于AOB,AOA的amoA基因丰度与土壤pH呈显著负相关,表明AOA更适合在酸性环境中生存,与Shi等[34]的发现一致。Shi等[34]发现,AOA对酸性环境具有高度适应性,其氨单加氧酶对低浓度NH3的亲和力远高于AOB。这种特性使AOA在酸性、低铵条件下成为硝化作用的主要驱动者。对于Clade A,虽然氨氧化动力学分析发现,它对NH3的半饱和常数约为63 nmol/L[25],但实际环境中的NH3浓度往往较高。本研究发现,虽然Clade A的amoA基因丰度高于AOA,但AOA才是硝态氮含量的主要贡献者。利用DNA-SIP技术,Shi[34]等同样发现,在酸性森林土壤中Clade A的amoA基因丰度较高,但AOA才是硝化作用的关键驱动因子。有研究表明,氨氧化微生物的丰度与活性并不同步,没有直接的关联[35]。Tan等[36]研究也表明,在3种酸性土壤中AOA是驱动硝化作用的主要氨氧化微生物,贡献率为67.4% ~ 83.2%,而Clade A的贡献率仅为3.3% ~ 8.5%。这可能是由于Clade A在亚热带森林土壤中拥有极高的亲和力,导致其最大氨氧化速率过低,从而使其对硝化作用的贡献低于AOA。而AOB通常在中性或碱性土壤中对硝化作用的贡献更大[37]。大多数研究表明,pH与硝态氮含量呈正相关,这是因为在较低pH条件下,氨氧化微生物活性往往会受到抑制,从而影响硝态氮的含量[38]。然而,在本研究供试土壤中,AOA对硝态氮含量起着主导作用,而AOA的amoA基因丰度和pH呈负相关,因此,AOA的影响可能掩盖了pH的直接影响。
4 结论本研究系统分析了福建省杉木人工林土壤中3类氨氧化微生物的分布格局及其与硝态氮含量的关系。结果表明,完全氨氧化菌Clade A和AOA在土壤中的丰度显著高于AOB,其中Clade A丰度最高。尽管Clade A在数量上占优势,但仅AOA的丰度与硝态氮含量呈显著正相关,表明AOA是该区域硝态氮形成的主要微生物驱动者。此外,AOA丰度与土壤pH显著负相关,与多种养分指标(如碳氮比、铵态氮含量和有效磷含量)显著正相关。随机森林分析与逐步回归分析进一步证实,AOA和pH是影响硝态氮含量的关键因子。本研究不仅明确了AOA在杉木人工林土壤硝化过程中的主导地位,还揭示了土壤酸化与营养条件对其生态功能的协同调控作用。这一发现对理解亚热带人工林氮循环微生物机制具有重要意义,并为改善土壤健康、提升林地氮素利用效率及指导可持续经营提供了理论依据。
| [1] |
Kowalchuk G A, Stephen J R. Ammonia-oxidizing bacteria: A model for molecular microbial ecology[J]. Annual Review of Microbiology, 2001, 55: 485-529 DOI:10.1146/annurev.micro.55.1.485 (  0) 0) |
| [2] |
贺纪正, 张丽梅. 土壤氮素转化的关键微生物过程及机制[J]. 微生物学通报, 2013, 40(1): 98-108 (  0) 0) |
| [3] |
Stein L Y, Klotz M G. The nitrogen cycle[J]. Current Biology, 2016, 26(3): R94-R98 DOI:10.1016/j.cub.2015.12.021 (  0) 0) |
| [4] |
Lehtovirta-Morley L E, Stoecker K, Vilcinskas A, et al. Cultivation of an obligate acidophilic ammonia oxidizer from a nitrifying acid soil[J]. PNAS, 2011, 108(38): 15892-15897 DOI:10.1073/pnas.1107196108 (  0) 0) |
| [5] |
Hu H W, He J Z. Comammox—a newly discovered nitrification process in the terrestrial nitrogen cycle[J]. Journal of Soils and Sediments, 2017, 17(12): 2709-2717 DOI:10.1007/s11368-017-1851-9 (  0) 0) |
| [6] |
郑翔, 刘琦, 曹敏敏, 等. 森林土壤氧化亚氮排放对氮输入的响应研究进展[J]. 土壤学报, 2022, 59(5): 1190-1203 (  0) 0) |
| [7] |
Zhang L M, Hu H W, Shen J P, et al. Ammonia-oxidizing Archaea have more important role than ammonia-oxidizing bacteria in ammonia oxidation of strongly acidic soils[J]. The ISME Journal, 2012, 6(5): 1032-1045 DOI:10.1038/ismej.2011.168 (  0) 0) |
| [8] |
Tourna M, Freitag T E, Nicol G W, et al. Growth, activity and temperature responses of ammonia-oxidizing Archaea and bacteria in soil microcosms[J]. Environmental Microbiology, 2008, 10(5): 1357-1364 DOI:10.1111/j.1462-2920.2007.01563.x (  0) 0) |
| [9] |
Verhamme D T, Prosser J I, Nicol G W. Ammonia concentration determines differential growth of ammonia-oxidising Archaea and bacteria in soil microcosms[J]. The ISME Journal, 2011, 5(6): 1067-1071 DOI:10.1038/ismej.2010.191 (  0) 0) |
| [10] |
Li H, Weng B S, Huang F Y, et al. pH regulates ammonia-oxidizing bacteria and Archaea in paddy soils in Southern China[J]. Applied Microbiology and Biotechnology, 2015, 99(14): 6113-6123 DOI:10.1007/s00253-015-6488-2 (  0) 0) |
| [11] |
冯蒙蒙, 林永新, 樊剑波, 等. 温度和氮添加对旱地红壤反硝化功能基因丰度的影响[J]. 土壤, 2023, 55(3): 562-568 DOI:10.13758/j.cnki.tr.2023.03.013 (  0) 0) |
| [12] |
张锦秀, 曹聪, 陆宇明, 等. 短期氮沉降对速生期杉木人工林细根性状的影响及其驱动因素[J]. 亚热带资源与环境学报, 2025, 20(1): 136-144 (  0) 0) |
| [13] |
郑威, 李晨曦, 谭玲, 等. 南亚热带桉树人工林与典型乡土树种人工林土壤磷组分及磷吸附特性比较[J]. 土壤, 2020, 52(5): 1017-1024 DOI:10.13758/j.cnki.tr.2020.05.020 (  0) 0) |
| [14] |
邓米林, 冯蒙蒙, 刘小飞, 等. 模拟氮沉降降低亚热带米槠天然林土壤氧化亚氮排放潜势[J]. 应用生态学报, 2022, 33(10): 2705-2710 (  0) 0) |
| [15] |
王梦娟, 黄志群, 张冰冰, 等. 不同林龄杉木人工林土壤硝化和反硝化作用[J]. 应用生态学报, 2023, 34(1): 18-24 (  0) 0) |
| [16] |
鲁如坤. 土壤农业化学分析方法[M].
中国农业科技出版社, 2000
(  0) 0) |
| [17] |
唐修峰, 秦华, 匡璐, 等. 基于宏基因组学的酸性森林土壤氨氧化微生物群落特征研究[J]. 土壤学报, 2021, 58(5): 1311-1321 (  0) 0) |
| [18] |
Jiang R, Wang J G, Zhu T, et al. Use of newly designed primers for quantification of complete ammonia-oxidizing (comammox) bacterial clades and strict nitrite oxidizers in the genus Nitrospira[J]. Applied and Environmental Microbiology, 2020, 86(20): e01775-20 (  0) 0) |
| [19] |
宛颂, 段春健, 樊剑波, 等. 旱地红壤反硝化功能基因丰度对长期施肥的响应[J]. 应用生态学报, 2020, 31(11): 3729-3736 (  0) 0) |
| [20] |
Bi R Y, Xu X T, Wang B X, et al. Evidence of complete ammonia-oxidizing microbial communities and their contribution to N2O emissions in typical vegetable fields across China[J]. Soil Biology and Biochemistry, 2024, 194: 109423 DOI:10.1016/j.soilbio.2024.109423 (  0) 0) |
| [21] |
Li C Y, Hu H W, Chen Q L, et al. Niche differentiation of clade A comammox Nitrospira and canonical ammonia oxidizers in selected forest soils[J]. Soil Biology and Biochemistry, 2020, 149: 107925 DOI:10.1016/j.soilbio.2020.107925 (  0) 0) |
| [22] |
Bartelme R P, McLellan S L, Newton R J. Freshwater recirculating aquaculture system operations drive biofilter bacterial community shifts around a stable nitrifying consortium of ammonia-oxidizing Archaea and comammox Nitrospira[J]. Frontiers in Microbiology, 2017, 8: 101 (  0) 0) |
| [23] |
Fowler S J, Palomo A, Dechesne A, et al. Comammox Nitrospira are abundant ammonia oxidizers in diverse groundwater-fed rapid sand filter communities[J]. Environmental Microbiology, 2018, 20(3): 1002-1015 DOI:10.1111/1462-2920.14033 (  0) 0) |
| [24] |
Xu Y, Lu J, Wang Y, et al. Diversity and abundance of comammox bacteria in the sediments of an urban lake[J]. Journal of Applied Microbiology, 2020, 128(6): 1647-1657 DOI:10.1111/jam.14593 (  0) 0) |
| [25] |
Kits K D, Sedlacek C J, Lebedeva E V, et al. Kinetic analysis of a complete nitrifier reveals an oligotrophic lifestyle[J]. Nature, 2017, 549(7671): 269-272 DOI:10.1038/nature23679 (  0) 0) |
| [26] |
Palomo A, Pedersen A G, Fowler S J, et al. Comparative genomics sheds light on niche differentiation and the evolutionary history of comammox Nitrospira[J]. The ISME Journal, 2018, 12(7): 1779-1793 DOI:10.1038/s41396-018-0083-3 (  0) 0) |
| [27] |
Yu Z P, Wang M H, Huang Z Q, et al. Temporal changes in soil C-N-P stoichiometry over the past 60 years across subtropical China[J]. Global Change Biology, 2018, 24(3): 1308-1320 DOI:10.1111/gcb.13939 (  0) 0) |
| [28] |
Koch H, van Kessel M A H J, Lücker S. Complete nitrification: Insights into the ecophysiology of comammox Nitrospira[J]. Applied Microbiology and Biotechnology, 2019, 103(1): 177-189 DOI:10.1007/s00253-018-9486-3 (  0) 0) |
| [29] |
Gubry-Rangin C, Hai B, Quince C, et al. Niche specialization of terrestrial archaeal ammonia oxidizers[J]. PNAS, 2011, 108(52): 21206-21211 DOI:10.1073/pnas.1109000108 (  0) 0) |
| [30] |
Shi X Z, Sun J W, Wang J Q, et al. Tree species richness and functional composition drive soil nitrification through ammonia-oxidizing Archaea in subtropical forests[J]. Soil Biology and Biochemistry, 2023, 187: 109211 DOI:10.1016/j.soilbio.2023.109211 (  0) 0) |
| [31] |
石秀丽, 郭萌萌, 张莹, 等. 单步硝化作用与全程氨氧化微生物研究进展[J]. 草业学报, 2018, 27(7): 196-203 (  0) 0) |
| [32] |
Ouyang Y, Norton J M, Stark J M, et al. Ammonia-oxidizing bacteria are more responsive than Archaea to nitrogen source in an agricultural soil[J]. Soil Biology and Biochemistry, 2016, 96: 4-15 DOI:10.1016/j.soilbio.2016.01.012 (  0) 0) |
| [33] |
Xie J, Wang Z F, Wang Y, et al. Manure combined with biochar reduces rhizosphere nitrification potential and amoA gene abundance of ammonia-oxidizing microorganisms in acid purple soil[J]. Applied Soil Ecology, 2023, 181: 104660 DOI:10.1016/j.apsoil.2022.104660 (  0) 0) |
| [34] |
Shi X Z, Hu H W, Wang J Q, et al. Niche separation of comammox Nitrospira and canonical ammonia oxidizers in an acidic subtropical forest soil under long-term nitrogen deposition[J]. Soil Biology and Biochemistry, 2018, 126: 114-122 DOI:10.1016/j.soilbio.2018.09.004 (  0) 0) |
| [35] |
Xia F, Wang J G, Zhu T, et al. Ubiquity and diversity of complete ammonia oxidizers (comammox)[J]. Applied and Environmental Microbiology, 2018, 84(24): e01390-18 (  0) 0) |
| [36] |
Tan C, Yin C, Zhang L, et al. Deciphering the functional importance of comammox vs. canonical ammonia oxidisers in nitrification and N2O emissions in acidic agricultural soils[J]. Soil Biology and Biochemistry, 2024, 193: 109415 DOI:10.1016/j.soilbio.2024.109415 (  0) 0) |
| [37] |
Shen J P, Zhang L M, Di H J, et al. A review of ammonia-oxidizing bacteria and Archaea in Chinese soils[J]. Frontiers in Microbiology, 2012, 3: 296 (  0) 0) |
| [38] |
Huygens D, Boeckx P, Templer P, et al. Mechanisms for retention of bioavailable nitrogen in volcanic rainforest soils[J]. Nature Geoscience, 2008, 1(8): 543-548 DOI:10.1038/ngeo252 (  0) 0) |
2. School of Geographical Sciences, Fujian Normal University, Fuzhou 350117, China
 2025, Vol. 57
2025, Vol. 57


