2. 中国科学院大学, 北京 100049;
3. 中国科学院红壤生态实验站, 江西鹰潭 335211
我国红壤分布广泛,总面积达218万km2,约占全国土地总面积的22.7%,在农业及经济社会可持续发展中占据关键地位[1]。总体而言,我国红壤区兼具地域广阔、气候条件优越(地处热带与亚热带湿润区)、开发潜力显著等特征[2]。然而,受长期土壤酸化影响,该区域作物生产普遍遭受土壤结构劣变、铝毒胁迫及磷素固定等多重因素制约,导致作物减产。破解红壤区农业生产瓶颈,已成为提升作物产能、保障国家粮食安全的核心任务。
酸性土壤中的有益微生物往往具有耐酸性特征,能够在低pH环境下存活并发挥功能。一些有益微生物能够通过多种机制改善植物生长和提高养分利用效率,帮助植物适应酸性环境[3]。例如,丛枝菌根真菌通过共生机制直接与植物根系互作,为植物提供磷等难溶养分;枯草芽孢杆菌可增强植物对磷的吸收[3];变形菌可以利用光合作用储存能量和降解有害物质,并且可以固定氮[4];酸杆菌通过分解生物螯合物、分泌细胞外多糖促进植物生长[5-6]。
合成菌群(SynCom)是指通过人工设计和构建的微生物群落,旨在实现特定生态功能或生物技术的应用[7]。在红壤中,合成菌群的应用对于提高植物抗逆性、促进养分吸收和改善土壤健康具有重要意义[8]。但是,由于难以定殖等原因,实验室制备的合成菌群难以应用到实际的农业生产中[9]。本文将系统阐释红壤合成菌群的成员筛选、菌群互作以及根际对话与定殖,旨在筛选适配菌株并构建高效合成菌群,以提升红壤稻区作物产量。
1 红壤合成微生物组成员的高效筛选 1.1 本土微生物在合成微生物组中的重要性目前的合成微生物菌群日趋成熟,在农业生产中的病害防治、养分利用等多方面起到了重要作用[10]。然而,基于实验室微生物功能筛选构建的合成菌群在红壤中的农业生产应用,其效果往往被低估[11]。这是由于“外来”菌群在红壤中的应用需要面临两个挑战。首先,酸性土壤是一个对微生物强筛选的环境,Al3+潜在的毒性对微生物具有损坏细胞膜、诱导细胞渗透调节混乱等毒害作用,快速抑制了微生物生长[12-13]。其次,接种至土壤中的微生物菌剂,往往会面对来自“本土”微生物的竞争[14-15],受到源于本土微生物产生的抗菌代谢产物的抑制[16],如具有广谱抗菌效能的短链脂肪酸(乙酸、丁酸)[17]。例如,土壤中存在的丁酸梭菌(Clostridium butyricum) 能通过产生丁酸对多种外来细菌产生拮抗作用[18];本土的吩嗪-1-羧酰胺产生菌假单胞菌(Pseudomonas chlororaphis PCL1391)通过控制番茄脚腐病和根腐病来防治由尖孢镰刀菌引起的病害[19]。
基于本土微生物设计合成菌群是应对上述挑战的一个解决方法[20]。与外来微生物相比,本土微生物表现出更高的定殖率以及更稳定的功能表达[21]。面对酸铝胁迫时,本土微生物能通过细胞质膜上的质子交换、细胞壁修饰等机制,保持较高的代谢活性,表现出明显耐酸铝特征。例如,一株从红壤稻田中分离出的本土微生物铜绿假单胞菌(Pseudomonas aeruginosa)通过胞外质子化作用,即将周边介质中的质子吸附到细胞膜表面,降低了周边介质中的pH。通过该过程,铜绿假单胞菌不仅维持了本身在酸性环境下的高代谢活性,同时提高了合成菌群其他成员的代谢活性[8]。
面对本土微生物的竞争,可基于本土核心微生物进行合成菌群的构建。一般而言,本土核心微生物往往具有以下优势,以帮助其在与其他微生物的竞争中形成稳定群落(图 1):①本土核心菌群往往具备高效的养分吸收能力,能够快速占据根系表面和土壤颗粒的可利用空间,从而抢占碳、氮、磷等关键资源[22];②许多本土核心微生物可通过释放如吩嗪类[23]、多肽类及糖肽类[24]等抗菌代谢产物抑制其他细菌或真菌的生长[25];③核心微生物常与植物形成互惠关系(如促进养分吸收、分泌根系诱导物),获得植物分泌的碳源。特别是这类碳源(如嘌呤和嘧啶等核苷酸类物质)能特异性地向核心有益微生物进行供给,从而帮助其在根际竞争中获得额外的资源支持[26]。这种植物–微生物–微生物的三方协同进一步强化核心菌群的竞争地位[27]。例如,将贝莱斯芽孢杆菌(Bacillus velezensis)接种到黄瓜根际,通过趋化吸引、代谢互养和混合生物膜形成等机制增加了土著益生菌施氏假单胞菌的定殖[28]。这些结果进一步佐证了基于本土微生物构建的合成微生物菌剂在农业生产中定殖及稳定发挥功能的重要作用。
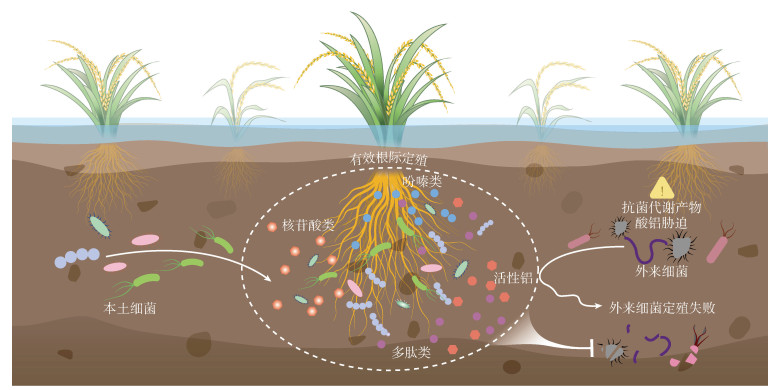
|
图 1 本土细菌的定殖优势 Fig. 1 The colonization advantage of local bacteria |
鉴于本土微生物在合成菌剂中的核心作用,特别是红壤存在的酸铝、养分胁迫等环境因素对微生物的筛选提出了更高要求。因此,筛选和鉴定具有耐逆特性的本土微生物显得尤为重要。目前,已有多种方法被开发用于土壤中原位分离筛选微生物菌株,例如通过iChip技术分离大量环境中的单株微生物[29]、通过D2O标记–单细胞拉曼技术原位筛选溶磷功能菌[30]等。其中,利用D2O标记–单细胞拉曼技术结合高通量功能筛选平台[30],能够对酸铝逆境关键功能菌进行高通量系统挖掘,实现如质子化耐酸铝、溶磷固氮等功能菌的筛选[8]。这项技术通过使用具有稳定同位素标记的重水,依据氘的摄入量作为细菌代谢活动的可靠指标进行筛选(表征细菌的代谢活性[31])。本课题组之前的一项研究中,基于拉曼–D2O方法,对从酸性土壤水稻根际中分离的248株细菌,评估了其中12株代表性菌株在pH = 4.0时的耐铝水平,通过评价C-D比值,筛选出具有耐铝胁迫的菌株[8]。其中筛选到的一株铜绿假单胞菌(Pseudomonas sp.)与一株红城红球菌(Rhodococcus sp.)通过胞外质子化作用缓释环境中的pH,提高了自身的酸铝耐受能力。
基于拉曼技术的单细胞高通量筛选技术,研究人员进一步开发了AI-RACS系统[32]。该系统包括4个核心功能模块:微流控芯片、单细胞拉曼光谱采集模块、光镊模块和单细胞收集模块。系统集成了人工智能辅助的图像识别算法实现精准单细胞识别[33],耦合光镊技术实现高质量单细胞拉曼光谱的采集[34],目标细胞自动路径规划分选。AI-RACS系统对于高速移动的细胞样品能通过微流控芯片模块将目标单细胞单独分散到下游单细胞测序单元的不同容器中,解决了之前FlowRACS平台等技术的挑战[35]。此外,对于静止的细胞样品,AI-RACS系统通过AI识别以及定位目标细胞,同时规划分离路径,再结合光镊技术对目标细胞进行分离。相较于之前的RAGE平台,极大程度地提高了分离单细胞的效率[34]。基于该系统,Li等[30]利用以Ca3(PO4)2为唯一无机磷源或以卵磷脂为唯一有机磷源的污水样本(预先去除可溶性磷源),结合D2O孵育,通过单细胞拉曼光谱中C-D特征峰的强弱,以单细胞精度定量表征污水中微生物的解磷活性,进而建立了原位解磷菌的单细胞识别–分选–培养流程,原位识别、分选和培养出了丛毛单胞菌(Comamonas spp.)、不动杆菌(Acinetobacter spp.)等高效原位有机解磷菌。
2 构建结构稳定的合成微生物群落 2.1 交叉喂养是构建稳定合成微生物群落的重要支撑交叉喂养指生物体或微生物在代谢过程中互相提供某些代谢产物,以满足对方的代谢需求[36]。交叉喂养在微生物群落中已被证明普遍存在于细菌、真菌、古菌群落中[37-38]。交叉喂养在微生物群落的结构与功能中扮演着至关重要的角色,通过共享资源、增强群落适应性、减少物种间竞争压力等多重机制,对维持群落的长期稳定性和多样性[39]具有深远影响。
交叉喂养通过提供人工不可合成的微生物必需养分,增加了可培养微生物数量。据估计,只有不到1% 的细菌物种可被实验室培养[40]。而在实验室的微生物培养条件中往往包含了足够的养分,通常不会因微生物竞争导致可培养微生物物种减少[39]。因此,一项研究认为,可能主要是由于不可合成且必需的微生物养分物质是培养微生物的关键限制因子[41]。例如,微生物普遍存在氨基酸的合成缺陷,代谢途径分析表明,24%[42] ~ 98%[43]的细菌缺乏合成至少一种氨基酸的能力。而相较于单独培养,大量氨基酸合成缺陷细菌的共培养实验表明,细菌共培养能提高实验室可培养细菌的数量[38, 44]。因此,识别与构建交叉喂养关系对挖掘土壤微生物资源以及构建稳定的合成微生物群落具有重要贡献。
交叉喂养通过增强群落适应性提高群落以及功能的稳定性。由红城红球菌(Rhodococcus erythropolis)和铜绿假单胞菌(Pseudomonas aeruginosa)组成的土壤细菌功能菌群的铝耐受性优于其单独成员。这是由于铜绿假单胞菌会产生次生代谢产物2-庚基-4-喹诺酮(2-heptyl-1H-quinolin-4-one,HHQ),该物质被红城红球菌高效降解,此降解过程减少了铜绿假单胞菌群体生长的限制,增强了铜绿假单胞菌在铝胁迫下的代谢活性;此外,红城红球菌通过分支酸合成途径将HHQ转化为色氨酸,促进肽聚糖的合成,从而提高细胞壁的稳定性和铝耐受性[45]。该项研究表明,借助交叉喂养,能同时增强合成菌群的两个成员对于红壤酸铝胁迫的适应性,以及行使耐逆功能的稳定性。
交叉喂养通过减少微生物群落间的竞争提高合成菌群的定殖能力。资源竞争和代谢交叉喂养是微生物群落组装的两种核心驱动力[46],接种到土壤中的合成微生物群落往往面对来自本土微生物的竞争从而降低预期功能强度[14]。研究表明,外来益生菌能否在本土微生物群落中成功定殖,与其是否处于交叉代谢网络中、是否与本土微生物建立合作关系密切相关[46]。
综上所述,考虑合成菌群内部以及与本土微生物间的交叉喂养,是挖掘土壤微生物潜力、增强合成菌群功能、提高合成菌群定殖能力的关键。
2.2 交叉喂养的识别技术及未来发展识别及利用微生物群落间的交叉喂养关系,首先需要利用基因组规模代谢模型(GSMMs),通过构建每个菌株的代谢网络,推断不同菌株GSMMs间的成对相互作用,构建代谢网络,并且结合代谢通量分析(FBA),预测群落内微生物种群之间可能存在的交叉喂养关系。目前已有一批自动化构建基因组规模代谢模型的工具开发,例如INAP2.0[47],其中包括了自动化构建基因组规模代谢模型的工具CarveMe,计算代谢互作系数的PhyloMint、SMETANA、Cobrapy等工具。然而,GSMM方法的一个局限性是GEM是化学计量模型,至少在其当前形势下,不能包含代谢物浓度或酶动力学(Vmax、Km、Kcat)。因此,结合动力学的建模可能有助于提高对微生物群落间代谢互作的了解[48]。
预测的微生物群落间潜在交叉喂养理论模型可通过共培养试验进行验证。例如,在一项研究中,通过比较单独培养红城红球菌(Rhodococcus erythropolis)和铜绿假单胞菌(Pseudomonas aeruginosa),结合代谢组与转录组分析,研究者确认了两株细菌间基于HHQ的交叉喂养机制[45]。此外,B族维生素营养缺陷株在微生物群落间广泛存在[49]。计算机预测的B族维生素通过互补的合成型/缺陷型网络驱动微生物群落之间的模块化相互作用[50]。通过体外培养试验进行了验证:共培养B3缺陷细菌与B5缺陷细菌,通过两株菌的交叉喂养保持了B3缺陷株的生物量[39],说明在这种条件下,合成群落间可以为缺陷菌株提供所需营养。
一些新的技术被运用到解析群落间的交叉喂养机制。可通过稳定同位素标记试验(SIP)结合代谢流技术解析交叉喂养的过程[51]。一项研究中利用13C代谢流分析(13C-MFA)技术,将13C标记的葡萄糖示踪剂用来培养在两个(或更多)平行培养基中生长的微生物,然后用气相色谱–质谱(GC-MS)来检测氨基酸、糖原结合葡萄糖和RNA结合核糖的同位素标记情况;之后利用Metran等13C-MFA软件对代谢流量进行估算,进而进行综合统计分析,确定拟合度并计算流量的置信区间,解析了微生物间的代谢互作过程[52]。此外,液滴微流控等技术的应用使得研究人员能够模拟复杂的生态环境,在精确控制的条件下观察微生物群落的代谢行为与资源交换[53]。
随着AI等一系列新技术的应用以及数据库的扩充,未来将进一步明晰微生物群落间的交叉喂养过程,设计并构建出更稳定的合成微生物群落。
3 红壤根际对话机制在合成菌群定殖中的作用 3.1 根际对话机制的概念与功能根际对话是指发生在植物根系与根际微生物之间的化学信号交互[54-55],这种交流在植物适应环境、提高养分吸收和抵抗非生物胁迫(如酸铝胁迫)等方面发挥着重要作用[8]。根际对话不仅包括植物根系分泌物与微生物之间的相互作用,还涉及植物与植物、微生物与微生物之间的复杂互动。根系分泌物在根际对话中扮演着关键角色,它们通过调控根际微生物群落的结构和功能,影响植物的生长和健康[56-57]。
在红壤环境中,由于其酸性较强、养分含量较低,植物面临较大的非生物胁迫,如酸铝胁迫和重金属污染。在这种条件下,植物会通过分泌有机酸、黄酮类化合物等物质来调节根际pH,提高磷的有效性,并吸引有益微生物定殖,从而增强对逆境的适应能力[58]。例如,酸铝环境诱导植物产生苹果酸等有机酸来缓解酸铝胁迫,同时为固氮螺菌属(Azospirillum)和草螺菌属(Herbaspirillum)等联合固氮菌提供可利用的碳源,促进其在根际的定殖与增殖,从而分泌生长素等促生物质,形成互惠关系[59]。吸引到根际定殖的有益微生物还可通过分泌铁载体、AHLs(酰基高丝氨酸内酯)等信号分子,调控植物的生长和防御反应[60]。此外,由于酸化环境会显著抑制拮抗性细菌的代谢活性,将增强病原真菌的优势地位与侵染能力[61],加剧植物的病害反应。例如,由于酸化土壤降低了芽孢杆菌等生物防治菌的丰度,因此炭疽菌等病原体引发的辣椒病害更加严重[62]。因此,根际环境可能会产生更多的免疫相关次生代谢物。
根际对话在植物与根际微生物之间的化学信号交换中起着核心作用,尤其在红壤等逆境环境中,根际对话通过调控根系分泌物的类型和通量,帮助植物更好地适应逆境环境,提高养分利用效率和抗逆性。
3.2 建立良性根际对话的合成菌群在红壤中,对于能够定殖在植物根际的微生物来说,需要达到以下3点:①识别根际趋化因子,快速向根际定殖,避免潜在的环境胁迫因子降低微生物活性甚至导致其死亡;②能够利用根际碳源,并进行对植物有益的生理代谢活动;③避免受到根际代谢物中微生物拮抗因子的影响,降低其丰度。因此,基于以上目标构建合成菌群对于实现菌群的有益功能十分重要。
植物根系分泌物(如有机酸、氨基酸、糖类等)是根际微生物定殖的关键信号分子。例如,苹果酸、柠檬酸等有机酸不仅可作为微生物的营养来源,还能作为信号分子引导有益微生物向根际迁移[63]。此外,根系分泌物中的挥发性有机化合物(VOCs)也能调节细菌的趋化性和竞争行为[63]。因此,合成菌群中的微生物需要具备识别并响应这些趋化因子的能力,以实现快速定殖并避免被环境胁迫因子(如低pH、高铝含量等)抑制[64]。
根际微生物需要利用根系分泌的碳源(如蔗糖、肌醇等)来维持其生长和代谢活动。例如,蔗糖可促进枯草芽孢杆菌的固氮作用,而肌醇则可作为碳源和信号分子,促进细菌的运动和定殖[63]。此外,基于“C-P”体系构建的水稻–合成菌群根际对话体系不仅有助于微生物自身的生存,还能高效溶解并向植物提供有效态磷源[8]。
根际微生物群落中存在复杂的相互作用,包括竞争、拮抗等机制。例如,一些病原微生物会通过分泌抗菌物质或干扰抗菌代谢来抑制有益微生物的定殖[65]。因此,合成菌群中的微生物需要具备抵抗这些拮抗因子的能力,例如通过竞争营养资源、分泌抗菌物质或利用植物诱导的防御机制来增强自身定殖能力。此外,构建合成菌群时,应选择具有协同作用的微生物,以提高其在根际中的稳定性和抗逆性[66]。
红壤根际对话机制不仅是植物与微生物共生关系的桥梁,更是合成菌群定殖的“化学语言”和“导航系统”。通过深入理解和利用这一机制,可以筛选适配菌株并构建高效合成菌群,推动农业可持续发展与生态环境修复。
4 总结与展望合成微生物组的设计正在成为农业可持续发展的关键技术之一,基于本土微生物的筛选与利用、微生物间交叉喂养机制解析,以及植物根际对话调控的深入理解帮助建立合成微生物组设计的基本方案(图 2)。通过挖掘适应性强、功能稳定的本土微生物资源,来构建与环境高度匹配的微生物组;而交叉喂养机制的研究则揭示了不同微生物之间协同或竞争的关系,是构建功能稳定群落的关键;同时,植物通过根系分泌物与微生物交流,对群落结构与功能产生深远影响,这种根际“语言”也是调控微生物组与植物互利共生的重要机制。
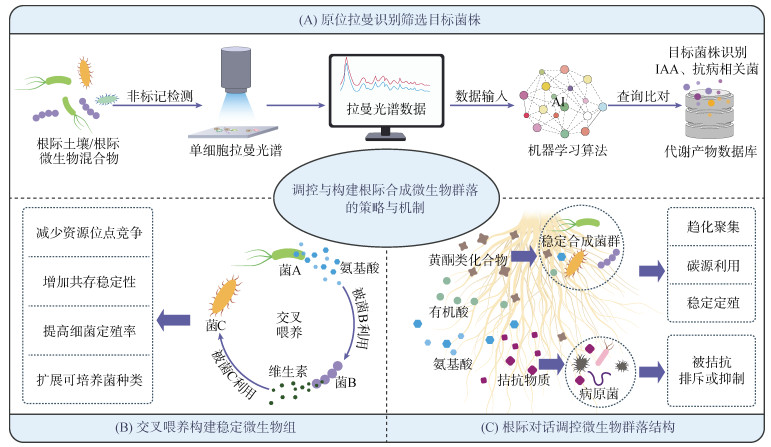
|
(A. 利用原位拉曼技术先筛后选,分离本土功能菌;B. 通过交叉代谢通路,解析菌群间的交互关系,设计稳定的合成菌群;C. 通过根际对话建立稳定定殖的合成菌群) 图 2 合成菌群的设计流程 Fig. 2 Design process of synthetic bacterial communities |
尽管研究已取得显著进展,但当前仍面临诸多挑战。例如,不同微生物之间复杂的相互作用难以准确预测,导致群落构建缺乏可控性;实验室中表现优异的微生物组在田间应用时常因环境扰动而稳定性下降;此外,植物−微生物−环境三者间的动态调控关系尚未被系统解析,制约了微生物组功能的可持续性。
未来的研究方向将更加注重智能化与系统集成。一方面,通过整合AI技术与大规模组学数据,有望提升微生物组功能的预测能力与设计效率;另一方面,构建从分子到生态系统的多尺度模型,将帮助实现微生物组在实际农业场景中的稳定运行。同时,还需推动合成微生物组技术与作物基因育种的深度融合,通过作物基因型与微生物组的精准匹配,实现植物营养获取、抗逆性与产量等多性状的协同优化,从而迈向真正智能、高效、可持续的农业生态系统。
| [1] |
中国农业科学院. 红黄壤地区综合治理与农业持续发展研究[M].
中国农业出版社, 北京, 2001
(  0) 0) |
| [2] |
黄国勤, 赵其国. 红壤生态学[J]. 生态学报, 2014, 34(18): 5173-5181 (  0) 0) |
| [3] |
de Santiago A, García-López A M, Quintero J M, et al. Effect of Trichoderma asperellum strain T34 and glucose addition on iron nutrition in cucumber grown on calcareous soils[J]. Soil Biology and Biochemistry, 2013, 57: 598-605 DOI:10.1016/j.soilbio.2012.06.020 (  0) 0) |
| [4] |
Timmermans M L, Paudel Y P, Ross A C. Investigating the biosynthesis of natural products from marine proteobacteria: A survey of molecules and strategies[J]. Marine Drugs, 2017, 15(8): 235 DOI:10.3390/md15080235 (  0) 0) |
| [5] |
Kalam S, Basu A, Ahmad I, et al. Recent understanding of soil acidobacteria and their ecological significance: A critical review[J]. Frontiers in Microbiology, 2020, 11: 580024 DOI:10.3389/fmicb.2020.580024 (  0) 0) |
| [6] |
Reji L, Zhang X N. Genome-resolved metagenomics informs the functional ecology of uncultured acidobacteria in redox oscillated Sphagnum peat[J]. mSystems, 2022, 7(5): e00055-22 (  0) 0) |
| [7] |
Ruan Z P, Chen K, Cao W M, et al. Engineering natural microbiomes toward enhanced bioremediation by microbiome modeling[J]. Nature Communications, 2024, 15: 4694 DOI:10.1038/s41467-024-49098-z (  0) 0) |
| [8] |
Liu C Y, Jiang M T, Yuan M M, et al. Root microbiota confers rice resistance to aluminium toxicity and phosphorus deficiency in acidic soils[J]. Nature Food, 2023, 4(10): 912-924 DOI:10.1038/s43016-023-00848-0 (  0) 0) |
| [9] |
Saad M M, Eida A A, Hirt H. Tailoring plant-associated microbial inoculants in agriculture: A roadmap for successful application[J]. Journal of Experimental Botany, 2020, 71(13): 3878-3901 DOI:10.1093/jxb/eraa111 (  0) 0) |
| [10] |
张瑞福, 沈其荣. 微生物肥料新型功能作用机理与根际定殖增强策略[J]. 微生物学杂志, 2024, 44(1): 1-11 (  0) 0) |
| [11] |
Berg G, Smalla K. Plant species and soil type cooperatively shape the structure and function of microbial communities in the rhizosphere[J]. FEMS Microbiology Ecology, 2009, 68(1): 1-13 DOI:10.1111/j.1574-6941.2009.00654.x (  0) 0) |
| [12] |
Yaganza E S, Rioux D, Simard M, et al. Ultrastructural alterations of Erwinia carotovora subsp. atroseptica caused by treatment with aluminum chloride and sodium metabisulfite[J]. Applied and Environmental Microbiology, 2004, 70(11): 6800-6808 DOI:10.1128/AEM.70.11.6800-6808.2004 (  0) 0) |
| [13] |
Zheng S J, Lin X Y, Yang J L, et al. The kinetics of aluminum adsorption and desorption by root cell walls of an aluminum resistant wheat (Triticum aestivum L.) cultivar[J]. Plant and Soil, 2004, 261(1): 85-90 (  0) 0) |
| [14] |
Eisenhauer N, Schulz W, Scheu S, et al. Niche dimensionality links biodiversity and invasibility of microbial communities[J]. Functional Ecology, 2013, 27(1): 282-288 DOI:10.1111/j.1365-2435.2012.02060.x (  0) 0) |
| [15] |
Yang T J, Wei Z, Friman V P, et al. Resource availability modulates biodiversity-invasion relationships by altering competitive interactions[J]. Environmental Microbiology, 2017, 19(8): 2984-2991 DOI:10.1111/1462-2920.13708 (  0) 0) |
| [16] |
Chin-A-Woeng T F, Bloemberg G V, Mulders I H, et al. Root colonization by phenazine-1-carboxamide-producing bacterium Pseudomonas chlororaphis PCL1391 is essential for biocontrol of tomato foot and root rot[J]. Molecular Plant-Microbe Interactions, 2000, 13(12): 1340-1345 DOI:10.1094/MPMI.2000.13.12.1340 (  0) 0) |
| [17] |
Kennedy G M, Min M Y, Fitzgerald J F, et al. Inactivation of the bacterial pathogens Staphylococcus pseudintermedius and Acinetobacter baumannii by butanoic acid[J]. Journal of Applied Microbiology, 2019, 126(3): 752-763 DOI:10.1111/jam.14180 (  0) 0) |
| [18] |
Honda S, Eguchi H, Okino Y, et al. The probiotic strain Clostridium butyricum TO-a produces butyrate by utilizing lactate and acetate[J]. International Journal of Molecular Sciences, 2025, 26(7): 2951 DOI:10.3390/ijms26072951 (  0) 0) |
| [19] |
Chin-A-Woeng T F C, van den Broek D, Lugtenberg B J J, et al. The Pseudomonas chlororaphis PCL1391 sigma regulator psrA represses the production of the antifungal metabolite phenazine-1-carboxamide[J]. Molecular Plant-Microbe Interactions, 2005, 18(3): 244-253 DOI:10.1094/MPMI-18-0244 (  0) 0) |
| [20] |
Zhou Y Y, Liu D H, Li F Q, et al. Superiority of native soil core microbiomes in supporting plant growth[J]. Nature Communications, 2024, 15: 6599 DOI:10.1038/s41467-024-50685-3 (  0) 0) |
| [21] |
Jiang M T, Delgado-Baquerizo M, Yuan M M, et al. Home-based microbial solution to boost crop growth in low-fertility soil[J]. New Phytologist, 2023, 239(2): 752-765 DOI:10.1111/nph.18943 (  0) 0) |
| [22] |
Benaissa A. Plant beneficial bacteria from rhizosphere as biocontrol agents of phytopathogens[J]. Vegetos, 2023, 36(4): 1566-1569 DOI:10.1007/s42535-022-00555-4 (  0) 0) |
| [23] |
Zhou Y Q, Wang H K, Sun J X, et al. Phenazines contribute to microbiome dynamics by targeting topoisomerase Ⅳ[J]. Nature Microbiology, 2025, 10(10): 2396-2411 DOI:10.1038/s41564-025-02118-0 (  0) 0) |
| [24] |
Islam T, Rabbee M F, Choi J, et al. Biosynthesis, molecular regulation, and application of bacilysin produced by Bacillus Species[J]. Metabolites, 2022, 12(5): 397 DOI:10.3390/metabo12050397 (  0) 0) |
| [25] |
Thakur M P, Geisen S. Trophic regulations of the soil microbiome[J]. Trends in Microbiology, 2019, 27(9): 771-780 DOI:10.1016/j.tim.2019.04.008 (  0) 0) |
| [26] |
Liu H J, Su Y W, Ye C, et al. Nucleotides enriched under heat stress recruit beneficial rhizomicrobes to protect plants from heat and root-rot stresses[J]. Microbiome, 2025, 13(1): 160 DOI:10.1186/s40168-025-02126-6 (  0) 0) |
| [27] |
Niu B, Paulson J N, Zheng X Q, et al. Simplified and representative bacterial community of maize roots[J]. Proceedings of the National Academy of Sciences of the United States of America, 2017, 114(12): E2450-E2459 (  0) 0) |
| [28] |
Sun X L, Xu Z H, Xie J Y, et al. Bacillus velezensis stimulates resident rhizosphere Pseudomonas stutzeri for plant health through metabolic interactions[J]. The ISME Journal, 2022, 16(3): 774-787 DOI:10.1038/s41396-021-01125-3 (  0) 0) |
| [29] |
Nichols D, Cahoon N, Trakhtenberg E M, et al. Use of ichip for high-ThroughputIn SituCultivation of "uncultivable" microbial species[J]. Applied and Environmental Microbiology, 2010, 76(8): 2445-2450 DOI:10.1128/AEM.01754-09 (  0) 0) |
| [30] |
Li H Z, Peng J J, Yang K, et al. Single-cell exploration of active phosphate-solubilizing bacteria across diverse soil matrices for sustainable phosphorus management[J]. Nature Food, 2024, 5(8): 673-683 DOI:10.1038/s43016-024-01024-8 (  0) 0) |
| [31] |
Li H Z, Yang K, Liao H, et al. Active antibiotic resistome in soils unraveled by single-cell isotope probing and targeted metagenomics[J]. PNAS, 2022, 119(40): e2201473119 DOI:10.1073/pnas.2201473119 (  0) 0) |
| [32] |
Diao Z D, Jing X Y, Hou X B, et al. Artificial intelligence-assisted automatic Raman-activated cell sorting (AI-RACS) system for mining specific functional microorganisms in the microbiome[J]. Analytical Chemistry, 2024, 96(46): 18416-18426 DOI:10.1021/acs.analchem.4c03213 (  0) 0) |
| [33] |
Cutler K J, Stringer C, Lo T W, et al. Omnipose: A high-precision morphology-independent solution for bacterial cell segmentation[J]. Nature Methods, 2022, 19(11): 1438-1448 DOI:10.1038/s41592-022-01639-4 (  0) 0) |
| [34] |
Xu T, Gong Y H, Su X L, et al. Phenome–genome profiling of single bacterial cell by Raman-activated gravity-driven encapsulation and sequencing[J]. Small, 2020, 16(30): 2001172 DOI:10.1002/smll.202001172 (  0) 0) |
| [35] |
Wang X X, Ren L H, Su Y T, et al. Raman-activated droplet sorting (RADS) for label-free high-throughput screening of microalgal single-cells[J]. Analytical Chemistry, 2017, 89(22): 12569-12577 DOI:10.1021/acs.analchem.7b03884 (  0) 0) |
| [36] |
D'Souza G, Shitut S, Preussger D, et al. Ecology and evolution of metabolic cross-feeding interactions in bacteria 1[J]. Natural Product Reports, 2018, 35(5): 455-488 DOI:10.1039/C8NP00009C (  0) 0) |
| [37] |
Goldford J E, Lu N X, Bajić D, et al. Emergent simplicity in microbial community assembly[J]. Science, 2018, 361(6401): 469-474 DOI:10.1126/science.aat1168 (  0) 0) |
| [38] |
Kost C, Patil K R, Friedman J, et al. Metabolic exchanges are ubiquitous in natural microbial communities[J]. Nature Microbiology, 2023, 8(12): 2244-2252 DOI:10.1038/s41564-023-01511-x (  0) 0) |
| [39] |
Clegg T, Gross T. Cross-feeding creates tipping points in microbiome diversity[J]. Proceedings of the National Academy of Sciences of the United States of America, 2025, 122(19): e2425603122 (  0) 0) |
| [40] |
Rappé M S, Giovannoni S J. The uncultured microbial majority[J]. Annual Review of Microbiology, 2003, 57: 369-394 DOI:10.1146/annurev.micro.57.030502.090759 (  0) 0) |
| [41] |
Pande S, Kost C. Bacterial unculturability and the formation of intercellular metabolic networks[J]. Trends in Microbiology, 2017, 25(5): 349-361 DOI:10.1016/j.tim.2017.02.015 (  0) 0) |
| [42] |
D'Souza G, Waschina S, Pande S, et al. Less is more: Selective advantages can explain the prevalent loss of biosynthetic genes in bacteria[J]. Evolution, 2014, 68(9): 2559-2570 DOI:10.1111/evo.12468 (  0) 0) |
| [43] |
Mee M T, Wang H H. Engineering ecosystems and synthetic ecologies[J]. Molecular BioSystems, 2012, 8(10): 2470-2483 DOI:10.1039/c2mb25133g (  0) 0) |
| [44] |
Lewis W H, Tahon G, Geesink P, et al. Innovations to culturing the uncultured microbial majority[J]. Nature Reviews Microbiology, 2021, 19(4): 225-240 DOI:10.1038/s41579-020-00458-8 (  0) 0) |
| [45] |
Ma Z Y, Jiang M T, Liu C Y, et al. Quinolone-mediated metabolic cross-feeding develops aluminium tolerance in soil microbial consortia[J]. Nature Communications, 2024, 15: 10148 DOI:10.1038/s41467-024-54616-0 (  0) 0) |
| [46] |
Machado D, Maistrenko O M, Andrejev S, et al. Polarization of microbial communities between competitive and cooperative metabolism[J]. Nature Ecology & Evolution, 2021, 5(2): 195-203 (  0) 0) |
| [47] |
Peng X, Feng K, Yang X S, et al. iNAP 2.0: Harnessing metabolic complementarity in microbial network analysis[J]. iMeta, 2024, 3(5): e235 DOI:10.1002/imt2.235 (  0) 0) |
| [48] |
Sen P, Orešič M. Metabolic modeling of human gut microbiota on a genome scale: An overview[J]. Metabolites, 2019, 9(2): 22 DOI:10.3390/metabo9020022 (  0) 0) |
| [49] |
Sharma V, Rodionov D A, Leyn S A, et al. B-vitamin sharing promotes stability of gut microbial communities[J]. Frontiers in Microbiology, 2019, 10: 1485 DOI:10.3389/fmicb.2019.01485 (  0) 0) |
| [50] |
Molina Ortiz J P, Read M N, McClure D D, et al. High throughput genome scale modeling predicts microbial vitamin requirements contribute to gut microbiome community structure[J]. Gut Microbes, 2022, 14(1): 2118831 DOI:10.1080/19490976.2022.2118831 (  0) 0) |
| [51] |
Abernathy M H, Yu J J, Ma F F, et al. Deciphering cyanobacterial phenotypes for fast photoautotrophic growth via isotopically nonstationary metabolic flux analysis[J]. Biotechnology for Biofuels, 2017, 10: 273 DOI:10.1186/s13068-017-0958-y (  0) 0) |
| [52] |
Long C P, Antoniewicz M R. High-resolution 13C metabolic flux analysis[J]. Nature Protocols, 2019, 14(10): 2856-2877 DOI:10.1038/s41596-019-0204-0 (  0) 0) |
| [53] |
Huys G R, Raes J. Go with the flow or solitary confinement: A look inside the single-cell toolbox for isolation of rare and uncultured microbes[J]. Current Opinion in Microbiology, 2018, 44: 1-8 DOI:10.1016/j.mib.2018.05.002 (  0) 0) |
| [54] |
Chagas F O, de Cassia Pessotti R, Caraballo-Rodríguez A M, et al. Chemical signaling involved in plant–microbe interactions[J]. Chemical Society Reviews, 2018, 47(5): 1652-1704 DOI:10.1039/C7CS00343A (  0) 0) |
| [55] |
Bonkowski M, Tarkka M, Razavi B S, et al. Spatiotemporal dynamics of maize (Zea mays L.) root growth and its potential consequences for the assembly of the rhizosphere microbiota[J]. Frontiers in Microbiology, 2021, 12: 619499 DOI:10.3389/fmicb.2021.619499 (  0) 0) |
| [56] |
Bais H P, Weir T L, Perry L G, et al. The role of root exudates in rhizosphere interactions with plants and other organisms[J]. Annual Review of Plant Biology, 2006, 57: 233-266 DOI:10.1146/annurev.arplant.57.032905.105159 (  0) 0) |
| [57] |
陆玉芳, 施卫明. 根际化学信号物质与土壤养分转化[J]. 生物技术通报, 2020, 36(9): 14-24 (  0) 0) |
| [58] |
Chen Y, Bonkowski M, Shen Y, et al. Root ethylene mediates rhizosphere microbial community reconstruction when chemically detecting cyanide produced by neighbouring plants[J]. Microbiome, 2020, 8(1): 4 DOI:10.1186/s40168-019-0775-6 (  0) 0) |
| [59] |
Bashan Y, de-Bashan L E. How the plant growth-promoting bacterium Azospirillum promotes plant growth—a critical assessment[J]. Advances in Agronomy, 2010, 108: 77-136 (  0) 0) |
| [60] |
Haldar S, Sengupta S. Plant-microbe cross-talk in the rhizosphere: Insight and biotechnological potential[J]. The Open Microbiology Journal, 2015, 9: 1-7 DOI:10.2174/1874285801509010001 (  0) 0) |
| [61] |
Rousk J, Brookes P C, Bååth E. Contrasting soil pH effects on fungal and bacterial growth suggest functional redundancy in carbon mineralization[J]. Applied and Environmental Microbiology, 2009, 75(6): 1589-1596 DOI:10.1128/AEM.02775-08 (  0) 0) |
| [62] |
Peng Y X, Zhou C, Qiu F Y, et al. Acid-resistant Bacillus velezensis effectively controls pathogenic Colletotrichum capsici and improves plant health through metabolic interactions[J]. Applied and Environmental Microbiology, 2025, 91(7): e00340-25 (  0) 0) |
| [63] |
Chen L, Liu Y P. The function of root exudates in the root colonization by beneficial soil rhizobacteria[J]. Biology, 2024, 13(2): 95 DOI:10.3390/biology13020095 (  0) 0) |
| [64] |
Cheng L, Liu Q, Nian H, et al. Microbial enhancement of plant aluminum tolerance[J]. Biology and Fertility of Soils, 2025, 61(6): 985-997 DOI:10.1007/s00374-025-01920-6 (  0) 0) |
| [65] |
Raaijmakers J M, Paulitz T C, Steinberg C, et al. The rhizosphere: A playground and battlefield for soilborne pathogens and beneficial microorganisms[J]. Plant and Soil, 2009, 321(1): 341-361 (  0) 0) |
| [66] |
李江, 靳艳玲, 赵海. 根际促生菌对植物生长的影响及其作用机制[J]. 黑龙江农业科学, 2023(10): 132-137 (  0) 0) |
2. University of Chinese Academy of Sciences, Beijing 100049, China;
3. Ecological Experimental Station of Red Soil, Chinese Academy of Sciences, Yingtan, Jiangxi 335211, China
 2025, Vol. 57
2025, Vol. 57


