2. 中国科学院土壤环境与污染修复重点实验室(南京土壤研究所),南京 210008
多环芳烃(polycyclic aromatic hydrocarbons,PAHs)是由两个或者两个以上的苯环以线形、角状或簇状方式稠合在一起的持久性有机污染物[1]。PAHs能够通过土壤、水体、大气、生物体进行长距离的迁移,具有生物毒性、生物蓄积性和半挥发性等特性,部分PAHs还有很强的致畸、致癌、致突变作用,故引起全球广泛关注[2-5]。美国和欧盟将PAHs中的16种列为优先控制污染物,1990年我国也将7种致癌性PAHs列入中国环境优先污染物黑名单。
生物修复中的植物修复技术具有修复成本低、不引起二次污染并且在修复的同时能够美化环境等特点,特别适合于大面积中低浓度PAHs污染土壤的修复[6]。由于土壤中的PAHs类污染物水溶性差,辛醇-水分配系数高,能被植物直接吸收的少[7]。因此对于PAHs污染土壤的植物修复,其修复机理主要是通过根系分泌物,选择性地刺激植物根际与污染物降解相关的微生物生长,增强与PAHs降解相关的功能菌株活性的方式来降解PAHs[8-9]。有研究显示在PAHs污染土壤中加入玉米、黑麦草、向日葵、三叶草等特定植物的根际分泌物后,土壤中PAHs类污染物的矿化速度明显加快,同时随着添加物浓度的增加,土壤微生物生物量碳、微生物呼吸商也相应增强[10-13]。根系分泌物也会影响土壤微生物群落组成,Joner和Leyval[14]研究发现,植物根系分泌物可增加专性降解菌的数量,改变种群结构及促进共代谢作用。但总体而言由于根系分泌物组成成分复杂,目前相关研究主要集中在总根系分泌物对修复的影响方面,针对根系分泌物中具体成分在修复中的作用及其对土壤微生物群落的影响研究还较少。
前期研究发现能够促进PAHs高效降解的植物其根系分泌物中大多含有大量的亚油酸,向污染土壤中添加富含亚油酸等物质的植物根系分泌物可以促进芘和苯并[a]芘等多环芳烃的降解[15]。目前为止,将亚油酸直接加入污染土壤中进行PAHs修复的研究相对很少,特别对其添加到土壤中后对土壤中细菌、真菌的群落结构及相关功能微生物的影响尚未看到报道。本研究采用向PAHs污染土壤中加入适量亚油酸钠,通过HPLC分析PAHs残余量,研究添加亚油酸钠对PAHs污染土壤的修复作用。通过高通量测序分析修复过程中土壤微生物群落变化,探究亚油酸钠修复PAHs污染土壤的机理,为其修复PAHs污染土壤的实际应用提供理论依据。
1 材料与方法 1.1 供试土壤及材料供试土壤采自南京郊区某钢铁企业附近表层土壤(0 ~ 20 cm),风干后捡出植物根系、石砾等残留物,过2 mm不锈钢筛,充分混匀,待用。使用标准法[16]测定土壤的基本理化性质:pH(H2O)8.24,有机质41.84 g/kg,有效磷23.53 mg/kg,速效钾72.66 mg/kg,阳离子交换量16.89 cmol/kg。供试亚油酸钠购买于济南鑫森源化工有限公司,工业级(> 95%)。
1.2 试验设计试验设置3个处理,3个重复,每个处理瓶装供试土壤150 g。即:①对照(CK);②施肥(F);③施肥+亚油酸钠(FSL)。试验期间,土壤的含水量保持在大约60% 的田间持水量。施肥+亚油酸钠处理中加入亚油酸钠为1 000 mg/kg。无机营养素(NH4)2SO4和K2HPO4被添加到施肥和施肥+亚油酸钠处理中,使土壤中的初始N为250 mg/kg,P为100 mg/kg。60 d后,收集土壤,一部分土壤样品在真空冷冻干燥器中冻干以测定15种PAHs(LMW-PAHs:萘(NAP),苊(ACE),芴(FLU),菲(PHE),蒽(ANT);HMW-PAHs:荧蒽(FLUA),芘(PYR),苯并[a]蒽(BaA),屈(CHR),苯并[b]荧蒽(BbF),苯并[k]荧蒽(BkF),苯并[a]芘(BaP),二苯并[a, h]蒽(DahA),茚并[1, 2, 3-cd]芘(IcdP)和苯并[g, h, i]芘(BghiP))的含量。剩余部分存放在-25℃冰箱中用于土壤微生物分析。
1.3 PAHs分析PAHs分析方法根据ISO 13877——1998标准进行。将约3.0 g冷冻干燥的土壤样品(60目)与相同重量的无水硫酸钠混合;然后将混合的样品置于索氏提取管中,并在54 ℃水浴中用70 ml二氯甲烷萃取24 h;用旋转蒸发器蒸发产物,将茄形瓶中的残余物溶于2 ml环己烷中;将等份的0.5 ml环己烷溶液过含有1 g活性硅胶(200 ~ 325目)的内径1 cm层析柱;用1:1(V/V)正己烷:二氯甲烷混合物进行洗脱;弃去最初的0.5 ml洗脱液,将洗脱液转移到带刻度的试管中并用氮吹到1 ml,加入乙腈(色谱纯)定容为2 ml;再次用氮吹到1 ml,定容为2 ml;用0.22 μm有机过滤器过滤后置于棕色样品瓶中。
使用配备有荧光检测器(RF-10AXL)的Class-VP HPLC系统(Shimadzu,Kyoto,Japan)分析15种PAHs浓度,使用的柱是C18反相柱(VP-ODS 250 × 4.6 mm I.D.,粒径5 μm),在整个过程中柱温保持在32 ℃。HPLC测定PAHs的详细色谱条件,参阅Ni等[17]。
1.4 高通量测序称取土壤样品0.5 g,利用土壤总DNA提取试剂盒FastDNA® Spin Kit for Soil提取DNA样品。使用引物515 F 5′-GTGCCAGCMGCCGCGG-3′和907 R 5′-CCGTCAATTCMTTTRAGTTT-3′对细菌16S核糖体RNA基因的V4 ~ V5区进行PCR扩增;使用引物ITS1 F 5′-CTTGGTCATTTAGAGGAAGTAA-3′和ITS2 R 5′-GCTGCGTTCTTCATCGATGC-3′扩增ITS2区域。具体的测序方案和引物设计由中国上海美吉生物医药科技有限公司提供。根据Hou等[9]的实验程序和方法进行PCR扩增、扩增子提取和测序读数优化和分析。对于每个样品标准化12 502条细菌序列和51 434条真菌序列,使用具有97% 相似性截止值的UPARSE(版本7.1)对操作分类单元(OTU)进行聚类,并使用UCHIME去除嵌合序列。使用RDP分类器算法在70% 置信度阈值下对Silva (SSU123)16S rRNA和ITS rRNA数据库进行分析。多样性指数由MOTHUR计算,主要包括Shannon、Ace、Sobs和Chao指数。通过主坐标分析(PCoA)利用Bray-Curtis距离矩阵分析处理间整体群落差异。使用LEfSe软件检测并找到丰度有显著性差异的微生物类群,采用线性判别分析(LDA)来估算物种丰度对差异效果影响的大小。
1.5 统计分析单向ANOVA分析处理之间的差异,并通过LSD测试在5% 保护水平下比较平均值。使用Excel、SPSS 21和Origin 9.1软件进行数据分析。
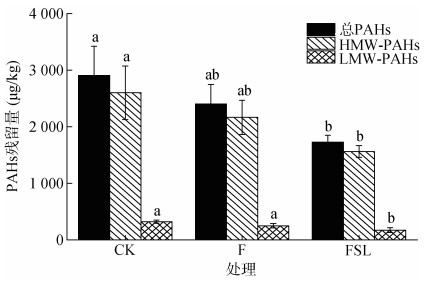
2 结果 2.1 PAHs去除60 d后测定各处理土壤样品中的PAHs残余量。对照土壤(CK)中总PAHs的浓度为2 921 μg/kg。施肥处理F中的总PAHs虽然相较于CK处理降低了17.4%,但是无论总PAHs,还是2 ~ 3环的低分子量(low molecular weight,LMW)PAHs和4 ~ 6环的高分子量(high molecular weight,HMW)PAHs含量,与CK相比均没有显著差异。FSL处理中的总PAHs、LMW-PAHs和HMW-PAHs显著低于CK处理,分别减少了40.6%、46.5% 和39.9%(图 1)。
|
(CK:对照,F:施肥,FSL:施肥+亚油酸钠;误差线表示标准偏差(n= 3),根据Duncan的多范围检验,图中小写字母不同表示处理间差异达P < 0.05显著水平;下图同) 图 1 不同处理中总PAHs、HMW-PAHs和LMW-PAHs的残留量 Fig. 1 Residual contents of PAHs in different treatments |
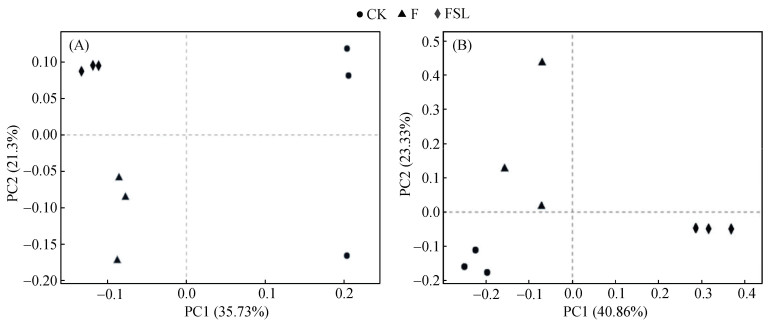
测序数据在序列优化后获得共448 209条细菌和746 578条真菌序列。每个样品细菌和真菌序列的平均数量分别为37 351条(12 502 ~ 106 068条)和62 215条序列(51 434 ~ 72 373条)。对97% 水平划分的OTU在Bray-Curtis相似性度量上进行了主坐标分析(PCoA),以揭示各处理间微生物群落的差异。如图 2所示,发现相同处理的样品聚在一起,不同处理间的样品在微生物群落结构上相互分异。细菌的PCoA图显示了CK、F和FSL处理的细菌微生物群落具有差异。真菌的PCoA图中,第一轴将仅施肥处理F和FSL处理分开,第二轴将CK和FSL处理分开。微生物群落多样性指数如表 1所示,CK处理土壤细菌群落的Shannon、Sobs和Chao指数显著高于F和FSL处理。真菌群落多样性指数差异与细菌群落类似,而且真菌群落的F、FSL处理的Ace指数也显著低于CK。
|
(每个处理3次重复,括号中的百分比表示每个排序轴的变化比例) 图 2 不同处理土壤中细菌(A)和真菌(B)群落结构的变化 Fig. 2 Community compositions of soil bacteria(A) and fungi(B) in different treatments |
|
|
表 1 不同处理的细菌和真菌基因序列的多样性指数 Table 1 Diversity indexes of gene sequences of bacteria and fungi in different treatments |
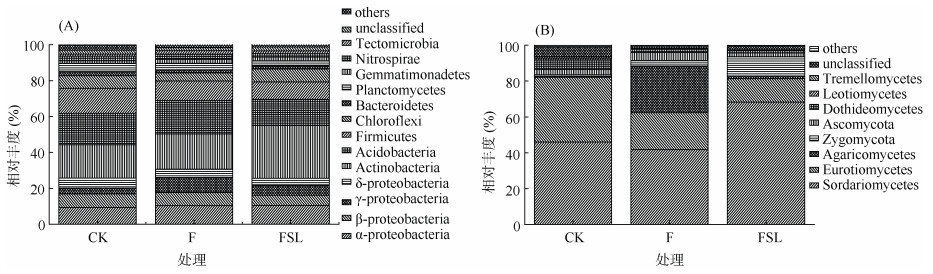
对各处理微生物群落于80% 置信度水平进行分析,扩增的细菌序列分为40个门、101个纲,真菌序列分为7个门、24个纲。不同处理的总微生物组成相似,而每个门或纲的分布各不相同(图 3),说明不同处理对不同微生物群落产生了不同程度的影响。
|
(在所有处理中相对丰度 < 1%的细菌或真菌被归类为“其他”,无法分类为任何已知组的序列被指定为“未分类”) 图 3 在门或纲水平(只有变形菌是在纲的水平分析)的细菌群落(A)以及纲水平上真菌群落(B)的比较 Fig. 3 Comparison of bacterial community(A) at phylum level or classes (only for Proteobacteria) and fungal community(B) at class level |
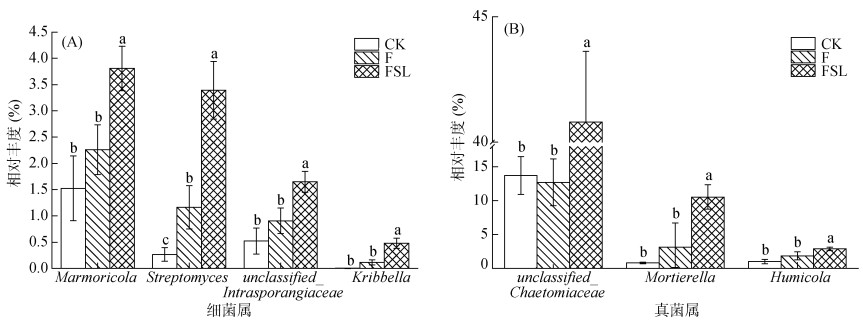
除了没有鉴定到具体门类的序列,细菌群落组成分析显示,变形菌门(Proteobacteria)、放线菌门(Actinobacteria)、酸杆菌门(Acidobacteria)、绿弯菌门(Chloroflexi)和厚壁菌门(Firmicutes)是所有处理中的5个主要门,占整体序列的82.7% 以上(图 3A)。具体来说,FSL处理中放线菌门和厚壁菌门的相对丰度与CK和F处理相比有所提高。其中,FSL处理中放线菌门的丰度占群落组成的29.3%,而在CK和F处理中,放线菌门的丰度仅占18.5% 和19.5%。放线菌门是导致FSL处理组与CK和F处理组细菌群落组成差异的主要来源。将放线菌门的微生物进一步从属水平分析发现,FSL处理中的Marmoricola(3.81%)、Streptomyces(3.39%)、unclassified_Intrasporangiaceae (1.65%)和Kribbella(0.48%)均显著高于CK和F处理组(图 4A)。
|
图 4 细菌(A)和真菌(B)群落在不同处理中相对丰度有所增加的属 Fig. 4 Genera of bacterial(A) and fungal(B) communities with increased relative abundance in different treatments |
关于真菌群落组成,除了没有鉴定到具体纲的序列,粪壳菌纲(Sordariomycetes)、散囊菌纲(Eurotiomycetes)、伞菌纲(Agaricomycetes)和接合菌纲(Zygomycota)是所有处理中的4个主要纲类,占整体序列的83.5% 以上(图 3B)。其中FSL(68.3%)中粪壳菌纲的相对丰度高于CK(46.0%)和F(41.8%)。粪壳菌纲是导致FSL处理组与CK和F处理组真菌群落组成差异的主要来源。将粪壳菌纲的微生物进一步从属水平分析发现,FSL处理中的unclassified_Chaetomiaceae(40.8%)和Humicola(2.89%)均显著高于CK和F处理组(图 4B)。接合菌纲在FSL处理中相对丰度为10.7%,而在CK和F中分别为0.83%、3.15%。另外,属水平分析发现接合菌纲的Mortierella的相对丰度显著高于CK和F处理组。
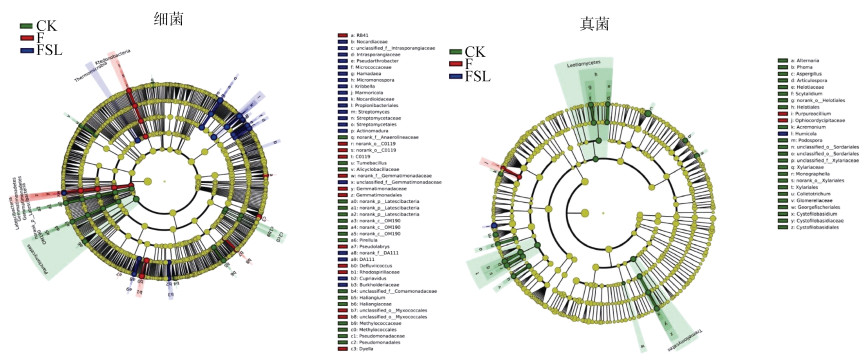
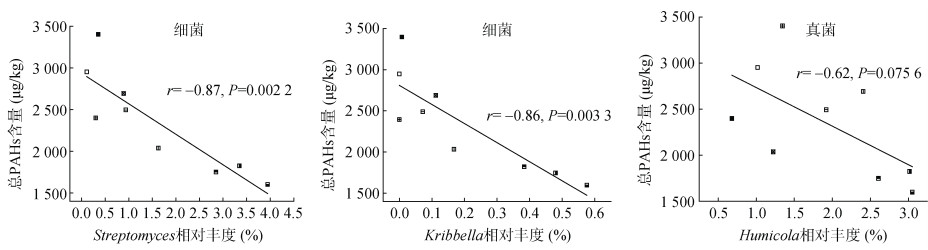
进一步对不同处理组群落组成运用基于LDA值的LEFSe分析,其中由内至外辐射的圆圈表示由门至属的分类级别。在不同分类级别上的每一个小圆圈代表该水平下的一个分类,小圆圈的直径大小与相对丰度大小呈正比。差异物种(biomarker)跟随组进行着色,红色节点表示在红色组别(F)中起到重要作用的微生物类群,蓝色节点表示在蓝色组别(FSL)中起到重要作用的微生物类群,其他圈颜色意义类同。图 5显示,Streptomyces和Kribbella 是FSL处理中的细菌标记物,Humicola 是FSL处理中的真菌标记物。将FSL处理3种差异物种的相对丰度与总PAHs含量作相关性分析,发现Streptomyces和Kribbella的相对丰度与总PAHs含量呈负相关(图 6),但是Humicola的相对丰度与总PAHs含量无相关性。
|
(根据该分类的排名最高的组,对判别分类的节点进行着色并对分支进行着色;如果分类单元在样本组之间没有显著差异,则相应的节点为黄色;选定的高丰度分类群用字母表示) 图 5 不同处理细菌群落和真菌群落组成的最小判别分析(LDA) Fig. 5 Least discriminant analysis (LDA) on effect size taxonomic cladogram to compare samples collected from different treatment |
|
图 6 Streptocymes、Kribbella 和Humicola 的相对丰度与土壤中PAHs含量的相关性分析 Fig. 6 Correlation between abundances of Streptocymes, Kribbella, Humicola and PAHs in soils |
根系分泌物通过刺激其周围微生物降解有机类污染物是修复PAHs污染土壤的主要途径之一[18]。在本研究中,施肥(F)对LMW-PAHs和HMW-PAHs的去除均没有显著影响。可以看出添加N、P等养分,虽然可为微生物提供营养,促进微生物的生长[9],但是效果有限。因为N、P元素的添加能够促进大多数的微生物增长,对PAHs降解微生物的特异选择性不强,进而使得本研究中的污染物降解效果不显著[19]。而添加亚油酸钠(FSL)处理不仅提高了LMW-PAHs降解效率,同时也提高了HMW-PAHs的降解效率(图 1)。
为了进一步了解添加亚油酸钠对污染土壤的修复机理,本研究通过高通量测序技术探究了试验过程中微生物群落组成和变化,以深入了解在修复期间PAHs降解的生物过程。基于OTU组成的PCoA分析(图 2)显示,FSL处理与CK和F之间的细菌和真菌群落明显分异。此外,细菌和真菌的多样性指数表明,FSL处理的Shannon、Sobs和Chao指数显著低于CK处理(表 1)。这个结果与我们之前的研究结果相似,即污染物的去除效率与细菌多样性无关,而与富集的特定细菌群落相关[9]。添加亚油酸钠刺激土著微生物的竞争与繁殖,其中包括有利于PAHs降解的微生物群,这种特异性选择作用,对于生物修复的成功至关重要。
微生物降解是土壤中PAHs主要的降解方式[20]。已知超过50属的细菌和真菌具有降解PAHs的能力[21-24]。有报道称亚油酸钠能够促进革兰氏阳性菌的增长[14],在本研究中,相较于对照和施肥处理,添加亚油酸钠处理组FSL中放线菌门中的Marmoricola、Streptomyces、unclassified_Intrasporangiaceae和Kribbella 等革兰氏阳性细菌显著富集(图 3,图 4)。LDA分析结果显示,Streptomyces和Kribbella是FSL处理中的细菌标记物(图 5)。有关放线菌群的研究显示,Marmoricola和Streptomyces的细菌在PAHs污染场地富集,具有降解PAHs的能力[25-26],在本研究中,Streptomyces的相对丰度与PAHs在土壤中的残余量呈负相关(图 6)。unclassified_Intrasporangiaceae未鉴别出具体属别,前人研究发现属于Intrasporangiaceae的Intrasporangium、Terrabacter spp.、Janibacter spp. 和JY11等,均与PAHs的降解相关[27-28]。Kribbella是土壤或者植物根际常见的细菌,其在堆肥及降解硝基苯酚试验中被富集,但是其具体作用尚不明确[29-30]。本研究中,Kribbella的相对丰度与PAHs在土壤中的残余量呈负相关关系(图 6),说明PAHs的降解可能与该菌相对丰度的增长有关。此外,变形菌门、酸杆菌门、绿弯菌门和厚壁菌门也是细菌群落的优势菌门,这些门类都含有PAHs降解菌群[31-33]。
真菌一般通过不同的酶系统降解HMW- PAHs[34-35]。研究表明,子囊菌门是污染环境中的主要真菌,具有转化或去除LMW- PAHs和HMW-PAHs的能力[36-37]。本研究中,Sordariomycetes菌纲(属于子囊菌门)是所有处理组中的主要类别(图 3,图 4)。添加亚油酸钠显著提高了Sordariomycetes的相对丰度,Sordariomycetes是一组真菌类群,包括相对丰度变化显著的unclassified_Chaetomiaceae和Humicola,这两种菌属均是已知的PAHs降解菌[38-39]。添加亚油酸钠处理的FSL处理中,接合菌纲的Mortierella相对丰度也显著增加,研究表明Mortierella具有降解有机污染物如石油烃类的能力[40-41]。这些结果表明,添加亚油酸钠显著改变了微生物群落结构与组成以及与PAHs降解相关菌的生长,从而提高土壤中PAHs的降解效率。
4 结论本研究通过微宇宙模拟试验对外源添加根系分泌物刺激PAHs污染土壤的微生态机理进行了探究。通过HPLC对土壤中PAHs组分进行综合分析,结果表明,添加亚油酸钠对LMW-PAHs和HMW-PAHs的降解效率均有显著提高。高通量测序数据分析显示,这一降解效率的提高,与微生物群落多样性无关,而与特定的微生物群落丰度变化相关。在添加亚油酸钠处理中,PAHs降解相关功能菌Marmoricola、Streptomyces、unclassified_Intrasporangiaceae、Kribbella、unclassified_Chaetomiaceae、Mortierella和Humicola的相对丰度显著增加,其中,Streptomyces、Kribbella和Humicola是亚油酸钠处理土壤中的主要微生物标记物,且Streptomyces、和Kribbella相对丰度与土壤中PAHs含量呈负相关。该研究结果揭示了亚油酸钠强化土壤PAHs生物降解的微生态进程,同时指导我们在进行PAHs污染土壤修复研究中应多加关注PAHs降解相关微生物菌群。
| [1] |
申国兰, 李利, 陈莎. 微生物降解石油源多环芳香烃的研究进展[J]. 土壤, 2018, 50(1): 16-27 (  0) 0) |
| [2] |
Chiari M, Ettori C, Righetti P G, et al. Oxidation of cysteine to cysteic acid in proteins by peroxyacids, as monitored by immobilized pH gradients[J]. Electrophoresis, 1991, 12(5): 376-377 DOI:10.1002/elps.1150120510 (  0) 0) |
| [3] |
周燕, 卢新卫. 西安市公园土壤多环芳烃污染特征、来源及风险评价[J]. 环境科学, 2017, 38(11): 4800-4808 (  0) 0) |
| [4] |
Adekunle A S, Oyekunle J A O, Ojo O S, et al. Determination of polycyclic aromatic hydrocarbon levels of groundwater in Ife north local government area of Osun state, Nigeria[J]. Toxicology Reports, 2017, 4: 39-48 DOI:10.1016/j.toxrep.2016.10.002 (  0) 0) |
| [5] |
倪妮, 宋洋, 王芳, 等. 多环芳烃污染土壤生物联合强化修复研究进展[J]. 土壤学报, 2016, 53(3): 561-571 (  0) 0) |
| [6] |
刘玲, 谢影, 汪承润, 等. 植物修复多环芳烃(PAHs)研究进展[J]. 江苏农业科学, 2012, 40(3): 309-312 DOI:10.3969/j.issn.1002-1302.2012.03.119 (  0) 0) |
| [7] |
Wenzel W, Adriano D, Salt D, et al. Phytoremediation: a plant-microbe-based remediation system[EB/OL]. 1999
(  0) 0) |
| [8] |
Liu W X, Hou J Y, Wang Q L, et al. Collection and analysis of root exudates of Festuca arundinacea L. and their role in facilitating the phytoremediation of petroleum- contaminated soil[J]. Plant and Soil, 2015, 389(1/2): 109-119 (  0) 0) |
| [9] |
Hou J Y, Liu W X, Wang B B, et al. PGPR enhanced phytoremediation of petroleum contaminated soil and rhizosphere microbial community response[J]. Chemosphere, 2015, 138: 592-598 DOI:10.1016/j.chemosphere.2015.07.025 (  0) 0) |
| [10] |
Yoshitomi K J, Shann J R. Corn (Zea mays L.) root exudates and their impact on 14C-pyrene mineralization[J]. Soil Biology and Biochemistry, 2001, 33(12/13): 1769-1776 (  0) 0) |
| [11] |
Xie X M, Liao M, Yang J, et al. Influence of root-exudates concentration on pyrene degradation and soil microbial characteristics in pyrene contaminated soil[J]. Chemosphere, 2012, 88(10): 1190-1195 DOI:10.1016/j.chemosphere.2012.03.068 (  0) 0) |
| [12] |
Tejeda-Agredano M C, Gallego S, Vila J, et al. Influence of the sunflower rhizosphere on the biodegradation of PAHs in soil[J]. Soil Biology and Biochemistry, 2013, 57: 830-840 DOI:10.1016/j.soilbio.2012.08.008 (  0) 0) |
| [13] |
王悦, 郭美霞, 金京华, 等. 三叶草根系分泌物对多环芳烃微生物降解及加氧酶的影响[J]. 应用生态学报, 2014, 25(11): 3145-3151 (  0) 0) |
| [14] |
Joner E J, Leyval C. Rhizosphere gradients of polycyclic aromatic hydrocarbon (PAH) dissipation in two industrial soils and the impact of arbuscular mycorrhiza[J]. Environmental Science & Technology, 2003, 37(11): 2371-2375 (  0) 0) |
| [15] |
Yi H, Crowley D E. Biostimulation of PAH degradation with plants containing high concentrations of linoleic acid[J]. Environmental Science & Technology, 2007, 41(12): 4382-4388 (  0) 0) |
| [16] |
鲁如坤. 土壤农业化学分析方法[M].
中国农业科技出版社, 北京, 2000
(  0) 0) |
| [17] |
Ni J Z, Luo Y M, Wei R, et al. Distribution patterns of polycyclic aromatic hydrocarbons among different organic carbon fractions of polluted agricultural soils[J]. Geoderma, 2008, 146(1/2): 277-282 (  0) 0) |
| [18] |
吴林坤, 林向民, 林文雄. 根系分泌物介导下植物-土壤-微生物互作关系研究进展与展望[J]. 植物生态学报, 2014, 38(3): 298-310 (  0) 0) |
| [19] |
Bell T H, Yergeau E, F Juck D, et al. Alteration of microbial community structure affects diesel biodegradation in an Arctic soil[J]. FEMS Microbiology Ecology, 2013, 85(1): 51-61 DOI:10.1111/1574-6941.12102 (  0) 0) |
| [20] |
Wang F, Su Z C, Yang H, et al. Microbial degradation of soil polycyclic aromatic hydrocarbons (PAHs) and its relations to soil bacterial population diversity[J]. Ying Yong Sheng Tai Xue Bao, 2009, 20(12): 3020-3026 (  0) 0) |
| [21] |
Seo J S, Keum Y S, Li Q. Bacterial degradation of aromatic compounds[J]. International Journal of Environmental Research and Public Health, 2009, 6(1): 278-309 DOI:10.3390/ijerph6010278 (  0) 0) |
| [22] |
Agrawal N, Verma P, Shahi S K. Degradation of polycyclic aromatic hydrocarbons (phenanthrene and pyrene) by the ligninolytic fungi Ganoderma lucidum isolated from the hardwood stump[J]. Bioresources and Bioprocessing, 2018, 5(1): 1-9 DOI:10.1186/s40643-017-0187-z (  0) 0) |
| [23] |
Marchand C, St-Arnaud M, Hogland W, et al. Petroleum biodegradation capacity of bacteria and fungi isolated from petroleum-contaminated soil[J]. International Biodeterioration & Biodegradation, 2017, 116: 48-57 (  0) 0) |
| [24] |
Reyes-César A, Absalón Á E, Fernández F J, et al. Biodegradation of a mixture of PAHs by non-ligninolytic fungal strains isolated from crude oil-contaminated soil[J]. World Journal of Microbiology and Biotechnology, 2014, 30(3): 999-1009 DOI:10.1007/s11274-013-1518-7 (  0) 0) |
| [25] |
Duran R, Bielen A, Paradžik T, et al. Exploring Actinobacteria assemblages in coastal marine sediments under contrasted Human influences in the West Istria Sea, Croatia[J]. Environmental Science and Pollution Research, 2015, 22(20): 15215-15229 DOI:10.1007/s11356-015-4240-1 (  0) 0) |
| [26] |
Zafra G, Absalón Á E, Cuevas M D C, et al. Isolation and selection of a highly tolerant microbial consortium with potential for PAH biodegradation from heavy crude oil-contaminated soils[J]. Water, Air, & Soil Pollution, 2014, 225(2): 1-18 DOI:10.1007/s11270-013-1826-4 (  0) 0) |
| [27] |
Zhang G Y, Ling J Y, Sun H B, et al. Isolation and characterization of a newly isolated polycyclic aromatic hydrocarbons-degrading Janibacter anophelis strain JY11[J]. Journal of Hazardous Materials, 2009, 172(2/3): 580-586 (  0) 0) |
| [28] |
DeBruyn J M, Mead T J, Sayler G S. Horizontal transfer of PAH catabolism genes in Mycobacterium: evidence from comparative genomics and isolated pyrene-degrading bacteria[J]. Environmental Science & Technology, 2012, 46(1): 99-106 (  0) 0) |
| [29] |
Wang K, Mao H L, Li X K. Functional characteristics and influence factors of microbial community in sewage sludge composting with inorganic bulking agent[J]. Bioresource Technology, 2018, 249: 527-535 DOI:10.1016/j.biortech.2017.10.034 (  0) 0) |
| [30] |
Min J, Wang B, Hu X K. Effect of inoculation of Burkholderia sp. strain SJ98 on bacterial community dynamics and Para -nitrophenol, 3-methyl-4-nitrophenol, and 2-chloro-4-nitrophenol degradation in soil[J]. Scientific Reports, 7(1): 1 DOI:10.1038/s41598-016-0028-x (  0) 0) |
| [31] |
Han X M, Hu H W, Shi X Z, et al. Effects of different agricultural wastes on the dissipation of PAHs and the PAH-degrading genes in a PAH-contaminated soil[J]. Chemosphere, 2017, 172: 286-293 DOI:10.1016/j.chemosphere.2017.01.012 (  0) 0) |
| [32] |
Lee D W, Lee H, Lee A H, et al. Microbial community composition and PAHs removal potential of indigenous bacteria in oil contaminated sediment of Taean Coast, Korea[J]. Environmental Pollution, 2018, 234: 503-512 DOI:10.1016/j.envpol.2017.11.097 (  0) 0) |
| [33] |
Song M K, Jiang L F, Zhang D Y, et al. Bacteria capable of degrading anthracene, phenanthrene, and fluoranthene as revealed by DNA based stable-isotope probing in a forest soil[J]. Journal of Hazardous Materials, 2016, 308: 50-57 DOI:10.1016/j.jhazmat.2016.01.009 (  0) 0) |
| [34] |
Kadri T, Rouissi T, Kaur Brar S, et al. Biodegradation of polycyclic aromatic hydrocarbons (PAHs) by fungal enzymes: a review[J]. Journal of Environmental Sciences, 2017, 51: 52-74 DOI:10.1016/j.jes.2016.08.023 (  0) 0) |
| [35] |
Memić M, Vrtačnik M, Boh B, et al. Biodegradation of PAHs by ligninolytic fungi hypoxylon fragiforme and Coniophora puteana[J]. Polycyclic Aromatic Compounds, 2020, 40(2): 206-213 DOI:10.1080/10406638.2017.1392326 (  0) 0) |
| [36] |
Marco-Urrea E, García-Romera I, Aranda E. Potential of non-ligninolytic fungi in bioremediation of chlorinated and polycyclic aromatic hydrocarbons[J]. New Biotechnology, 2015, 32(6): 620-628 DOI:10.1016/j.nbt.2015.01.005 (  0) 0) |
| [37] |
吴宇澄, 林先贵. 多环芳烃污染土壤真菌修复进展[J]. 土壤学报, 2013, 50(6): 1191-1199 (  0) 0) |
| [38] |
Saito T, Hong P, Kato K, et al. Purification and characterization of an extracellular laccase of a fungus (Family Chaetomiaceae) isolated from soil[J]. Enzyme and Microbial Technology, 2003, 33(4): 520-526 DOI:10.1016/S0141-0229(03)00158-3 (  0) 0) |
| [39] |
Cerniglia C E, Sutherland J B. Degradation of polycyclic aromatic hydrocarbons by fungi[M]//Handbook of Hydrocarbon and Lipid Microbiology. Berlin, Heidelberg: Springer Berlin Heidelberg, 2010: 2079-2110.
(  0) 0) |
| [40] |
Dave D, Ghaly A E. Remediation technologies for marine oil spills: a critical review and comparative analysis[J]. American Journal of Environmental Sciences, 2011, 7(5): 423-440 DOI:10.3844/ajessp.2011.423.440 (  0) 0) |
| [41] |
Hara E, Uchiyama H. Degradation of petroleum pollutant materials by fungi[M]//Soil Biology. Berlin, Heidelberg: Springer Berlin Heidelberg, 2012: 117-133.
(  0) 0) |
2. Key Laboratory of Soil Environment and Pollution Remediation, Institute of Soil Science, Chinese Academy of Sciences, Nanjing 210008, China
 2020, Vol. 52
2020, Vol. 52


