2. 中国科学院大学,北京 100049;
3. 合肥工业大学资源与环境工程学院,合肥 230009;
4. 南京农业大学资源与环境科学学院,南京 210095;
5. 生态环境部南京环境科学研究所,南京 210042
抗生素是一类具有消除或抑制微生物(例如细菌、真菌或原生动物)生长的天然、半合成或人工合成化合物[1-3]。根据抗生素的化学结构或作用机制可以将其分为不同的亚组,如糖类、多肽类、多烯类、大环内酯类、四环素类、嘌呤类、磺胺类等[2]。一些细菌对抗生素具有抗性,这种抗性分为内在抗性和获得性抗性[4]。这些对抗生素具有抗性的细菌被称之为抗生素抗性细菌(antibiotic resistance bacteria,ARB)。细菌的抗生素抗性机制主要包括3个类别:改变抗生素的靶位点、减少细胞对抗生素的摄取或增加外排以及酶促失活或修饰[5]。而抗生素抗性基因(antibiotic resistance genes,ARGs)往往存在于细胞的移动基因元件(mobile genetic elements,MGEs)上,如转座子、整合子或质粒[6],因此可通过水平基因转移在相同或不同种的细菌中进行传播和扩散[7]。许多研究表明,MGEs可以将ARGs向已知的人类共生细菌和病原体中转移[8-9],这种基因转移在肠道微生物组中最易发生[10]。ARGs的进化和传播主要来源于抗生素的选择性压力,尤其是人类在临床和农业环境中使用的抗生素药物[10-12]。
土壤和植物是人类和动物生活中最重要的环境组成,土壤-植物系统不但可以为人类和动物提供优良的环境条件,还是人类和动物生存的物质保障。近年来,大量的抗生素在农业生产中被用作兽药、畜禽饲料添加剂,以及水果和作物生产中的杀菌剂[1],而过量使用的抗生素会通过各种直接或间接途径进入到土壤-植物系统中[2, 13]。目前,抗生素已经成为一种新型大分子污染物,进入到土壤环境中的抗生素及其代谢产物能在土壤中维持较长时间的活性,并在土壤-植物(作物)系统中引起全新的生态环境风险。不但会造成土壤环境的化学污染,还极有可能会诱导产生抗生素抗性细菌(ARB)/抗性基因(ARGs)。土壤中的抗生素/ARB/ARGs会逐渐迁移进入植物体系,最终通过食物链直接或间接地引起人类病原细菌耐药性风险的增加,威胁人类公共健康安全。因此,深入探究抗生素/ARB/ARGs在土壤-植物系统中的迁移、转化、定殖、积累等行为及其潜在的生态毒理学机制,有利于管控抗生素抗性污染及提高食品质量。本文将从农田土壤-植物系统中抗生素的迁移转化路径、抗生素/ARB/ARGs的生态环境风险,以及抗性风险的阻控和消减技术角度,综述近年来该领域的研究进展,并提出未来研究展望。
1 抗生素/ARB/ARGs在土壤-植物系统中的迁移转化 1.1 抗生素/ARB/ARGs在土壤-植物系统中的环境行为通过各种途径进入土壤中的抗生素会发生复杂的迁移转化过程(图 1),首先大部分抗生素会被吸附,这一过程会影响抗生素在农田土壤中的环境行为和生物有效性。抗生素在土壤系统中可通过物理和化学吸附吸附在土壤颗粒上。物理吸附主要是指抗生素通过范德华力和氢键等分子间作用力被土壤颗粒物表面吸附位点或有机质相吸附[14];而化学吸附则是指抗生素的官能基团与土壤颗粒物或者有机质发生化学反应而形成络合物或螯合物的过程[15]。抗生素在土壤中的吸附能力越强,迁移性就越差,也越容易在表层土壤中富集[16-17]。此外,当土壤环境条件(pH、有机质含量、含水量等)发生变化或在生物作用下,吸附态抗生素也可重新转化成溶解态。除吸附外,一部分土壤中的抗生素会通过淋溶作用进入更深的土壤层及地下水系统,或随地表径流迁移。目前,在土壤、地表水、地下水、沉积物甚至饮用水中被发现的抗生素已经达到了数十种[18-19]。抗生素的淋溶和径流主要受降雨和土壤酸碱度等因素的影响,近年来,酸雨范围的扩大、土壤酸碱度的降低以及极端降雨事件的频发[13],可能会加剧抗生素向深层土壤/地下水的迁移。抗生素迁移转化的另一途径是被降解。降解可分为以化学降解、水解和光解等途径为主的非生物降解和以微生物降解为主的生物降解[20]。土壤中微生物种群丰富,其可通过体内转化及体外分泌相应的酶来使抗生素失活,或直接将抗生素逐步分解为H2O和CO2 [20]。抗生素在土壤系统中的环境行为由土壤条件(如土壤类型、土著菌群、CEC和pH)、抗生素本身的化学特性(如初始浓度、挥发性、溶解性和吸附性)等多种因素决定[21-23]。
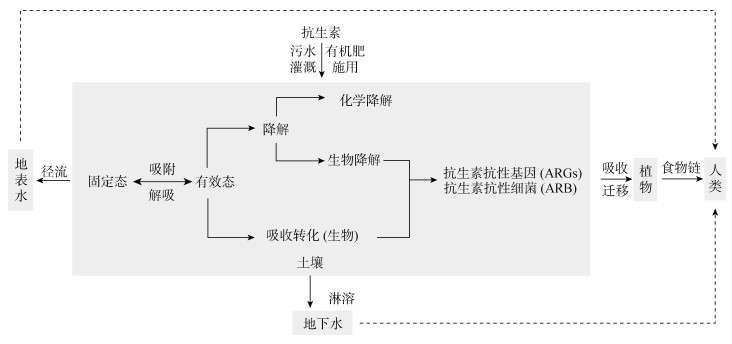
|
图 1 抗生素/ARB/ARGs在土壤-植物系统中的环境行为 Fig. 1 Environmental behavior of antibiotics /ARB/ARGs in soil-plant system |
土壤中未被降解的抗生素以及其代谢产物还会进入植物体系,被植物根系吸收以后,部分抗生素会直接积累在根系中,其余则会随蒸腾作用上行传输,沿木质部向茎、叶迁移,继而贮存在植物体的各种有机组分中[16, 24]。近年来已经在多种作物中发现了抗生素的吸收转运现象,包括甜燕麦、水稻、萝卜、胡萝卜、马铃薯、黄瓜、小米和莴苣等[25-30]。植物对抗生素的吸收主要受植物类型和抗生素种类的影响,另外,同一植物的不同部位中抗生素的累积情况也不同。Kong等[31]发现,土霉素会通过紫花苜蓿的主动吸收而在其根部大量蓄积。朱峰等[32]在小白菜培养研究中发现,比起磺胺二甲基嘧啶,土壤中的磺胺嘧啶更容易被小白菜吸收,这主要是由于磺胺二甲基嘧啶比磺胺嘧啶在结构上多两个甲基,因此更容易被土壤颗粒所吸附,迁移能力和生物有效性相对较弱,但磺胺二甲基嘧啶与磺胺嘧啶主要都分布在小白菜的茎叶部分。Kumar等[33]在植物对泰乐菌素和金霉素的吸收研究中发现,洋葱、玉米和甘蓝体内金霉素含量与有机肥施肥量呈明显的正相关,但在这3种植物体内都未检出泰乐菌素,这主要是由于土壤对泰乐菌素具有极强的吸附能力,从而使得泰乐菌素难以从土壤中迁移至植物体内[34]。Dolliver等[35]也发现莴苣、玉米和土豆均可吸收磺胺甲嘧啶,且作物体内抗生素含量与施肥量有关。Migliore等[36-37]通过研究认为C4植物吸收和积累磺胺二甲氧嘧啶的能力比C3植物更强。
土壤-作物系统中的ARB和ARGs主要来源于有机肥或污水等污染源以及土壤菌群在抗生素选择性压力下的进化。此外,土壤-植物系统中的ARGs还会通过基因传递的方式在环境中传播,导致环境中ARB和ARGs的丰度上升。ARGs传播的方式主要包括垂直基因传播(lateral gene transmission, LGT)和水平基因转移(horizontal gene transfer, HGT)[38]。LGT是指ARGs通过ARB的增殖由亲代传递给子代,只能发生在同一种属之间。HGT则是指外在环境诱导下,ARGs经整合子、转座子、噬菌体及质粒等可移动基因原件介导,以接合(conjugation)、转导(transduction)和转化(transformation)等途径在同种或不同种微生物个体间进行传播[7],在这一过程中ARGs会通过植物内生菌或叶际微生物的定殖和繁殖而向植物系统迁移。另外,同抗生素一样,土壤中的ARB和ARGs也会随地表径流和雨水淋溶而进入地表水和地下水系统[39]。
在经过人工修复处理,或在自然状态下经历足够长的时间以后,随着残留的抗生素逐渐被降解,土壤系统的选择性压力减小,微生物群落结构会逐渐发生变化,ARB的丰度也会随之下降。死亡后的ARB将会被其他微生物所分解,释放出的ARGs也会被逐渐降解。最终,土壤-植物系统中ARB/ARGs的丰度会趋于一个较低的稳定值。
1.2 抗生素/ARB/ARGs在土壤-植物系统中的生态环境风险在土壤-植物系统中残留的抗生素/ARB/ARGs不但会对环境中的微生物群落、动物和植物产生生态毒性,还极有可能会引起耐药性的传播。抗生素可通过抑制微生物细胞壁形成、干扰蛋白质的合成及代谢、影响胞浆通透性等途径来对土壤微生物造成损伤[40]。尽管抗生素都是广谱的,但大部分抗生素都有其特定的抗性谱,因此当某类抗生素进入土壤环境中,其抗性谱内的微生物被抑制后,土壤中其他微生物的生长可能会出现一定程度的增强。此外,抗生素的存在会对土壤中的微生物群落提供一定的选择压力,为了应对抗生素的毒性,部分土壤微生物的遗传特征及生理生化过程将会发生改变,进一步增强抗生素抗性的产生与进化,导致土壤抗药菌(ARB)大量繁殖,最终形成抗生素抗性类微生物菌群[9, 41]。而ARGs则会通过基因水平转移和垂直传递在同种或不同种菌群间进行传播扩散,引起耐药性的传播[7]。这些路径最终都会使得土壤微生物的群落结构发生改变,环境耐药性增强。微生物群落的生态功能多样性也会受到一定影响,进而影响土壤特性以及土壤营养元素循环、土壤呼吸作用和分解作用等土壤生态过程[42]。
对土壤动物而言,通常抗生素的生物有效性会由于土壤微生物及土壤颗粒对抗生素的吸附和稀释而降低,并且大部分土壤动物对抗生素具有较强抵抗能力,这使得抗生素对土壤动物的直接影响较小,但当环境中抗生素浓度超过一定值时,将会对土壤动物产生严重的毒性效应。Sidhu等[43]在陆地生物对环丙沙星(ciprofloxacin, CIP)和阿奇霉素(azithromycin, AZ)的生物利用度的研究中,通过为期28 d的蚯蚓亚慢性毒性试验探究了环丙沙星(0.015 ~ 1.8 mg/kg)和阿奇霉素(0.008 9 ~ 0.16 mg/kg)对蚯蚓的毒性,发现蚯蚓并未出现明显毒性反应;研究还发现蚯蚓体内环丙沙星和阿奇霉素的浓度分别为环境浓度的4倍和7倍,说明蚯蚓还具有富集环境中的抗生素的能力。Wang等[44]在环丙沙星(0 ~ 51.2 mg/kg)对蚯蚓的影响研究中发现环丙沙星暴露15 d后,蚯蚓体内的超氧化物歧化酶和抗坏血酸过氧化物酶的活性和同工酶水平均有所提高,活性氧种类过剩,从而产生氧化损伤蛋白(如羰基化蛋白)。另外,由于土壤动物多以土壤微生物为食,因此抗生素会通过改变微生物群落而对土壤动物的生长产生间接影响[41, 45]。
此外,土壤中的抗生素及其代谢产物还会通过直接或间接途径对植物产生不同程度的影响。首先,被植物根部吸收,然后转移至植物茎叶或果实中的抗生素及其代谢产物可通过影响植物酶的活性以及细胞的分裂来直接影响作物的生长发育[31, 46-48]。抗生素对植物的作用主要受抗生素自身的化学性质、浓度和作物类型等因素的影响[35, 46]。抗生素大多表现为在高浓度时抑制植物生长,而在低浓度时对植物生长具有一定的促进作用[48]。但相同浓度的抗生素积累,对某些植物而言可有效提高植物体内Ca、Mg、K、N等养分的含量从而促进植物的生长,而对其他植物的生长却不产生任何影响或具有抑制作用[46-47]。因此,明确抗生素对不同植物的剂量效应有助于准确预判土壤抗生素污染将会对作物产生何种影响。此外,抗生素对土壤微生物群落丰度、结构及功能的影响,会对土壤生态系统中的营养物质循环产生影响,最终对植物的生长发育产生间接影响[49]。此外,农田土壤中ARB和ARGs还会通过进入动植物体内或者定殖在植物表面等途径进入食物链,危害食品安全,进而增加人体正常菌群与病原菌的耐药性,危害人类健康。目前,中国、欧洲、海湾和美洲国家均有关于ARB和ARGs存在于农场、批发或零售店的蔬菜中的报道[50-54]。例如具有超广谱β-内酰胺酶(ESBL)表型的Rahnella spp.[55],以及对头孢噻肟和头孢他啶具有抗性的Pseudomonas teessidea和Rahnella aquaticus等[56]。
2 抗生素/ARB/ARGs在土壤-植物系统中的阻控消减鉴于抗生素/ARB/ARGs在土壤-植物系统中迁移转化可能带来的生态环境风险,开发有针对性的技术来控制抗生素耐药性污染和潜在的传播扩散显得非常重要。
2.1 源头管控对进入农田土壤中的抗生素进行源头管控可有效减少土壤-作物系统中抗生素/ARB/ARG的总量,是防范土壤-作物系统中抗生素及抗性污染的常用手段。具体包括以下几个方面:对农业生产中抗生素兽药及杀菌剂的使用进行规范管理,严格限制使用对象、使用量及使用条件[9];其次,避免未经处理的养殖及生活污水直接用于农田灌溉[8];另外,人畜粪便必须经堆肥发酵后再用于农业生产,并可通过改善有机肥生产过程中各种处理条件(包括改变通风方式、提高堆体的最高温度、延长高温时段等)来有效提高抗生素及ARB的处理效果,降低其在有机肥终产品中的含量[57]。
此外,科学的农业操作管理,包括合理的农耕措施和调整作物的收获时间也可有效降低土壤-作物系统中抗生素/ARB/ARGs的生态风险,这一做法被许多发达国家采用。例如加拿大的安大略省不允许从使用了生物固体或未经处理的污水污泥施肥后的土地上收获应季蔬菜;在美国,环境保护署第503部分《生物固体法规》规定,从施用B类生物肥料到收获供人类消费的地上作物之间应有14个月的间隔,而地下作物则应该有20个月的间隔;另外,世界卫生组织也提出了几项减少人类粪便危害的措施,包括施肥前处理、允许病原体死亡的作物收割延迟,以及限制在施用有机肥后的土地上种植的作物类型等[58]。相关操作应在合理的司法管辖下开展,但目前我国还没有关于农田土壤中抗生素浓度限定以及有机肥中残留抗生素/ARB/ARGs浓度限定的相关标准。
2.2 阻控对于已经进入土壤系统的抗生素/ARB/ARGs而言,通过添加阻控材料来降低抗性污染物的迁移性和生物有效性是管控生态风险常用的措施。较为常见的阻控材料主要包括:生物质炭、蛭石、粉煤灰和改性黏土矿物等吸附材料[59-60]。大部分的阻控材料都具有成本低廉、多孔结构、高比表面积和较大容积比等特点,因此对难降解有机污染物和微生物具有较强的亲和性,可通过吸附作用来降低污染物的生物有效性,以及有效防止ARB/ARGs在土壤系统中的迁移传播[14, 61]。另一方面,阻控材料还可通过改善土壤环境的理化性质来促进抗生素/ARB/ARGs的消散,降低其在农田系统中的总量,从而有效减小其生态风险。Duan等[62]发现向土壤-生菜体系中施用生物质炭后,不但抑制了土壤和生菜中氧四环素的含量,还可通过减少人类致病菌丰度来降低土壤和生菜组织中的ARGs。Vithanage等[63]采用生物质炭改性技术在添加和不添加生物质炭的处理下,研究了磺胺甲嗪在土壤中的滞留和运移,发现经生物质炭处理后,沙壤土和黏土中磺胺甲嗪的滞留率分别提高了89% 和82%,有效抑制土壤中磺胺甲嗪的迁移行为;并发现磺胺甲嗪被生物质炭吸附的主要机制包括π-π电子供体受体相互作用和静电阳离子交换。Chang等[64]在不同pH和离子强度条件下研究了伊利石(IMt-2)对四环素去除效果的影响,发现四环素在伊利石上的吸附量能达到32 mg/g,并表明吸附是通过阳离子交换而实现。这些研究也表明了,阻控材料的应用效果会受土壤环境的理化性质影响,包括pH、含水量、阳离子交换量等。将粪便转化为生物质炭来替代传统有机肥也可作为阻控抗生素/ARB/ARGs向环境中传播的有效手段。Zhou等[65]分别将猪粪堆肥产品和从猪粪中提取的生物质炭作为有机肥添加到小白菜盆栽试验的土壤中,并研究土壤中耐药菌群分布、移动遗传因子(MGEs)和细菌群落变化,发现生物质炭处理土壤的总ARGs和MGEs丰度明显低于堆肥处理土壤。Jiao等[66]发现玉米生物质炭和硫酸盐改性蛋壳两种吸附材料可促进土壤中水溶性抗生素的消散,降低土壤抗生素对马铃薯根系的毒性作用;同时,两种吸附材料均能有效降低马铃薯可食性部位中ARB和ARGs的种类和丰度。
2.3 消减除了阻控以外,加速农田土壤中抗生素/ARB/ ARGs的衰减过程也是缓解复合抗性污染风险的有效手段。抗生素污染农田土壤一般都具有覆盖面积大、复合抗性污染物叠加输入等特点。因此污染场地土壤修复技术中常用的芬顿、臭氧、氯气等化学高级氧化法技术由于处理成本相对较高、对环境扰动大等原因,往往不适合大面积农田土壤中抗生素/ARB/ARGs污染的消减处理。目前,针对此类污染土壤,研究较多的消减技术主要有农艺调控、纳米材料施用和生物修复等。
由于土壤中抗生素的降解过程受土壤理化性质、土壤微生物活动影响较大,因此改善土壤环境的理化性质可促进抗生素/ARB/ARGs在农田系统的消减[59],但每种抗生素的最佳降解条件又有所差别。Dorival- García等[67]在对6种喹诺酮类抗生素在不同条件中的降解情况进行研究时发现,氧化还原条件可影响抗生素降解,硝化条件下抗生素的去除率远高于好氧条件。但也有研究发现土壤反硝化基因(nirK、nirS和nosZ)与磺胺类ARGs(sulI和sulII)呈显著负相关,反硝化作用过程可促进土壤中磺胺嘧啶及ARGs消减;因此,可以通过向土壤中添加NO3--N来提高土壤反硝化速率,从而促进土壤中磺胺嘧啶浓度和ARGs丰度快速降低[68]。
另外,针对土壤中的ARB,还可考虑纳米颗粒的应用。作为抗微生物剂,纳米颗粒已对多种微生物显示出高效的活性抑制能力[58-59]。Dallas等[69]已经证明银纳米颗粒(AgNPs)对革兰氏阳性和革兰氏阴性细菌都具有很强的抑制能力,包括多重耐药菌株(如耐甲氧西林金黄色葡萄球菌)。除了AgNPs以外,氧化铜(CuO)、氧化锌(ZnO)和氧化钛(TiO2)等金属氧化物纳米颗粒对一系列病原体如耐甲氧西林金黄色葡萄球菌和大肠杆菌均显示出了很好的抑制能力[70-71]。但由于存在成本较高和靶向性较弱的原因,该技术在农田系统中的应用也受到了一定程度的限制。
相比于物理和化学修复而言,生物修复带来二次污染的概率较小,显得更加环境友好且成本低廉,较适合农田土壤污染的修复方式,常用的生物修复技术有植物修复、微生物修复和酶修复技术。植物修复主要是利用超积累植物吸收、植物根系固定等途径来去除土壤抗生素或降低其生物有效性,从而降低抗生素污染的生态风险[14, 72]。裴孟等[73]在针对黑麦草降解土壤中抗生素能力的研究中发现,黑麦草对四环素、恩诺沙星、金霉素、诺氟沙星等抗生素都具有较高的吸收能力。酶技术又被称为白色生物技术,介于生物和物理化学过程之间,具有降解复杂化合物的能力,且反应速率快,目前已经出现了一些酶法降解抗生素的研究[74]。Park等[75]发现,谷胱甘肽s转移酶(GSTs)可将四环素、磺胺噻唑、氨苄西林转化成对微生物无毒的成分,且转化率在60% ~ 70%。与传统的处理方法相比,酶处理的优点是可以降解高浓度或低浓度的复杂化合物,以及可在大范围的盐度、温度和pH下发挥作用[74]。微生物修复技术有生物吸附、生物降解以及噬菌体修复等。生物吸附法去除抗生素最重要的考虑因素是抗生素对微生物细胞的亲和力[61]。而生物降解主要是利用对抗生素具有抗性的微生物对抗生素进行摄入并降解[61]。Hirth等[76]通过添加磺胺甲嘧啶降解菌株来对磺胺甲嘧啶污染土壤进行修复,结果显示磺胺甲嘧啶降解菌株在46 d内成功地将土壤原生磺胺甲嘧啶的矿化率提高到57%。生物吸附和生物降解都是基于微生物的抗生素抗性,只对抗生素的去除具有一定效果,但并不能有效防止ARB以及ARGs在环境中传播可能带来的风险。
噬菌体通常被认为是可感染特定宿主细菌并能在宿主中进行复制的细菌病毒[77-78],它们是生物圈中最丰富的生物实体,估计其数量级约在1031 [79-80]。而噬菌体疗法是指将噬菌体应用于裂解宿主病原体,是一种可靶向去除ARB的技术[81-82],目前已在各个行业显示出了巨大的应用潜力[83-84]。包括在食品行业被用于减少食源性病原体[85-86],在医疗行业被用于治疗多耐药病原菌感染等[87-89]。近年来,噬菌体治疗还被用于污水处理过程中耐药病原菌的去除[89-92],以及靶向灭活土壤-植物体系中的植物致病菌[93-95]。Askora等[96]从农田土壤中分离纯化得到针对青枯雷尔氏菌的4株专性噬菌体(φRSL、φRSA、φRSM和φRSS);Frampton等[97]从猕猴桃果园土壤中筛选出了可专性灭活丁香假单胞菌的噬菌体;Ye等[69]发现采用多价噬菌体Podoviridae YSZ-1和生物质炭联合处理的方法,可高效、靶向灭活土壤-生菜体系中的耐药大肠杆菌K-12和铜绿假单胞菌PAO1,处理后,K-12和PAO1的数量从107 cfu/g分别下降到102和103 cfu/g土壤,ARGs的丰度也从108 copies/g下降到103 copies/g,该处理还能有效提高土壤-作物体系中细菌群落的多样性和稳定性。相比于其他修复技术,噬菌体治疗具有靶向特性,只会裂解目标病原细菌,可针对性消除抗生素ARB,防止ARGs的扩散,而不会对其他有益菌群造成破坏,甚至还能增加土壤微生物群落多样性。在实际修复工程中,各种修复技术通常会被联合使用,常用的有植物-微生物联合修复、植物-物理联合修复、物理-化学联合修复等技术,联合修复技术可弥补单一修复技术存在的不足,以达到更好的修复效果。
3 现有研究的不足及未来研究展望目前国内外关于土壤-植物系统中抗生素/ARB/ ARGs等抗性污染物的研究还存在一些不足:①对抗生素的迁移转化研究主要集中在物理迁移上,而对其化学转化过程中各种中间产物的毒理学研究却相对较少,这将使全面准确评估抗生素降解中间产物的生态风险的目标不易实现;②现有研究多关注抗生素本身化合物的污染毒性,而对人体健康产生直接影响的ARB和ARGs等新型抗性污染的关注相对较少;③在污染修复技术方面,现有的修复技术在成本、环境友好性、靶向性以及去除效果方面还有待提升;④我国尚未有针对土壤-植物系统中抗生素/ARB/ARGs等抗性污染物质风险管控的相关标准和导则。
基于上述研究现状,以及目前土壤环境领域的发展需求,该领域未来研究的重点如下:
1) 构建快速检测土壤-植物体系中抗生素抗性污染物的分析方法。土壤-植物体系中抗生素/ARB/ ARGs含量及丰度往往较低,且种类繁多。但现有的检测技术还存在成本较高、检测周期较长等问题,不利于检测技术的应用推广。因此,开发针对土壤-植物体系中低浓度抗生素/ARB/ARGs等抗性污染物的快速、实时定量、可视化动态分子监测与检测技术十分必要,其可有助于学者进一步探明复合抗生素抗性污染物在土壤-植物系统中的迁移转化行为。
2) 探明土壤-植物体系中ARB/ARGs与土著益生功能菌群的生态关系。土壤-作物系统中残留抗生素/ARB/ARGs等抗性污染物的自然衰减是生态风险消除的重要途径之一,其中土著菌群,例如非抗性功能益生菌群(C、N、P、S元素转化功能细菌群)或植物内生菌群的生态作用不可忽视。因此,抗生素胁迫下,土壤-植物体系中ARB与非抗性益生菌群功能多样性与稳定性的交互影响,以及生态位的演替规律也值得深入探究。
3) 研发针对土壤-植物体系中抗生素/ARB/ARGs等复合抗性污染的修复材料和技术。开发环境友好、成本低廉、便捷的高效阻控材料和新型绿色修复技术十分必要;此外,还应该关注各种阻控材料与治理技术在污染修复过程中及修复后是否存在潜在的二次生态毒理风险。
4) 加速制定针对土壤-植物系统中抗生素/ARB/ARGs等抗性污染物质风险管控的标准和导则。科学合理的管控标准和导则可为土壤-植物系统的生态风险管控提供科学依据,从源头上减少土壤-植物系统中抗生素/ARB/ARGs的输入量,更有助于各级生态环境管理部门及时有效地控制土壤-植物系统中抗生素抗性污染。
| [1] |
Silbergeld E K, Graham J, Price L B. Industrial food animal production, antimicrobial resistance, and human health[J]. Annual Review of Public Health, 2008, 29: 151-169 DOI:10.1146/annurev.publhealth.29.020907.090904 (  0) 0) |
| [2] |
Gao L H, Shi Y L, Li W H, et al. Environmental behavior and impacts of antibiotics[J]. Environmental Chemistry, 2013, 32(9): 1619-1633 (  0) 0) |
| [3] |
Kümmerer K. Antibiotics in the aquatic environment - A review - Part Ⅱ[J]. Chemosphere, 2009, 75(4): 435-441 DOI:10.1016/j.chemosphere.2008.12.006 (  0) 0) |
| [4] |
Morley P S, Apley M D, Besser T E, et al. Antimicrobial drug use in veterinary medicine[J]. Journal of Veterinary Internal Medicine, 2005, 19(4): 617-629 DOI:10.1111/j.1939-1676.2005.tb02739.x (  0) 0) |
| [5] |
Hawkey P M. The origins and molecular basis of antibiotic resistance[J]. BMJ (Clinical Research Ed.), 1998, 317(7159): 657-660 DOI:10.1136/bmj.317.7159.657 (  0) 0) |
| [6] |
Thomas C M, Nielsen K M. Mechanisms of, and barriers to, horizontal gene transfer between bacteria[J]. Nature Reviews Microbiology, 2005, 3(9): 711 DOI:10.1038/nrmicro1234 (  0) 0) |
| [7] |
Allen H K, Donato J, Wang H H, et al. Call of the wild: antibiotic resistance genes in natural environments[J]. Nature Reviews Microbiology, 2010, 8(4): 251 DOI:10.1038/nrmicro2312 (  0) 0) |
| [8] |
Witte W. Medical consequences of antibiotic use in agriculture[J]. Science, 1998, 279(5353): 996-997 DOI:10.1126/science.279.5353.996 (  0) 0) |
| [9] |
Aarestrup F M. Veterinary drug usage and antimicrobial resistance in bacteria of animal origin[J]. Basic & Clinical Pharmacology & Toxicology, 2005, 96(4): 271-281 (  0) 0) |
| [10] |
Salyers A A, Gupta A, Wang Y P. Human intestinal bacteria as reservoirs for antibiotic resistance genes[J]. Trends in Microbiology, 2004, 12(9): 412-416 DOI:10.1016/j.tim.2004.07.004 (  0) 0) |
| [11] |
Nies D H. Efflux-mediated heavy metal resistance in prokaryotes[J]. FEMS Microbiology Reviews, 2003, 27(2/3): 313-339 (  0) 0) |
| [12] |
Poole K. Efflux-mediated antimicrobial resistance[J]. Journal of Antimicrobial Chemotherapy, 2005, 56(1): 20-51 (  0) 0) |
| [13] |
Christoph K, Sven J, Ingrid R, et al. Abundance and transferability of antibiotic resistance as related to the fate of sulfadiazine in maize rhizosphere and bulk soil[J]. Fems Microbiology Ecology, 2012, 1: 125-134 (  0) 0) |
| [14] |
Li B, Zhang T. Biodegradation and adsorption of antibiotics in the activated sludge process[J]. Environmental Science & Technology, 2010, 44(9): 3468-3473 (  0) 0) |
| [15] |
鲍艳宇, 周启星, 万莹, 等. 3种四环素类抗生素在褐土上的吸附和解吸[J]. 中国环境科学, 2010, 30(10): 1383-1388 (  0) 0) |
| [16] |
Wang L P, Zhang M K. Effects of soil properties on sorption of antibiotics[J]. Chinese Journal of Soil Science, 2009, 40(2): 420-423 (  0) 0) |
| [17] |
Kulshrestha P, Giese R F, Aga D S. Investigating the molecular interactions of oxytetracycline in clay and organic matter: insights on factors affecting its mobility in soil[J]. Environmental Science & Technology, 2004, 38(15): 4097-4105 (  0) 0) |
| [18] |
Chen C Q, Li J, Chen P P, et al. Occurrence of antibiotics and antibiotic resistances in soils from wastewater irrigation areas in Beijing and Tianjin, China[J]. Environmental Pollution, 2014, 193: 94-101 DOI:10.1016/j.envpol.2014.06.005 (  0) 0) |
| [19] |
Ji X L, Shen Q H, Liu F, et al. Antibiotic resistance gene abundances associated with antibiotics and heavy metals in animal manures and agricultural soils adjacent to feedlots in Shanghai, China[J]. Journal of Hazardous Materials, 2012, 235/236: 178-185 DOI:10.1016/j.jhazmat.2012.07.040 (  0) 0) |
| [20] |
Koba O, Golovko O, Kodešová R, et al. Antibiotics degradation in soil: a case of clindamycin, trimethoprim, sulfamethoxazole and their transformation products[J]. Environmental Pollution, 2017, 220: 1251-1263 DOI:10.1016/j.envpol.2016.11.007 (  0) 0) |
| [21] |
MacKay A A, Canterbury B. Oxytetracycline sorption to organic matter by metal-bridging[J]. Journal of Environmental Quality, 2005, 34(6): 1964-1971 DOI:10.2134/jeq2005.0014 (  0) 0) |
| [22] |
Figueroa R A, Leonard A, MacKay A A. Modeling tetracycline antibiotic sorption to clays[J]. Environmental Science & Technology, 2004, 38(2): 476-483 (  0) 0) |
| [23] |
Thiele-Bruhn S. Pharmaceutical antibiotic compounds in soils - a review[J]. Journal of Plant Nutrition and Soil Science, 2003, 166(2): 145-167 DOI:10.1002/jpln.200390023 (  0) 0) |
| [24] |
王敏, 唐景春. 土壤中的抗生素污染及其生态毒性研究进展[J]. 农业环境科学学报, 2010, 29(B03): 261-266 (  0) 0) |
| [25] |
Liu F, Ying G G, Tao R, et al. Effects of six selected antibiotics on plant growth and soil microbial and enzymatic activities[J]. Environmental Pollution, 2009, 157(5): 1636-1642 DOI:10.1016/j.envpol.2008.12.021 (  0) 0) |
| [26] |
Eapen S, George L. Influence of phytohormones, carbohydrates, aminoacids, growth supplements and antibiotics on somatic embryogenesis and plant differentiation in finger millet[J]. Plant Cell, Tissue and Organ Culture, 1990, 22(2): 87-93 DOI:10.1007/BF00043683 (  0) 0) |
| [27] |
Zhang Y P, Sallach J B, Hodges L, et al. Effects of soil texture and drought stress on the uptake of antibiotics and the internalization of Salmonella in lettuce following wastewater irrigation[J]. Environmental Pollution, 2016, 208: 523-531 DOI:10.1016/j.envpol.2015.10.025 (  0) 0) |
| [28] |
Wu J, Tan W F, Yan W Z, et al. Effects of different antibiotics on genetic transformation of sweet potato[J]. Southwest China Journal of Agricultural Sciences, 2005, 18(1): 77-79 (  0) 0) |
| [29] |
Chung H S, Lee Y J, Rahman M M, et al. Uptake of the veterinary antibiotics chlortetracycline, enrofloxacin, and sulphathiazole from soil by radish[J]. Science of The Total Environment, 2017, 605/606: 322-331 DOI:10.1016/j.scitotenv.2017.06.231 (  0) 0) |
| [30] |
Grzebelus E, Skop L. Effect of β-lactam antibiotics on plant regeneration in carrot protoplast cultures[J]. In Vitro Cellular & Developmental Biology. Plant, 2014, 50(5): 568-575 (  0) 0) |
| [31] |
Kong W D, Zhu Y G, Liang Y C, et al. Uptake of oxytetracycline and its phytotoxicity to alfalfa (Medicago sativa L.)[J]. Environmental Pollution, 2007, 147(1): 187-193 DOI:10.1016/j.envpol.2006.08.016 (  0) 0) |
| [32] |
朱峰, 苏丹, 安婧, 等. 磺胺类抗生素在土壤-植物系统中的迁移特征[J]. 生态学杂志, 2017, 36(5): 1402-1407 (  0) 0) |
| [33] |
Kumar K, C Gupta S, Chander Y, et al. Antibiotic use in agriculture and its impact on the terrestrial environment[J]. 2005, 87: 1-54.
(  0) 0) |
| [34] |
Blackwell P A, Kay P, Boxall A B A. The dissipation and transport of veterinary antibiotics in a sandy loam soil[J]. Chemosphere, 2007, 67(2): 292-299 DOI:10.1016/j.chemosphere.2006.09.095 (  0) 0) |
| [35] |
Dolliver H, Kumar K, Gupta S. Sulfamethazine uptake by plants from manure-amended soil[J]. Journal of Environmental Quality, 2007, 36(4): 1224-1230 DOI:10.2134/jeq2006.0266 (  0) 0) |
| [36] |
Migliore L, Brambilla G, Cozzolino S, et al. Effect on plants of sulphadimethoxine used in intensive farming (Panicum miliaceum, Pisum sativum and Zea mays)[J]. Agriculture, Ecosystems & Environment, 1995, 52(2/3): 103-110 (  0) 0) |
| [37] |
Migliore L, Civitareale C, Brambilla G, et al. Effects of sulphadimethoxine on cosmopolitan weeds (Amaranthus retroflexus L., Plantago major L. and Rumex acetosella L.)[J]. Agriculture, Ecosystems & Environment, 1997, 65(2): 163-168 (  0) 0) |
| [38] |
Martínez J L. Antibiotics and antibiotic resistance genes in natural environments[J]. Science, 2008, 321(5887): 365-367 DOI:10.1126/science.1159483 (  0) 0) |
| [39] |
Luby E M. Fate and transport of antibiotic resistant bacteria and resistance genes in artificially drained agricultural fields receiving swine manure application[J]. Science of the Total Environment, 2014, 550: p. 1126-1133 (  0) 0) |
| [40] |
Rooklidge S J. Environmental antimicrobial contamination from terraccumulation and diffuse pollution pathways[J]. Science of The Total Environment, 2004, 325(1/2/3): 1-13 (  0) 0) |
| [41] |
Ingerslev F, Halling-Sørensen B. Biodegradability of metronidazole, olaquindox, and tylosin and formation of tylosin degradation products in aerobic soil-manure slurries[J]. Ecotoxicology and Environmental Safety, 2001, 48(3): 311-320 DOI:10.1006/eesa.2000.2026 (  0) 0) |
| [42] |
王加龙, 刘坚真, 陈杖榴, 等. 恩诺沙星残留对土壤微生物数量及群落功能多样性的影响[J]. 应用与环境生物学报, 2005, 11(1): 86-89 DOI:10.3321/j.issn:1006-687X.2005.01.021 (  0) 0) |
| [43] |
Sidhu H, O'Connor G, Ogram A, et al. Bioavailability of biosolids-borne ciprofloxacin and azithromycin to terrestrial organisms: Microbial toxicity and earthworm responses[J]. Science of The Total Environment, 2019, 650: 18-26 DOI:10.1016/j.scitotenv.2018.09.004 (  0) 0) |
| [44] |
Wang C R, Rong H, Liu H T, et al. Detoxification mechanisms, defense responses, and toxicity threshold in the earthworm Eisenia foetida exposed to ciprofloxacin- polluted soils[J]. Science of The Total Environment, 2018, 612: 442-449 DOI:10.1016/j.scitotenv.2017.08.120 (  0) 0) |
| [45] |
Baguer A J, Jensen J, Krogh P H. Effects of the antibiotics oxytetracycline and tylosin on soil fauna[J]. Chemosphere, 2000, 40(7): 751-757 DOI:10.1016/S0045-6535(99)00449-X (  0) 0) |
| [46] |
Batchelder A R. Chlortetracycline and oxytetracycline effects on plant growth and development in soil systems[J]. Journal of Environmental Quality, 1982, 11(4): 675-678 (  0) 0) |
| [47] |
Boxall A B A, Johnson P, Smith E J, et al. Uptake of veterinary medicines from soils into plants[J]. Journal of Agricultural and Food Chemistry, 2006, 54(6): 2288-2297 DOI:10.1021/jf053041t (  0) 0) |
| [48] |
Migliore L, Cozzolino S, Fiori M. Phytotoxicity to and uptake of enrofloxacin in crop plants[J]. Chemosphere, 2003, 52(7): 1233-1244 DOI:10.1016/S0045-6535(03)00272-8 (  0) 0) |
| [49] |
Vaclavik E, Halling-Sørensen B, Ingerslev F. Evaluation of manometric respiration tests to assess the effects of veterinary antibiotics in soil[J]. Chemosphere, 2004, 56(7): 667-676 DOI:10.1016/j.chemosphere.2004.02.018 (  0) 0) |
| [50] |
Boehme S, Werner G, Klare I, et al. Occurrence of antibiotic-resistant enterobacteria in agricultural foodstuffs[J]. Molecular Nutrition & Food Research, 2004, 48(7): 522-531 (  0) 0) |
| [51] |
Walia S, Rana S W, Maue D, et al. Prevalence of multiple antibiotic-resistant Gram-negative bacteria on bagged, ready-to-eat baby spinach[J]. International Journal of Environmental Health Research, 2013, 23(2): 108-118 DOI:10.1080/09603123.2012.708916 (  0) 0) |
| [52] |
D'Costa V M, McGrann K M, Hughes D W, et al. Sampling the antibiotic resistome[J]. Science, 2006, 311(5759): 374-377 DOI:10.1126/science.1120800 (  0) 0) |
| [53] |
Bhullar K, Waglechner N, Pawlowski A, et al. Antibiotic resistance is prevalent in an isolated cave microbiome[J]. PLoS One, 2012, 7(4): e34953 DOI:10.1371/journal.pone.0034953 (  0) 0) |
| [54] |
Chen Q L, An X L, Zhu Y G, et al. Application of struvite alters the antibiotic resistome in soil, rhizosphere, and phyllosphere[J]. Environmental Science & Technology, 2017, 51(14): 8149-8157 (  0) 0) |
| [55] |
Ruimy R, Brisabois A, Bernede C, et al. Organic and conventional fruits and vegetables contain equivalent counts of Gram-negative bacteria expressing resistance to antibacterial agents[J]. Environmental Microbiology, 2010, 12(3): 608-615 DOI:10.1111/j.1462-2920.2009.02100.x (  0) 0) |
| [56] |
Raphael E, Wong L K, Riley L W. Extended-spectrum Beta-lactamase gene sequences in gram-negative saprophytes on retail organic and nonorganic spinach[J]. Applied and Environmental Microbiology, 2011, 77(5): 1601-1607 DOI:10.1128/AEM.02506-10 (  0) 0) |
| [57] |
潘寻, 强志民, 贲伟伟. 高温堆肥对猪粪中多类抗生素的去除效果[J]. 生态与农村环境学报, 2013, 29(1): 64-69 DOI:10.3969/j.issn.1673-4831.2013.01.011 (  0) 0) |
| [58] |
Rahube T O, Marti R, Scott A, et al. Impact of fertilizing with raw or anaerobically digested sewage sludge on the abundance of antibiotic-resistant coliforms, antibiotic resistance genes, and pathogenic bacteria in soil and on vegetables at harvest[J]. Applied and Environmental Microbiology, 2014, 80(22): 6898-6907 DOI:10.1128/AEM.02389-14 (  0) 0) |
| [59] |
陈义群, 董元华. 土壤改良剂的研究与应用进展[J]. 生态环境学报, 2008, 17(3): 1282-1289 DOI:10.3969/j.issn.1674-5906.2008.03.078 (  0) 0) |
| [60] |
Grieco S A, Ramarao B V. Removal of TCEP from aqueous solutions by adsorption with zeolites[J]. Colloids and Surfaces A: Physicochemical and Engineering Aspects, 2013, 434: 329-338 DOI:10.1016/j.colsurfa.2013.04.042 (  0) 0) |
| [61] |
Wu C X, Spongberg A L, Witter J D. Sorption and biodegradation of selected antibiotics in biosolids[J]. Journal of Environmental Science and Health. Part A, Toxic/Hazardous Substances & Environmental Engineering, 2009, 44(5): 454-461 (  0) 0) |
| [62] |
Duan M L, Li H C, Gu J, et al. Effects of biochar on reducing the abundance of oxytetracycline, antibiotic resistance genes, and human pathogenic bacteria in soil and lettuce[J]. Environmental Pollution, 2017, 224: 787-795 DOI:10.1016/j.envpol.2017.01.021 (  0) 0) |
| [63] |
Vithanage M, Rajapaksha A U, Tang X Y, et al. Sorption and transport of sulfamethazine in agricultural soils amended with invasive-plant-derived biochar[J]. Journal of Environmental Management, 2014, 141: 95-103 DOI:10.1016/j.jenvman.2014.02.030 (  0) 0) |
| [64] |
Chang P H, Li Z H, Jean J S, et al. Adsorption of tetracycline on 2: 1 layered non-swelling clay mineral illite[J]. Applied Clay Science, 2012, 67-68: 158-163 DOI:10.1016/j.clay.2011.11.004 (  0) 0) |
| [65] |
Zhou X, Qiao M, Su J Q, et al. Turning pig manure into biochar can effectively mitigate antibiotic resistance genes as organic fertilizer[J]. Science of The Total Environment, 2019, 649: 902-908 DOI:10.1016/j.scitotenv.2018.08.368 (  0) 0) |
| [66] |
Jiao W T, Du R J, Ye M, et al. 'Agricultural Waste to Treasure' - Biochar and eggshell to impede soil antibiotics/ antibiotic resistant bacteria (genes) from accumulating in Solanum tuberosum L[J]. Environmental Pollution, 2018, 242: 2088-2095 DOI:10.1016/j.envpol.2018.06.059 (  0) 0) |
| [67] |
Dorival-García N, Zafra-Gómez A, Navalón A, et al. Removal and degradation characteristics of quinolone antibiotics in laboratory-scale activated sludge reactors under aerobic, nitrifying and anoxic conditions[J]. Journal of Environmental Management, 2013, 120: 75-83 (  0) 0) |
| [68] |
Sun M M, Ye M, Liu K, et al. Dynamic interplay between microbial denitrification and antibiotic resistance under enhanced anoxic denitrification condition in soil[J]. Environmental Pollution, 2017, 222: 583-591 DOI:10.1016/j.envpol.2016.10.015 (  0) 0) |
| [69] |
Ye M, Sun M M, Zhao Y C, et al. Targeted inactivation of antibiotic-resistant Escherichia coli and Pseudomonas aeruginosa in a soil-lettuce system by combined polyvalent bacteriophage and biochar treatment[J]. Environmental Pollution, 2018, 241: 978-987 DOI:10.1016/j.envpol.2018.04.070 (  0) 0) |
| [70] |
Tuchina E S, Tuchin V V. TiO2 nanoparticle enhanced photodynamic inhibition of pathogens[J]. Laser Physics Letters, 2010, 7(8): 607-612 DOI:10.1002/lapl.201010030 (  0) 0) |
| [71] |
Mirhosseini M, Firouzabadi F B. Antibacterial activity of zinc oxide nanoparticle suspensions on food-borne pathogens[J]. International Journal of Dairy Technology, 2013, 66(2): 291-295 DOI:10.1111/1471-0307.12015 (  0) 0) |
| [72] |
Chen N, Chen S, Chao L, et al. Study on plant-microbial remediation of antibiotic and heavy metal contaminated soil[J]. Applied Mechanics and Materials, 2014, 587-589: 816-819 DOI:10.4028/www.scientific.net/AMM.587-589.816 (  0) 0) |
| [73] |
裴孟, 梁玉婷, 易良银, 等. 黑麦草对土壤中残留抗生素的降解及其对微生物活性的影响[J]. 环境工程学报, 2017, 11(5): 3179-3186 (  0) 0) |
| [74] |
Karam J, Nicell J A. Potential applications of enzymes in waste treatment[J]. Journal of Chemical Technology & Biotechnology, 1997, 69(2): 141-153 (  0) 0) |
| [75] |
Park H, Choung Y K. Degradation of antibiotics (tetracycline, sulfathiazole, ampicillin) using enzymes of glutathion S-transferase[J]. Human and Ecological Risk Assessment: an International Journal, 2007, 13(5): 1147-1155 DOI:10.1080/10807030701506223 (  0) 0) |
| [76] |
Hirth N, Topp E, Dörfler U, et al. An effective bioremediation approach for enhanced microbial degradation of the veterinary antibiotic sulfamethazine in an agricultural soil[J]. Chemical and Biological Technologies in Agriculture, 2016, 3(1): 1-11 DOI:10.1186/s40538-015-0051-3 (  0) 0) |
| [77] |
Lyon J. Phage therapy's role in combating antibiotic- resistant pathogens[J]. JAMA, 2017, 318(18): 1746-1748 DOI:10.1001/jama.2017.12938 (  0) 0) |
| [78] |
Pires D, Melo L, Vilas Boas D, et al. Phage therapy as an alternative or complementary strategy to prevent and control biofilm-related infections[J]. Current Opinion in Microbiology, 2017, 39: 48-56 DOI:10.1016/j.mib.2017.09.004 (  0) 0) |
| [79] |
Pires D P, Vilas Boas D, Sillankorva S, et al. Phage therapy: a step forward in the treatment of pseudomonas aeruginosa infections[J]. Journal of Virology, 2015, 89(15): 7449-7456 DOI:10.1128/JVI.00385-15 (  0) 0) |
| [80] |
Yu P F, Mathieu J, Yang Y, et al. Suppression of enteric bacteria by bacteriophages: importance of phage polyvalence in the presence of soil bacteria[J]. Environmental Science & Technology, 2017, 51(9): 5270-5278 (  0) 0) |
| [81] |
Rajapaksha A U, Vithanage M, Ahmad M, et al. Enhanced sulfamethazine removal by steam-activated invasive plant-derived biochar[J]. Journal of Hazardous Materials, 2015, 290: 43-50 DOI:10.1016/j.jhazmat.2015.02.046 (  0) 0) |
| [82] |
Ye M, Sun M M, Feng Y F, et al. Effect of biochar amendment on the control of soil sulfonamides, antibiotic- resistant bacteria, and gene enrichment in lettuce tissues[J]. Journal of Hazardous Materials, 2016, 309: 219-227 DOI:10.1016/j.jhazmat.2015.10.074 (  0) 0) |
| [83] |
Keen E C, Bliskovsky V V, Malagon F, et al. Novel "superspreader" bacteriophages promote horizontal gene transfer by transformation[J]. mBio, 2017, 8(1): e02115-2116 DOI:10.1128/mbio.02115-16 (  0) 0) |
| [84] |
Khalifa L, Brosh Y, Gelman D, et al. Targeting Enterococcus faecalis biofilms with phage therapy[J]. Applied and Environmental Microbiology, 2015, 81(8): 2696-2705 DOI:10.1128/AEM.00096-15 (  0) 0) |
| [85] |
de Smet J, Hendrix H, Blasdel B G, et al. Pseudomonas predators: understanding and exploiting phage-host interactions[J]. Nature Reviews Microbiology, 2017, 15(9): 517 DOI:10.1038/nrmicro.2017.61 (  0) 0) |
| [86] |
Drulis-Kawa Z, Majkowska-Skrobek G, Maciejewska B. Bacteriophages and phage-derived proteins——application approaches[J]. Current Medicinal Chemistry, 2015, 22(14): 1757-1773 DOI:10.2174/0929867322666150209152851 (  0) 0) |
| [87] |
Squires R. Bacteriophage therapy for management of bacterial infections in veterinary practice: what was once old is new again[J]. New Zealand Veterinary Journal, 2018, 66(5): 229-235 DOI:10.1080/00480169.2018.1491348 (  0) 0) |
| [88] |
Casey E, van Sinderen D, Mahony J. In vitro characteristics of phages to guide 'real life' phage therapy suitability[J]. Viruses, 2018, 10(4): 163 DOI:10.3390/v10040163 (  0) 0) |
| [89] |
Górski A, Jończyk-Matysiak E, Łusiak-Szelachowska M, et al. Phage therapy in allergic disorders?[J]. Experimental Biology and Medicine (Maywood, N.J.), 2018, 243(6): 534-537 DOI:10.1177/1535370218755658 (  0) 0) |
| [90] |
Kazi M, Annapure U S. Bacteriophage biocontrol of foodborne pathogens[J]. Journal of Food Science and Technology, 2016, 53(3): 1355-1362 DOI:10.1007/s13197-015-1996-8 (  0) 0) |
| [91] |
Amarillas L, Cháidez-Quiroz C, Sañudo-Barajas A, et al. Complete genome sequence of a polyvalent bacteriophage, phiKP26, active on Salmonella and Escherichia coli[J]. Archives of Virology, 2013, 158(11): 2395-2398 DOI:10.1007/s00705-013-1725-4 (  0) 0) |
| [92] |
Gu J M, Liu X H, Lu R, et al. Complete genome sequence of Staphylococcus aureus bacteriophage GH15[J]. Journal of Virology, 2012, 86(16): 8914-8915 DOI:10.1128/JVI.01313-12 (  0) 0) |
| [93] |
Bhunchoth A, Phironrit N, Leksomboon C, et al. Isolation of Ralstonia solanacearum-infecting bacteriophages from tomato fields in Chiang Mai, Thailand, and their experimental use as biocontrol agents[J]. Journal of Applied Microbiology, 2015, 118(4): 1023-1033 DOI:10.1111/jam.12763 (  0) 0) |
| [94] |
Fujiwara A, Fujisawa M, Hamasaki R, et al. Biocontrol of Ralstonia solanacearum by treatment with lytic bacteriophages[J]. Applied and Environmental Microbiology, 2011, 77(12): 4155-4162 DOI:10.1128/AEM.02847-10 (  0) 0) |
| [95] |
Bae J Y, Wu J, Lee H J, et al. Biocontrol potential of a lytic bacteriophage PE204 against bacterial wilt of tomato[J]. Journal of Microbiology and Biotechnology, 2012, 22(12): 1613-1620 DOI:10.4014/jmb.1208.08072 (  0) 0) |
| [96] |
Askora A, Kawasaki T, Fujie M, et al. Insights into the diversity of φRSM phages infecting strains of the phytopathogen Ralstonia solanacearum complex: regulation and evolution[J]. Molecular Genetics and Genomics, 2014, 289(4): 589-598 DOI:10.1007/s00438-014-0835-3 (  0) 0) |
| [97] |
Frampton R A, Taylor C, Holguín Moreno A V, et al. Identification of bacteriophages for biocontrol of the kiwifruit canker phytopathogen Pseudomonas syringae pv. actinidiae[J]. Applied and Environmental Microbiology, 2014, 80(7): 2216-2228 DOI:10.1128/AEM.00062-14 (  0) 0) |
2. University of Chinese Academy of Sciences, Beijing 100049, China;
3. School of Resources and Environmental Engineering, Hefei University of Technology, Hefei 230009, China;
4. College of Resources and Environmental Sciences, Nanjing Agricultural University, Nanjing 210095, China;
5. Nanjing Institute of Environmental Science, Ministry of Environmental Protection of China, Nanjing 210042, China
 2020, Vol. 52
2020, Vol. 52


