2. 中国科学院大学, 北京 100049
干旱胁迫是陆地生态系统常见的环境胁迫之一。通常,长期干旱导致环境中的微生物细胞失水,从而直接影响其生长代谢甚至导致死亡,进而影响生态系统物质循环和能量流动。近年来,气候变化导致的极端干旱在全球范围内的频率和强度都在增加[1],微生物对环境变化十分敏感,极端气象事件势必会对微生物的组成和功能活性造成影响[2]。因此,定量分析和预测微生物对极端干旱现象增加的适应性机制是当前亟待解决的问题。
环境历史遗留效应被认为是当前环境微生物群落和功能的决定因素[3],因此,在土壤科学研究过程中,历史风干土壤标本发挥了很重要的科学价值,由于这些土壤在保存过程中处于持续脱水状态,对这些土壤标本中现存的微生物信息的探究也越来越受到人们的关注。众多证据表明,风干保存的土壤样品也可用于土壤细菌群落组成和功能的研究[2, 4-6]。在土壤科学研究中,新鲜土壤可以最直观最有力地提供土壤原位信息,包括理化性质和生物信息等,然而新鲜土壤的保存一直以来是一个困扰实际研究过程的重要问题,目前被认为最好的保存方式是尽快使用冷冻/冷藏装置保藏新鲜土壤样品,以最大化地保持样品的原貌,但是在实际操作过程中,自然风干的方法似乎是更加合适的策略。土壤标本是国家长期以来通过大型系统调查和实验需要获得并存档的土壤样品,对这些珍贵样品的调查研究为土壤现状的研究提供了强大的历史证据,很大程度上直接反映了土壤质量和性质随时间推移的变化情况,并能够为未来的样品采集与保存、土地管理等提供一定的理论支撑[7]。与自然干旱土壤相比,被作为标本保管的干燥土壤经历了持续的水枯竭,没有像田间那样的间歇性降水过程,并且保管过程中防止了植被和动物活动等环境扰动。此外,风干土壤样品可以提供来自非干旱生态系统的微生物群落的耐旱性信息。因此,风干保存的土壤标本可为研究微生物抗逆性和对持续干旱的适应性机制提供独特的样品。Zhao等[8]使用保存超过70年的风干土壤标本样品进行湿润再培养,评估经过自然风干长期保存的土壤样品中细菌组成和丰度,发现这些土壤中的许多细菌仍然存活并能够迅速复苏,证实风干土壤对细菌组成差异的分析仍存在较大价值,为土壤细菌对长期持续干旱的响应机制和抗逆性的研究提供了一定的试验依据。
然而,虽然对风干土壤中整体微生物的评估能够表征其活性和群落结构,但对于功能复杂多样的土壤微生物却不能解释其某一特定功能存在与否和活性强弱,因此,对一类特定的功能微生物的研究就显得十分重要。本课题组前期对风干土壤中氨氧化功能微生物的研究表明,风干土壤在加水培养后氨氧化微生物的数量和功能能够迅速恢复,与新鲜土壤相比群落结构并未产生较大差异[4, 9],这为研究氨氧化微生物的功能及其生态与进化机制提供了重要参考。好氧甲烷氧化菌是土壤中一种重要的功能微生物,绝大多数甲烷氧化菌以甲烷为唯一碳源和能源物质,在全球碳氮循环和温室气体减排方面具有重要的生态学意义。自1906年荷兰科学家Söhngen[10]分离出第一株好氧甲烷氧化菌Bacillus methanicum以来,科学家对甲烷氧化菌进行了广泛的分离、鉴定及生理生态研究。迄今为止,已经分离鉴定或富集出70种以上的好氧甲烷氧化菌[11]。好氧甲烷氧化菌主要来自于γ-变形菌纲(Gammaproteobacteria)、α-变形菌纲(Alphaproteobacteria)和疣微菌门(Verrucomicrobia)的Methylacidiphilum属[12-14],按照传统的分类方法γ-变形菌纲可分为Type Ⅰa、Type Ⅰb等类型,代表属包括甲基杆菌属(Methylobacter)、甲基单胞菌属(Methylomonas)、甲基八叠球菌属(Methylosarcina)、甲基球菌属(Methylococcus)和甲基暖菌属(Methylocaldum)等10余个;α-变形菌纲主要分为Type Ⅱa和Type Ⅱb两类,代表属包括甲基孢囊菌属(Methylocystis)、甲基弯菌属(Methylosinus)、Methylocella、Methylocapsa和Methyloferula等。甲烷氧化菌具有两种甲烷单加氧酶(methane monooxygenase,MMO),即颗粒性甲烷单加氧酶(pMMO)和溶解性甲烷单加氧酶(sMMO),大多数甲烷氧化菌都含有pMMO,而sMMO一般存在于Type Ⅱ甲烷氧化菌。除MMO的区别之外,不同类型的甲烷氧化菌在碳氮等核心代谢途径方面也存在较大差异[15]。甲烷氧化菌具有较强的环境适应性,广泛分布于湿地、湖泊、森林、草地等自然环境和稻田、垃圾填埋场、污水处理系统等人工生境,在环境条件温和且营养较为丰富的环境中,甲烷氧化菌群落一般由Type Ⅰ主导,其生长过程一般依赖于充足的氮素,Type Ⅱ与Type Ⅰ相比则具有更强的抗逆性,在胁迫环境中更容易生长,如低浓度甲烷、低浓度氧气以及氮素匮乏等环境。以往有关甲烷氧化菌的抗逆性研究主要集中在强酸、盐胁迫、低氧、低甲烷等方面[16-20],关于甲烷氧化菌对干旱胁迫的研究则较少。目前已知甲烷氧化菌对于短期干湿交替的胁迫存在一定的响应机制,这种机制在实验室微宇宙培养实验中得到了表征[21-22],但其对于持续干旱胁迫的抗逆性在一定程度上尚不明确,甲烷氧化菌是否会因为长期干旱的胁迫而逐渐消亡甚至消失殆尽仍有待进一步探究。
本研究从中国科学院南京土壤研究所土壤标本馆获得了6种来源不同、利用方式不同、原位气候各异的风干保存超过80年的土壤标本样品,拟通过对样品进行微宇宙培养,结合16S rRNA基因和pmoA功能基因的扩增子高通量测序和长片段克隆测序,评估培养后的土壤细菌和甲烷氧化菌的组成和种类,以通过高分辨率的细菌丰度、多样性和分子生物信息揭示长期风干保存的土壤中包含的微生物信息尤其是甲烷氧化菌是否仍然存活,甲烷氧化菌复苏再培养后的甲烷氧化能力,及对胁迫环境有较强抗性的甲烷氧化菌种类,为后续微生物环境适应性机制的研究提供一定的理论支撑和试验依据。
1 材料与方法 1.1 土壤样品概况供试土壤样本保存时间为83 ~ 87年,采样时间为1934年10月至1938年10月,包括荒地、水田、旱地、林地和牧场等不同土地利用类型,采集自5个省份6个独立采样点,分别为福建龙岩荒地(LY)、江西南昌水田(NC)、四川华阳旱地(SC)、甘肃榆中林地(YZ)、青海湟源牧场(HY)和青海都兰牧场(DL),这6例样品分属不同土壤母质、不同土壤利用类型以及不同地理气候分区,具有一定的代表性。样品具体背景信息如表 1所示。
|
|
表 1 供试土壤样品背景信息 Table 1 Background information of tested samples |
样品的保存方式为:每次收集后不久,一小部分土壤风干,保存于室温干燥条件下的贮存盒中,作为中国科学院南京土壤研究所土壤档案的一部分,每个样品的存档土壤质量为15 ~ 50 g,大多数土壤样品呈颗粒状,目视观察完全干燥。本研究获得每种土壤不超过2 g用于实验室微宇宙培养试验和分子生物学研究。由于这些样品的数量和历史数据不足,因此没有对土壤的理化性质和培养前分子生物信息进行测试。
1.2 甲烷氧化菌的复苏与富集 1.2.1 培养基配制甲烷氧化菌的富集培养采用传统的NMS(nitrate mineral salt)液体培养基[23],培养基的成分为:KNO3 1 g/L,MgSO4·7H2O 1 g/L,CaCl2 0.2 g/L,Fe-EDTA 7.6 mg/L,Na2MoO4·2H2O 0.26 mg/L,微量元素溶液1 ml/L(包括Fe、Mn、B、Zn、Cu、Co、Ni等元素),pH为6.8的磷酸盐缓冲液10 ml/L。溶液完全溶解后置于120 ℃高温灭菌锅进行20 min灭菌处理,用于后续甲烷氧化微生物的培养。
1.2.2 甲烷氧化菌的复苏由于样品经过长期风干保存,试验中首先将样品加水润湿以使微生物复苏:取1 g风干土样置于25 ml玻璃瓶中,加入400 μl无菌去离子水将样品调成湿润状态,使得土壤含水量约达到其最大持水量的60%,并用橡胶塞和铝盖密闭,随后将顶空气体置换成含20% 氧气和80% 氮气的人工空气,将体系置于28 ℃恒温培养箱中避光静置培养1周。
1.2.3 甲烷氧化菌的富集1) 富集培养。土壤复苏结束后,向玻璃瓶中加入5ml NMS培养基并调整顶空气体组分为甲烷20%、氧气30%、氮气50%,随后将其置于28 ℃恒温振荡培养箱中进行甲烷氧化菌富集培养。富集过程中采集顶空气体监测甲烷浓度变化,使用气相色谱仪(Agilent 7890A,美国)测定顶空气体中甲烷和二氧化碳的浓度,当甲烷消耗殆尽时,将培养瓶抽真空并重新充入同样组分含量的气体继续培养至甲烷消耗殆尽,此时得到的土壤悬浊液为第一代富集液。
2) 传代培养。将第一代富集液静置,待泥土层与菌液出现明显分层后,取上层菌液按照1∶200的比例,转入装有40 ml NMS培养液的120 ml玻璃瓶中继续培养,顶空气体组分含量和培养条件同上,气体浓度测定方法同上,当甲烷气体消耗殆尽时,得到第二代富集液;将第二代富集液继续按照相同的方法传代培养,直至再将甲烷消耗殆尽并得到浑浊程度较高的培养液,此时得到第三代富集液。传代培养时,所有处理均设置3个生物学重复。
3) 生物量积累量。富集过程中的生物量积累量通过细菌细胞密度比较,富集液在600nm波长处的吸光值(即OD600值)可用于表征细菌细胞密度,反映细菌的生长情况。本研究使用微量分光光度计(Thermo Scientific Nanodrop One, 美国)测定相关指标。
1.3 富集物基因组DNA的提取和高通量测序 1.3.1 富集物基因组DNA的提取取20 ml第三代富集液在3 000×g下离心15 min,收集全部微生物细胞沉淀,使用FastDNA SpinTM Kit试剂盒(MP Biomedicals,美国)按照说明提取基因组DNA。使用微量分光光度计(Thermo Scientific NanoDrop One,美国)测定DNA浓度及纯度,并使用琼脂糖凝胶电泳检测DNA完整性。DNA浓度为50.4 ~ 123.6 ng/μl,A260/A280为1.86 ~ 1.92。
1.3.2 Illumina Miseq高通量测序将上述提取得到的DNA样本进行PCR扩增,得到扩增子用于高通量测序。针对整体细菌,PCR扩增引物为16S rRNA基因通用引物515F/907R[24]。PCR反应体系50 μl,包括2 μl DNA模板(模板浓度为10 ng/μl),引物515F和907R各1 μl(引物浓度为10 μmol/L),25 μl Premix Ex TaqTM(Takara,大连),21 μl灭菌的去离子水。PCR反应程序为:95 ℃预变性3 min,30个循环(95 ℃ 30 s,55 ℃ 30 s,72 ℃ 30 s),72℃ 10 min。使用Cycle Pure Kit试剂盒(Omega,美国)纯化PCR扩增产物,并用1.2% 的琼脂糖检测PCR产物的片段长度和扩增的特异性。此后,将各样品的DNA按照等摩尔混合。针对甲烷氧化菌的pmoA基因,PCR扩增引物为A189F/mb661R[25],其余步骤同上。利用Illumina MiSeq测序平台对16S rRNA基因(PE250)和pmoA基因(PE300)进行高通量测序。
1.4 克隆测序1) 全长16S rRNA基因克隆测序。使用通用引物27F/1492R[26]对基因组DNA进行PCR扩增。PCR反应体系为50 μl,PCR反应程序为:95 ℃预变性3 min,30个循环(95 ℃ 30 s,55 ℃ 30 s,72 ℃ 90 s),72℃ 10 min。用1.2% 的琼脂糖检测PCR产物的片段长度和扩增的特异性。用纯化后的PCR产物构建克隆文库,主要包括PCR产物克隆、转化、筛选、鉴定。克隆使用的载体为pMD18-T载体。经过感受态细胞的转化、筛选、阳性克隆鉴定后,每个样本挑选5 ~ 10个阳性克隆,在ABI 3730xl (Thermo Scientific,美国)上使用引物M13+/M13-执行双向测序。
2) pmoA功能基因克隆测序。使用引物A189F/mb682R[27]对基因组DNA进行PCR扩增。其余步骤同上。
1.5 测序数据分析 1.5.1 高通量测序数据分析1) 16S rRNA序列处理。使用QIIME(version 1.9.1)软件将得到的原始测序数据进行样本拆分,去除引物、嵌合体。使用QIIME2(version 2021.4)软件对拆分后的样本进行去噪(Denoise)分析,去噪过程使用Dada 2流程构建特征表(feature-table)并统计代表序列(rep-sequences),使用Green gene数据库gg_13_8_otus中的99_otus.fasta对分类器进行朴素贝叶斯分类训练;随后,使用训练后的分类器将得到的特征表和代表序列用于分类和注释分析,最终得到物种分类信息ASV表,其中包含物种注释、序列数量等。详细步骤参考https://docs.qiime2.org/2021.4/。
2) pmoA序列处理。使用mothur(version 1.43.0)软件[28]分析测序数据。将得到的原始测序数据进行样本序列对齐、去除嵌合体、质量过滤、比对聚类(设置聚类参数时,按照文献[28]提示及学界公认将相似度水平cutoff参数设置为97%)、注释等分析,最终得到样本的OTU聚类表,其中包含物种注释、序列数量等。
1.5.2 克隆测序数据处理使用mothur(version 1.43.0)软件将得到的克隆序列按前文步骤分析后得到最终的OTU聚类表。按照OTU表挑选代表序列后,上传至NCBI(https://www.ncbi.nlm.nih.gov)网站,使用Blast模块进行核苷酸比对,下载相似物种的核酸序列,使用MEGA7软件绘制甲烷氧化菌代表序列及其近缘物种的系统发育树。
1.6 甲烷氧化速率计算甲烷氧化速率按照公式(1)计算。
| $ \upsilon = \frac{{({C_0} - {C_t}) \times {V_{\text{g}}} \times M}}{{{V_M} \times {V_{\text{l}}} \times t}} $ | (1) |
式中:υ代表甲烷氧化速率,单位为μg/(ml·h) (以CH4的量计,下同);C代表顶空气体中的甲烷浓度,单位是μl/L,C0代表起始时刻浓度,Ct代表培养结束时的浓度;t代表体系的培养时间,单位为h;Vg代表顶空气体的体积,这里按玻璃瓶容积取值;Vl代表液体培养基的体积,单位为ml;VM代表标准大气压下的气体摩尔体积,这里按22.4 L/mol取值;M代表甲烷的摩尔质量,这里取16 g/mol。
使用OriginPro 2021学习版、IBM SPSS Statistics 24和Excel 2019对试验数据进行统计分析和可视化处理。处理间显著性检验采用one-way ANOVA单因素方差分析,显著性水平为P < 0.05。
2 结果与分析 2.1 富集培养的甲烷氧化动力学特征经过短暂的复苏及富集培养后,本研究发现6例土壤档案样品均表现出强烈的甲烷氧化活性,根据培养瓶顶空甲烷浓度动态监测结果,得到了甲烷氧化速率及消耗曲线,如图 1A、1B所示。前两代富集液甲烷氧化速率较小,第一代富集液初始甲烷氧化速率仅为1.4 ~ 1.6 μg/(ml·h)(以干土质量计,则为7 ~ 8 μg/(g·h),第二代富集液最高达到了3.8 μg/(ml·h),第三代富集液则达到了4.9 ~ 7.7 μg/(ml·h),与初始速率相比增长了4倍~ 5倍,各样品第三代富集液间甲烷氧化速率存在显著差异,NC土样的甲烷氧化速率最高,达到了7.7 μg/(ml·h),而YZ土样最低,仅为4.9 μg/(ml·h)。值得注意的是,第三代富集液在80 h的培养时间内消耗了几乎全部的甲烷,其中NC、LY、DL、HY这4种土壤富集液在培养结束时瓶内甲烷浓度几乎为0,说明这些富集物不仅可以氧化高浓度甲烷,还具有氧化低浓度大气甲烷的能力。不同样品间CO2释放量及生物积累量也显著不同。其中CO2排放量最低(终浓度104 666 μl/L)的YZ土样却积累了最高的生物量,OD600达到了1.7,而CO2排放量最高(终浓度192 150 μl/L)的SC土样的OD600最低,仅为1.2(图 1C、1D)。结果表明,随着水分和营养的恢复,长期保存的风干土壤样品中的甲烷氧化菌仍可迅速恢复活性并快速生长,甚至还表现出了高亲和力的大气甲烷氧化能力,但不同土样间甲烷氧化菌的复苏能力存在显著差异。
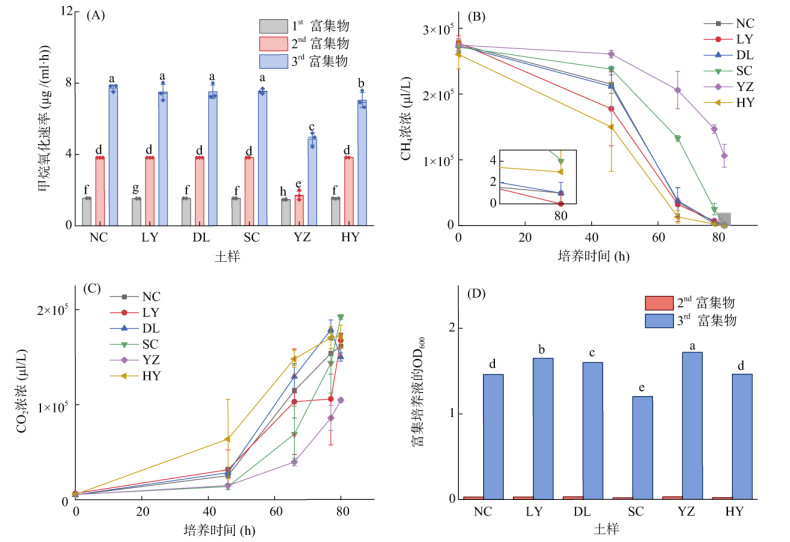
|
(柱图上方不同小写字母表示不同样品间差异在P < 0.05水平显著,图D中2nd是指第二代富集液转移到新培养液后零时刻的OD600) 图 1 甲烷氧化菌富集培养过程的生长动力学特征 Fig. 1 Characteristics of growth kinetics during resuscitation and enrichment culture |
基于16S rRNA基因的扩增子高通量测序和系统发育分析表征了不同土壤甲烷氧化富集物中细菌的物种组成特征。本次测序结果聚类分析后每个样品获得了11 171 ~ 74 886条高质量序列,在细菌门水平上(图 2A),6个土样的第三代富集物总共包含了23个细菌门类,其中DL土样仅包含9个门类,而HY土样包含了多达14个门类,优势门类为变形菌门(Proteobacteria)、厚壁菌门(Firmicutes)、酸杆菌门(Acidobacteria)和放线菌门(Actinobacteria),其中NC土样还包含较多的拟杆菌门(Bacteroidetes)。而不同土壤样品在微生物属水平表现出了更明显的分异特征(图 2B)。富集物的优势属为甲烷氧化菌Methylosinus(39% ~ 85%)、甲醇氧化菌Methylobacterium(0 ~ 31%),以及其他几种微生物类群,如共生小杆菌属(Symbiobacterium)、短波单胞菌属(Brevundimonas)、罗尔斯通氏菌属(Ralstonia)、短芽孢杆菌属(Brevibacillus)和柯恩氏菌属(Cohnella)等。不同样品间属水平的群落结构差异显著,说明不同样品中的微生物存在一定的地理分异,将Unifrac分析得到的距离矩阵进一步通过PCoA分析也发现了类似的规律(图 2C)。进一步结合Alpha多样性香农熵指数(图 2D)分析发现,各样品的多样性差别较大,香农指数分布在1.2 ~ 3.3,其中多样性最高的样品是LY土样,香农指数达到了3.1 ± 0.2。研究表明,微生物多样性会随着土壤干旱时间的延长而降低[29],而且由于富集物经历了两次传代,因此与以往新鲜样品的培养结果[30]相比,本试验结果Alpha多样性处于较低水平。各样本的Good_coverage指数均为1或0.999 9,说明本次测序的深度较为合理,并未遗漏或只遗漏了极少数低丰度物种。富集培养后,甲烷氧化菌的相对丰度在各样品的细菌总量中占比迅速增加,达到了39% ~ 85%,不同样品间存在一定差异。

|
图 2 基于扩增子测序的不同分类水平的物种组成特征(n=3) Fig. 2 Characteristics of species composition at different taxonomic levels based on amplicon sequencing |
pmoA功能基因的高通量测序结果和系统发育分析表征了不同富集物中甲烷氧化菌的群落组成特征(图 2E、2F)。将pmoA功能基因的测序结果聚类分析后,每个样品获得了44 768 ~ 118 014条高质量序列,这些富集物中的甲烷氧化菌几乎全部来自于Type Ⅱa中的Methylocystis属,仅SC土样中含有部分Methylosinus属和少量的Type Ⅱb型菌。在属水平上还检测到了极少数的Type Ⅰ甲烷氧化菌,如Methylobacter、USC-γ、JR3等。需要注意的是,根据16S rRNA基因的测序结果,富集物中优势甲烷氧化菌为Methylosinus属,而pmoA基因的测序结果表明优势甲烷氧化菌为Methylocystis属。
2.3 克隆测序分析富集物的细菌和甲烷氧化菌种类通过全长16S rRNA基因和pmoA基因的克隆测序,可进一步确认富集物中优势甲烷氧化菌的种类及系统进化关系。本研究从每个富集物样品中各获得10条全长16S rRNA基因的高质量序列,所有60条序列被分成9个OTU,经过系统发育树构建可知(图 3A),其中56条来自Alphaproteobacteria,3条来自Firmicutes,还有1条来自未培养类群Bacteria_ unclassified。属于甲烷氧化菌的有49条,其中有48条属于Methylocystis,最亲缘的序列为Methylocystis rosea strain BRCS1(序列号:CP044328.1),相似度99.86%,1条属于Methylocystaceae_unclassified,最亲缘的序列为Methylosinus sp. strain PRM1(序列号:MT355892.1),相似度99.45%。各样品中包含有6 ~ 10条属于甲烷氧化菌的序列,因此克隆测序结果证明富集物总细菌中甲烷氧化菌已超过60%(图 3C),与高通量测序的分析结果基本一致。
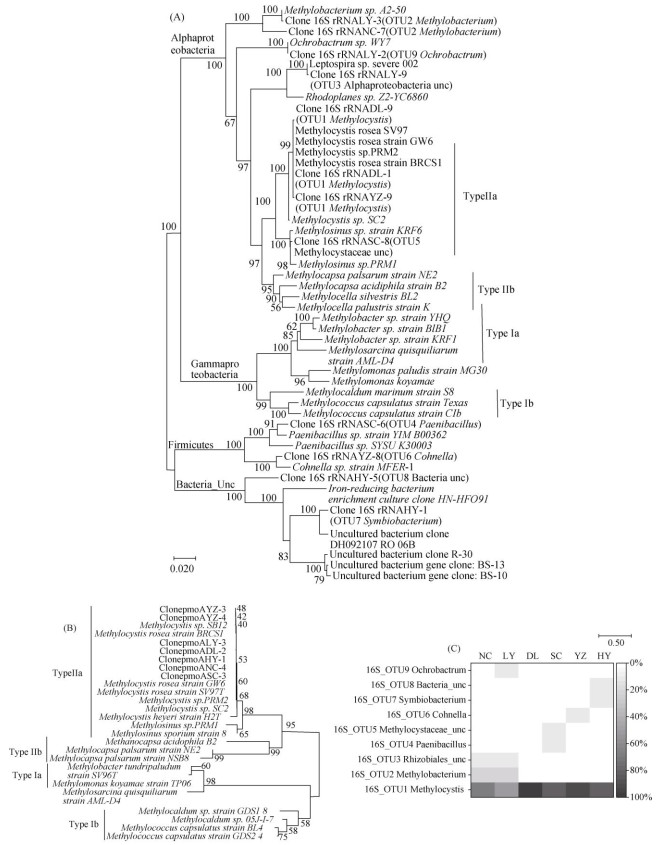
|
(图A、B中克隆序列的命名规则是Clone+基因名称+土壤样品编号) 图 3 基于16S rRNA(n=10)和pmoA(n=5)克隆测序的物种系统发育情况 Fig. 3 Phylogeny of species based on 16S rRNA and pmoA gene clone sequencing |
本研究中,pmoA基因克隆测序每个样品5条,共获得了30条克隆序列。按照相同方法进行分析(图 3B),所有样本的序列中被分类的29条全部属于Methylocystis属,1条低质量序列未被分类。系统发育分析结果表明,富集的Methylocystis和Methylocystis rosea种相似度最高,亲缘序列有Methylocystis rosea strain GW6(序列号:CP034086.1,相似度99.43%)、Methylocystis rosea strain BRCS1(序列号:CP044328.1,相似度99.05%),未检测到Methylosinus属以及其他类群的甲烷氧化菌。
3 讨论近年来,通过对极端环境中微生物的研究发现,许多微生物类群对高盐、酸碱、干旱等胁迫环境均具有较强的适应性并进化出了一些特定的应对机制[17, 31-33]。周期性的水旱交替过程广泛存在于陆地生态系统,对土壤微生物的群落结构、多样性及其功能演化具有重要影响[29, 34]。一些微生物在长期适应环境变化和气候演替中,可能占据了独特的生态位并发展了某些具有环境特异性的生理机制,并因此形成了各具特色的群落多样性。具有极高抗逆性的微生物群体对保持土壤生态系统的稳定性和功能性意义非凡。
在本研究中,因大型调查及科学研究的需要造就了土壤标本样品的一种特殊胁迫环境,而这种环境与自然界中的干湿交替不同,土样处于持续缺水的长期干旱胁迫条件且几乎不受外界干扰。这些样品有的采集自温暖湿润的亚热带沿海地区,有的则采集自高原地区,但无论是东南沿海还是西北高原,这些样品在重新加水湿润并提供营养富集培养后迅速恢复了生物活性,微生物呼吸强度和生物量急剧升高,说明在干旱气候区和非干旱气候区的土壤中均存在具有抗旱胁迫潜力的微生物物种。
本研究的结果显示,历经长期保存的风干土壤样品重新湿润并在实验室微宇宙培养过程中表现出极强的活性,结合16S rRNA基因和pmoA功能基因高通量测序的结果可知,在相同环境条件下保存的不同样品在富集培养后群落组成表现出了较大差异,说明这些不同来源的土壤样本在经历了80年的缺水胁迫后,仍然保留了一些原有的微生物特性,仍存在明显的地理分异。已有研究表明,在短期的干湿交替试验中,群落组成在加水恢复过程中不会发生明显变化[9]。因此,本试验的结果一定程度上可以反映原位的微生物群落组成情况。但是由于本研究中的土壤样品年代久远,原位理化性质和微生物信息已不可考,本文未能就胁迫前后的状态进行比较并甄别出前后的物种组成差异。
本试验富集的甲烷氧化菌具有很强的高浓度甲烷氧化活性,甚至比新鲜土壤样品培养的结果更强,而且在高浓度甲烷氧化刺激之后,具有氧化大气浓度甲烷(1.8 μl/L)的能力。本课题组之前对新鲜土壤的研究中[35]测定了10% 甲烷浓度下28 d内湖泊沉积物、盐碱地土壤和草地土壤的甲烷氧化速率,结果分别为4.55、4.68、1.96 μg/(g·h),而本文中第一代富集物在经过连续两次20% 甲烷浓度下共计50 d的培养后甲烷氧化速率就达到了7 ~ 8 μg/(g·h),显著高于新鲜土壤的培养结果。有研究表明,甲烷氧化菌在面临外界的干旱胁迫时会形成一些特殊形态并经历休眠期,如外生孢子和囊泡等[36],这也被认为是它们能够抵抗干旱胁迫的主要机制。本研究结果显示,在80多年保存的风干土壤中,甲烷氧化菌能够复苏并大量富集,Methylocystis属(Type Ⅱa)占绝对优势,除此之外也检测出了少量Type Ⅰ甲烷氧化菌(Methylobacter、USC-γ、JR3等),表明这些甲烷氧化菌可以长期(≥80年)抵抗外界的持续干旱胁迫,这可能与其独特的休眠形态有关。Ungar等[37]的研究结果表明,从自然干旱恢复的湿地中,Type Ⅰ甲烷氧化菌在干旱期间具有主导优势而非干旱期间很难检测到,Type Ⅱ甲烷氧化菌在干旱前后稳定存在,分布和相对丰度差异不大;Collet等[29]的研究结果显示,一些Type Ⅰ甲烷氧化菌对干旱胁迫具有抗逆性,同样可以从1 ~ 18年保存的风干水稻土中恢复,但会随着培养时间的延长,优势逐渐被高丰度的Type Ⅱ甲烷氧化菌替代;Ho等[38]的研究结果显示,经过人为干燥和热胁迫的样品再湿培养后,Methylosinus-Methylocystis种群规模呈指数增加,一些对该胁迫有抗性的γ-变形菌的相对丰度也有增加。这些报道与本文的结果一致。Ho等[39]分析了不同类型甲烷氧化菌的环境分异规律,Type Ⅱ甲烷氧化菌Metylocystis属能够形成休眠体,在频繁变化和胁迫环境中更易保持稳定存在,并且同时适应于高浓度和低浓度甲烷环境条件。正如本课题组之前的研究结果表明,传统Type Ⅱ甲烷氧化菌可能参与了水稻土大气甲烷氧化[16, 40]。此外,一些低丰度的USC-γ、JR3等大气甲烷氧化微生物类群也可能发挥了重要的甲烷氧化作用,其富集策略和菌种资源有待进一步挖掘。
甲烷氧化菌具有较高工业应用价值与前景,尤其是Type Ⅱ甲烷氧化菌,因独特的代谢性状和强大的抗逆性未来或可拥有更大的应用空间[41]。Hakobyan等[42]研究表明,部分Type Ⅱ甲烷氧化菌既可以以甲烷等单碳物质为碳源生长,也可以生长在乙酸盐等多碳培养基上,其中Methylocystis sp. SC2还可以在H2和CH4混合营养下生长;Microshnikov等[43]研究发现,许多Type Ⅱ甲烷氧化菌菌株具有光合作用基因且能够同化CO2;还有研究指出,部分Type Ⅱ甲烷氧化菌可用于生产多种与甲烷氧化途径相关的重要化学品,如甲醇、生物塑料聚-β-羟丁酸(PHB)等[44-45];由于两种甲烷单加氧酶的存在,部分Type Ⅱ甲烷氧化菌能够有效地减少受污染环境中的甲苯、三氯甲烷、三氯乙烯等有机污染物,可应用于环境修复[15, 18]。本文的研究思路和结果将能够为未来筛选适合生物代谢和生物催化工程的新菌株提供方向和策略,甲烷氧化菌将会成为一个重要的生物催化平台和生物工厂,对Type Ⅱ甲烷氧化菌各项功能性潜力的开发和应用将是未来绿色发展的亮点[15]。
本研究同时表明,高通量测序和克隆测序结果的规律趋于一致,但高通量测序具有分辨率高、获取信息量大的特点,与克隆测序相比能够更全面解析微生物组成和多样性,而克隆测序由于读长更长,具有能够更准确地比较和分析物种种类的优点,两者结合分析或可减少物种分类注释过程中的误差。本研究中,pmoA高通量测序结果表明Methylocystis属为优势甲烷氧化菌属,而16S rRNA高通量测序结果则显示富集物中优势甲烷氧化菌为Methylosinus属。为确认富集物中优势甲烷氧化菌的种类,本研究使用长片段16S rRNA基因和pmoA功能基因的克隆测序,进一步证明了富集物中优势甲烷氧化菌为Methylocystis属。核糖体16S rRNA扩增子高通量测序是微生物分类的金标准,然而其分类注释同样是基于序列相似性进行聚类并在现有数据库中进行比对。Methylocystis和Methylosinus同属于α-变型菌纲甲基孢囊菌科(Methylocystaceae),都是Type Ⅱ甲烷氧化菌,亲缘关系近,很可能在16S rRNA基因聚类或者注释过程中出现偏差。此外,高通量测序存在读长方面的劣势,也可能导致近缘菌之间的分类偏差。有研究者针对甲烷氧化功能基因pmoA序列整理了单独的数据库pmoA database[46],这个数据库包括了基于朴素贝叶斯分类方法的6 628个纯培养和未培养的甲烷营养生态型pmoA参考序列及其相关参考序列,对于甲烷氧化菌的分类更为详细,能够更好地区分不同分类单元,因此pmoA高通量测序和克隆测序得到了一致的结果。事实上,本研究中富集到的具有较高抗逆性的甲烷氧化菌仅有一类Type Ⅱa菌Methylocystis,由于pmoA功能基因和16S rRNA基因具有不同的分类体系,加之数据处理过程中选用的数据库差异和聚类的特异性,高通量测序结果才会出现上文提到的注释差异。本研究中,每个样品获得基于高通量测序的微生物pmoA基因序列数量44 768 ~ 118 014条,16S rRNA基因序列数量11 171 ~ 74 886条,而克隆测序仅能挑取有限的序列进行测序分析,因此克隆测序似乎很难全面地反映微生物的数量和物种组成,将高通量测序与克隆测序结合分析能够直接减少高通量数据处理过程中的系统误差,从而较为准确地判断微生物的优势物种。
4 结论1) 长期保存的风干土壤中仍存在甲烷氧化菌休眠体,经重新湿润、复苏和富集培养后,甲烷氧化菌能够恢复甲烷氧化活性并大量富集,但不同样品的复苏能力存在显著差异。
2) Type Ⅱ型甲烷氧化菌极具抗逆性。在本试验中,Type Ⅱa型Methylocystis属及其rosea亚属的甲烷氧化菌为群落中的主要优势物种,表明这一类群的微生物极具抗逆性,未来可对其应对胁迫的生理机制进行深入剖析,在单细胞水平刻画其系统发育。
3) 长期保存的风干土壤样品仍具有较高的潜在生物活性,因此风干保存的土壤样品同样具有重要的微生物学研究价值。
| [1] |
Pörtner H, Roberts D, Adams H, et al. Climate change 2022: Impacts, adaptation and vulnerability- Summary for policymakers. Contribution of Working Groups Ⅱ to the Sixth Assessment Report of the Intergovernmental Panel on Climate Change[R]. 2022: SPM-8.
(  0) 0) |
| [2] |
de Nobili M, Contin M, Brookes P C. Microbial biomass dynamics in recently air-dried and rewetted soils compared to others stored air-dry for up to 103 years[J]. Soil Biology and Biochemistry, 2006, 38(9): 2871-2881 DOI:10.1016/j.soilbio.2006.04.044 (  0) 0) |
| [3] |
Krause S M B, Meima-Franke M, Veraart A J, et al. Environmental legacy contributes to the resilience of methane consumption in a laboratory microcosm system[J]. Scientific Reports, 2018, 8: 8862 DOI:10.1038/s41598-018-27168-9 (  0) 0) |
| [4] |
Jia Z J, Hu X J, Xia W W, et al. Community shift of microbial ammonia oxidizers in air-dried rice soils after 22 years of nitrogen fertilization[J]. Biology and Fertility of Soils, 2019, 55(4): 419-424 DOI:10.1007/s00374-019-01352-z (  0) 0) |
| [5] |
Maienza A, Bååth E. Temperature effects on recovery time of bacterial growth after rewetting dry soil[J]. Microbial Ecology, 2014, 68(4): 818-821 DOI:10.1007/s00248-014-0446-x (  0) 0) |
| [6] |
Xia W W, Zhao J, Zheng Y, et al. Active soil nitrifying communities revealed by in situ transcriptomics and microcosm-based stable-isotope probing[J]. Applied and Environmental Microbiology, 2020, 86(23): e01807-e01820 (  0) 0) |
| [7] |
Dolfing J, Feng Y Z. The importance of soil archives for microbial ecology[J]. Nature Reviews Microbiology, 2015, 13(3): 1 DOI:10.1038/nrmicro3382-c1 (  0) 0) |
| [8] |
Zhao J, Chen D F, Gao W, et al. Resuscitation of soil microbiota after > 70-years of desiccation[J]. European Journal of Soil Biology, 2021, 103: 103290 DOI:10.1016/j.ejsobi.2021.103290 (  0) 0) |
| [9] |
周雪, 黄蓉, 宋歌, 等. 风干土壤中氨氧化微生物的恢复[J]. 微生物学报, 2014, 54(11): 1311-1322 (  0) 0) |
| [10] |
Söhngen N L. Welche methan als kohlenstoff nahrung and energiequelle gebrauchen[J]. Parasitenkd Infectionskr Abt, 1906, 2(15): 513-517 (  0) 0) |
| [11] |
Knief C. Diversity and habitat preferences of cultivated and uncultivated aerobic methanotrophic bacteria evaluated based on pmoA as molecular marker[J]. Frontiers in Microbiology, 2015, 6: 1346 (  0) 0) |
| [12] |
Dunfield P F, Yuryev A, Senin P, et al. Methane oxidation by an extremely acidophilic bacterium of the Phylum verrucomicrobia[J]. Nature, 2007, 450(7171): 879-882 DOI:10.1038/nature06411 (  0) 0) |
| [13] |
Qiu Q F, Conrad R, Lu Y H. Cross-feeding of methane carbon among bacteria on rice roots revealed by DNA-stable isotope probing[J]. Environmental Microbiology Reports, 2009, 1(5): 355-361 DOI:10.1111/j.1758-2229.2009.00045.x (  0) 0) |
| [14] |
贠娟莉, 王艳芬, 张洪勋. 好氧甲烷氧化菌生态学研究进展[J]. 生态学报, 2013, 33(21): 6774-6785 (  0) 0) |
| [15] |
Nguyen D T N, Lee O K, Nguyen T T, et al. Type Ⅱ methanotrophs: A promising microbial cell-factory platform for bioconversion of methane to chemicals[J]. Biotechnology Advances, 2021, 47: 107700 DOI:10.1016/j.biotechadv.2021.107700 (  0) 0) |
| [16] |
Cai Y F, Zheng Y, Bodelier P L E, et al. Conventional methanotrophs are responsible for atmospheric methane oxidation in paddy soils[J]. Nature Communications, 2016, 7: 11728 DOI:10.1038/ncomms11728 (  0) 0) |
| [17] |
Han D, Link H, Liesack W, Stams AJM. Response of Methylocystis sp. Strain SC2 to Salt Stress: Physiology, Global Transcriptome, and Amino Acid Profiles[J]. Applied and Environmental Microbiology, 2017, 83(20): e00866-00817 (  0) 0) |
| [18] |
He R, Su Y, Leewis M C, et al. Low O2 level enhances CH4-derived carbon flow into microbial communities in landfill cover soils[J]. Environmental Pollution, 2020, 258: 113676 DOI:10.1016/j.envpol.2019.113676 (  0) 0) |
| [19] |
Khadem A F, Pol A, Wieczorek A S, et al. Metabolic regulation of "Ca. Methylacidiphilum fumariolicum" SolV cells grown under different nitrogen and oxygen limitations[J]. Frontiers in Microbiology, 2012, 3: 266 (  0) 0) |
| [20] |
Stone K, Hilliard M, Badr K, et al. Comparative study of oxygen-limited and methane-limited growth phenotypes of Methylomicrobium buryatense 5GB1[J]. Biochemical Engineering Journal, 2020, 161: 107707 DOI:10.1016/j.bej.2020.107707 (  0) 0) |
| [21] |
Liu F Q, Zhang Y P, Liang H, et al. Resilience of methane cycle and microbial functional genes to drought and flood in an alkaline wetland: A metagenomic analysis[J]. Chemosphere, 2021, 265: 129034 DOI:10.1016/j.chemosphere.2020.129034 (  0) 0) |
| [22] |
刘蓓, Elberling B, 贾仲君. 不同水分条件下格陵兰岛冻土活性甲烷氧化菌群落分异规律[J]. 土壤, 2020, 52(1): 90-96 (  0) 0) |
| [23] |
Whittenbury R, Phillips K C, Wilkinson J F. Enrichment, isolation and some properties of methane-utilizing bacteria[J]. Journal of General Microbiology, 1970, 61(2): 205-218 DOI:10.1099/00221287-61-2-205 (  0) 0) |
| [24] |
Christner B C, Mosley-Thompson E, Thompson L G, et al. Isolation of bacteria and 16S rDNAs from Lake Vostok accretion ice[J]. Environmental Microbiology, 2001, 3(9): 570-577 DOI:10.1046/j.1462-2920.2001.00226.x (  0) 0) |
| [25] |
Costello A M, Lidstrom M E. Molecular characterization of functional and phylogenetic genes from natural populations of methanotrophs in lake sediments[J]. Applied and Environmental Microbiology, 1999, 65(11): 5066-5074 DOI:10.1128/AEM.65.11.5066-5074.1999 (  0) 0) |
| [26] |
DeLong E F. Archaea in coastal marine environments[J]. PNAS, 1992, 89(12): 5685-5689 DOI:10.1073/pnas.89.12.5685 (  0) 0) |
| [27] |
Holmes A J, Costello A, Lidstrom M E, et al. Evidence that participate methane monooxygenase and ammonia monooxygenase may be evolutionarily related[J]. FEMS Microbiology Letters, 1995, 132(3): 203-208 DOI:10.1111/j.1574-6968.1995.tb07834.x (  0) 0) |
| [28] |
Schloss P D, Westcott S L, Ryabin T, et al. Introducing mothur: Open-source, platform-independent, community-supported software for describing and comparing microbial communities[J]. Applied and Environmental Microbiology, 2009, 75(23): 7537-7541 DOI:10.1128/AEM.01541-09 (  0) 0) |
| [29] |
Collet S, Reim A, Ho A, et al. Recovery of paddy soil methanotrophs from long term drought[J]. Soil Biology and Biochemistry, 2015, 88: 69-72 DOI:10.1016/j.soilbio.2015.04.016 (  0) 0) |
| [30] |
夏围围, 贾仲君. 高通量测序和DGGE分析土壤微生物群落的技术评价[J]. 微生物学报, 2014, 54(12): 1489-1499 (  0) 0) |
| [31] |
Nicholson W L, Krivushin K, Gilichinsky D, et al. Growth of Carnobacterium spp. from permafrost under low pressure, temperature, and anoxic atmosphere has implications for Earth microbes on Mars[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(2): 666-671 DOI:10.1073/pnas.1209793110 (  0) 0) |
| [32] |
Reysenbach A L, Liu Y T, Banta A B, et al. A ubiquitous thermoacidophilic archaeon from deep-sea hydrothermal vents[J]. Nature, 2006, 442(7101): 444-447 DOI:10.1038/nature04921 (  0) 0) |
| [33] |
Shrestha U R, Bhowmik D, Copley J R, et al. Effects of pressure on the dynamics of an oligomeric protein from deep-sea hyperthermophile[J]. PNAS, 2015, 112(45): 13886-13891 DOI:10.1073/pnas.1514478112 (  0) 0) |
| [34] |
包丽君, 贾仲君. 模拟干湿交替对水稻土古菌群落结构的影响[J]. 土壤学报, 2017, 54(1): 191-203 (  0) 0) |
| [35] |
莫永亮, 郑燕, 金凤, 等. 内蒙古岗更诺尔湖泊退化情景下好氧甲烷氧化的微生物过程研究[J]. 微生物学报, 2019, 59(6): 1105-1115 (  0) 0) |
| [36] |
Whittenbury R, Davies S L, Davey J F. Exospores and cysts formed by methane-utilizing bacteria[J]. Journal of General Microbiology, 1970, 61(2): 219-226 DOI:10.1099/00221287-61-2-219 (  0) 0) |
| [37] |
Unger V, Liebner S, Koebsch F, et al. Congruent changes in microbial community dynamics and ecosystem methane fluxes following natural drought in two restored fens[J]. Soil Biology and Biochemistry, 2021, 160: 108348 DOI:10.1016/j.soilbio.2021.108348 (  0) 0) |
| [38] |
Ho A, Lüke C, Reim A, et al. Resilience of (seed bank) aerobic methanotrophs and methanotrophic activity to desiccation and heat stress[J]. Soil Biology and Biochemistry, 2016, 101: 130-138 DOI:10.1016/j.soilbio.2016.07.015 (  0) 0) |
| [39] |
Ho A, Kerckhof F M, Luke C, et al. Conceptualizing functional traits and ecological characteristics of methane-oxidizing bacteria as life strategies[J]. Environmental Microbiology Reports, 2013, 5(3): 335-345 DOI:10.1111/j.1758-2229.2012.00370.x (  0) 0) |
| [40] |
蔡元锋, 贾仲君. 土壤大气甲烷氧化菌研究进展[J]. 微生物学报, 2014, 54(8): 841-853 (  0) 0) |
| [41] |
Hakobyan A, Liesack W. Unexpected metabolic versatility among type Ⅱ methanotrophs in the Alphaproteobacteria[J]. Biological Chemistry, 2020, 401(12): 1469-1477 (  0) 0) |
| [42] |
Hakobyan A, Zhu J, Glatter T, et al. Hydrogen utilization by Methylocystis sp. strain SC2 expands the known metabolic versatility of type IIa methanotrophs[J]. Metabolic Engineering, 2020, 61: 181-196 (  0) 0) |
| [43] |
Miroshnikov K K, Belova S E, Dedysh S N. Genomic determinants of phototrophy in methanotrophic alphaproteobacteria[J]. Microbiology, 2019, 88(5): 548-555 (  0) 0) |
| [44] |
Rumah B L, Stead C E, Claxton Stevens B H, et al. Isolation and characterisation of Methylocystis spp. for poly-3-hydroxybutyrate production using waste methane feedstocks[J]. AMB Express, 2021, 11(1): 6 (  0) 0) |
| [45] |
Sahoo K K, Goswami G, Das D. Biotransformation of methane and carbon dioxide into high-value products by methanotrophs: Current state of art and future prospects[J]. Frontiers in Microbiology, 2021, 12: 636486 (  0) 0) |
| [46] |
Dumont M G, Lüke C, Deng Y C, et al. Classification of pmoA amplicon pyrosequences using BLAST and the lowest common ancestor method in MEGAN[J]. Frontiers in Microbiology, 2014, 5: 34 (  0) 0) |
2. University of Chinese Academy of Sciences, Beijing 100049, China
 2023, Vol. 55
2023, Vol. 55


