2. 中国科学院长春光学精密机械与物理研究所, 长春 130033;
3. 长春长光辰英生物科学仪器有限公司, 长春 130033
完整地认识地球微生物群落在生物圈和人类健康中的作用,是解决21世纪人类社会从能源、传染病到农业等诸多领域所面临许多难题的关键[1-3]。2015年,《Nature》呼吁启动“国际微生物组计划”,帮助人类全面认识地球环境中微生物群落的数量与功能,为解决上述问题提供了关键资源[3]。但随着研究的深入,越来越多的学者认识到基因组水平、蛋白质组学以及代谢组学层面的研究只能获得统计学上的相关性结果,缺乏因果性结论,其最核心问题在于缺乏菌种的验证性实验。因此菌种筛选被日益推到了学科的前沿,获取目标微生物后进行因果性的验证实验有助于科学家得到最直观准确的结论,进而推动相关科学发展及其在实际应用方面的进步。
实际上,科研工作者一直致力于筛选菌种。1996年,Rainey和Bailey[4]从英国牛津大学农场种植的甜菜叶子表面分离出的假单胞菌SBW25除了用于研究微生物-植物-土壤相互作用外,已成为研究进化过程的重要模式生物。2006年,Chen等[5]从台中市一处未经种植地区的土壤中筛选出了36株溶磷细菌,并获得其分泌的3种新型有机酸,为用于田间的磷酸溶解剂提供了更多选择。2015年,Yadav等[6]从印度喜马拉雅山西北部寒冷沙漠采集的不同样本中筛选出了232种细菌,丰富了人类对寒冷高海拔栖息地菌种组成的认识。同年,Daims等[7]从俄罗斯一口石油探井管壁上生长的微生物膜中培养分离出能一步完全硝化的微生物菌株,打破了科学家125年来认为铵根硝化需要两类微生物协作的认知。除了推动科学发展外,菌种筛选也解决了很多重大实际问题。2015年,诺贝尔生理学或医学奖获得者之一——日本科学家大村智于1974年从土壤中分离到一株链霉菌,通过与美国Merck公司合作,发现该链霉菌生产的二氢衍生物Ivermectin可制成药物用于彻底防治盘尾丝虫症(河盲症)和淋巴丝虫病(象皮病)[8-9]。1989年,Robertson等[10]报道了好氧反硝化细菌和好氧反硝化酶的存在,为生物脱氮技术提供了一种崭新的思路。2002年,Dean-Ross等[11]从大卡卢梅特河的沉积物中分离出两种能使多环芳烃降解的细菌(分枝杆菌和红霉菌),推动了生物修复工业污染物用地的进展。2009年,Hong等[12]从中国的8个红树林中筛选出与抗癌和抗感染相关的金黄色葡萄球菌等,为新药物的研发提供了可能性。2018年,Mitra等[13]从印度某钢铁厂附近水稻田的土壤中筛选到了有重金属抗性的S2菌株,为后续研究重金属污染农业土壤的生物修复奠定了基础。以上成果极大地推动了医学、工业和环境工程等领域的进步,同时也表明了纯菌株的分离具有极为重要的意义,应在未来的微生物研究中得到重视。
综上所述,获取纯菌株的微生物分离培养技术是微生物学和微生物研究相关学科的核心内容。地球上栖息着多样性丰富的生物资源,而人类对这些资源的认知和开发仅是冰山一角,定向发掘微生物功能对人类社会发展具有不可估量的意义[14]。基此,本文拟对当前国内外主流的微生物菌种筛选技术方法进行介绍,并阐明各自的优缺点,以及针对该领域未来发展方向提出些粗浅的看法,抛砖引玉。
1 传统方法及其优缺点传统微生物筛选方法的主要思路为先培养后筛选,即先选择合适的培养条件(例如培养基、培养环境等)筛分环境样品中的目标微生物,再从中挑选[15-16]。传统筛选方法能够获得纯菌株,了解目标微生物的种水平、基因组和性状,并使用纯菌株进行后续验证性实验。虽然该方法经过多年的发展与完善已形成较为成熟的体系,但仍存在两个无法避免的问题。
首先,当目标微生物的丰度很低或生长十分缓慢时极易被忽略。许多微生物,尤其是寡营养微生物的生长十分缓慢,需要较长的培养时间。只有抑制或降低群落中生长较快菌株的生长和丰度,才有可能筛选出此类细菌。例如,2005年Davis等[17]将土壤细菌置于黑暗条件下的聚乙烯袋中密封培养长达3个月,发现菌群数量持续不断增加,且之前难以分离出的菌株获取率也随着时间的推移而增加。2009年Song等[18]通过在黑暗低温条件下长达6个月的培养实验,成功从西太平洋海水样品中分离出滞后期较长的SAR11菌株。2005年Tamaki等[19]提出细菌的生长也可能受到混合培养中其他细菌释放的细菌素或培养基内抗菌物质的抑制。
其次,培养基的养分条件和菌株的原生环境差异大,导致目标微生物无法存活。例如,传统筛选方法使用的培养基大多富含营养物质,这类培养基利于生长较快的细菌,但适应贫乏营养环境及生长缓慢的菌种可能受到抑制[19]。2002年Connon和Giovannoni[20]发现使用稀释营养物质的培养基能成功培育出以前不可培养的水生和陆生细菌。但上述解决方法仍费时费力,有待进一步改进。2005年Kopke等[21]调查了不同基质和培养条件对潮汐沉积物中细菌生长的影响,发现不同的培养方法会分离出其特有的细菌,证实了培养条件对菌种的影响。2007年Kim[22]发现许多细菌的生长依赖于某些信号分子,而信号分子只存在于自然栖息地中。当此类细菌面对缺少信号分子这一生存要素的培养基时,可能会选择一种暂时进入低代谢活动状态的生存策略,从而无法在培养介质上增殖并形成菌落[22-23]。由于上述情况,传统的平板筛分方法获得的菌种数量和多样性很低,无法满足农业生产、环境治理、医疗卫生等领域的需求。
2 新技术方法及其应用新的微生物筛选技术方法的思路主要为先筛选后培养,可以有效规避传统方法的劣势。而该思路的实现主要归功于从复杂样本中识别及分离单个微生物细胞技术的进步、计算方法和成像技术的改进,以及用于数据解析的生物信息学工具的革新。这些技术的发展极大提高了微生物学家筛分微生物的成功率,并减少了人力成本。下面将简单介绍几种技术方法。
2.1 基于光学镊子结合拉曼光谱的细胞分选技术光学镊子结合拉曼光谱(laser tweezers and Raman spectroscopy,LTRS)是一种新兴的细胞筛分技术。其中,光学镊子是建立在光辐射压原理上,采用高度聚焦的激光束提供引力或斥力,从而以物理方式固定和移动微观中性物体的技术[24];拉曼光谱则是1928年被印度物理学家Raman所发现[25],它蕴含着细胞在特定生理状态下丰富的生化信息,如核酸、蛋白质、脂质等,可作为“化学指纹”用于描述细胞的分子组成、生理状态和生态功能[26]。二者相结合则可实现无标记检测、区分及分选单细胞[27-28]。
2007年黄超等[29]提出将LTRS技术与微流控芯片结合检测红细胞的拉曼光谱,发现二者结合后检测更为准确、快速。2009年,Huang等[27]应用LTRS识别毛细血管中的细胞,再通过毛细血管将其分选,为LTRS技术用于细胞分选提供了可能性。2015年,Berry等[28]利用重水(D2O)的稳定同位素(D)探测来快速识别小鼠盲肠菌群的活性微生物,明确了样本中两种主要微生物的活动模式并发现了不明细菌。该研究证明拉曼–FISH预筛选的应用,可在一定程度上解决因目标细胞尺寸小和易损性而导致的手动LTRS分选过程慢等问题,进而使LTRS技术能用于筛分复杂样品的菌种。2016年Casabella等[30]为了提高细胞分选率,将LTRS技术与微流体芯片结合,该应用使单个细胞能自动被捕获并通过拉曼检测分析,大大节省了人工成本。
LTRS技术在实际应用方面仍存在两个问题。首先,高通量LTRS技术的实现依赖于结合微流体技术,而该技术需将样品细胞悬浮于溶液中进行操作。但许多样本中细胞分布在生物膜、沉积物、土壤及人/动物粪便中,上述复杂样品中的大颗粒物质或碎屑易堵塞微流体设备中的喷嘴和通道,使得操作十分困难[31];其次,另一个待解决的问题是激光对细胞的损伤,特别是一些反应中心对光损伤敏感的光养生物。光损伤效应取决于波长,现已发现经735、785、835 nm的辐射会不可逆地抑制微藻Trachydiscus sp.的光化学活性;而功率为25 mW的885、935和1 064 nm的激光则无不利影响[32]。此外,激光对非光养细胞也有不利影响,例如能量为0.36 J的1 064 nm激光镊子会影响大肠杆菌的细胞分裂,能量为0.54 J时会影响细胞生长[33]。上述两个问题该如何解决将成为LTRS技术能否广泛应用的关键。
2.2 荧光活化细胞分选法荧光活化细胞分选法(fluorescence-activated cell sorting,FACS)是一项新型的、发展迅速的细胞分析技术。其原理是采用激光束激发悬浮在液流中的单行细胞或生物颗粒,探测其荧光及散射光,从而完成对细胞物理和生化特性的高度定量分析与分选[34-35]。流式细胞仪(flow cytometer, FCM)就是以FACS为核心的仪器。它因具有测量指标多、检测速度快、分选纯度高等优点,而被广泛应用于临床医学、细胞学、环境检测等领域的科学研究和实际应用中。它在新兴的生物技术(如系统生物学,稀有细胞检测或干细胞分离)方面也有巨大的潜力[36]。
1979年,Parks等[37]就已证明只要有荧光信号,即使特异性标记抗原结合细胞在样品中含量极少,也可用FACS将其分选出。1988年,Webster等[38]提出FACS的独特能力可使细胞在培养时间最小化的同时产量最大化,从而使得科学家可分析研究在体外寿命极其有限的成肌细胞。2009年,蔡海英等[39]证明FACS可应用于体外培养的兔角膜缘上皮细胞的SP细胞分离,且分离出的细胞具有较强的增生能力。2015年,Lawson等[40]利用高敏感度FACS分析并分选到小鼠的原发性肿瘤细胞和转移性细胞,推进了对转移性疾病的预防及治疗策略的开发。2018年,金鑫等[41]利用多参数FACS研究发现慢性粒单核细胞白血病(CMML)和反应性增多的单核细胞的免疫学分型结果存在较多差异,综合这些鉴别点可辅助区分二者,对CMML的诊断具有重要意义。
FACS也存在不可避免的缺点。其一,它的台式系统笨重且成本高昂,因此它们大多为各种生物研究或临床实验室所共有,可用性与易用性较低;其二,传统FCM通常为开放式系统,因此科学家处理传染性样本需极其谨慎,以防基于水滴的分析与分选机制会产生有害气溶胶[42];其三,确保样品分选质量的程序费时费力,如无RNase处理需要对FCM的整个管路和过滤器系统进行灭菌及清洁[36];其四,FACS需要大量的样品和试剂进行分析,进一步增加了总成本,使许多应用变得困难或不可能[35]。综上所述,尽管FACS有强大的功能,但仍有诸多因素限制其在各种研究和应用中的利用。因此,克服问题并改进仪器将成为FACS进一步发展的要点。
2.3 基于激光诱导向前转移技术的细胞分选技术分选单细胞需运用激光诱导向前转移(laser- induced forward transfer, LIFT)技术,该技术基于激光与物质的相互作用原理,可非接触地从复杂生物样本中将目标单细胞弹射至接收器中,实现准确、无损的单细胞分离(图 1),且分选后的细胞可直接进行全基因组扩增或纯培养等后续实验[35, 43]。基于LIFT技术的细胞筛分方法相比于传统的细胞分离方法具有分选精准、流程简易、适用广泛等优势。此外,LIFT细胞分选技术与FACS的区别在于前者具有高分辨率的显微成像系统,可利用目标微生物的细胞形态、尺寸等自身特征无标记地识别生物体与非生物颗粒,再进行目标单细胞的自动或手动分选。总而言之,LIFT细胞分选技术突破了传统的微生物研究领域,开创了微生物单细胞基因组学研究的新方法,或将拓展人类对未培养微生物的认知。
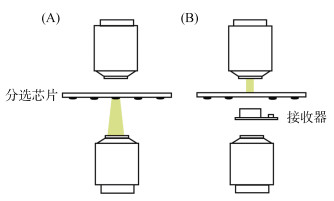
|
(A:显微识别模式;B:分选模式) 图 1 LIFT分选单细胞原理示意图 Fig. 1 LIFT sorting single cell principle |
2013年,Wang等[44]运用LIFT技术精准快速地从复杂的口腔细菌样本中分离出5种不同类型的细菌,验证了该方法的可行性。2017年,Song等[43]利用LIFT技术从复杂的海水样品中分离产类胡萝卜素的光合细菌,从而通过单细胞全基因组测序进一步研究类胡萝卜素合成途径;同年他们还使用相同的方法从泰晤士河中分离出抗性微生物并测序分析,建立了表型与基因型的对应关系[45]。
LIFT细胞分选技术虽然可实现从复杂的样品中分离出目标微生物,但如何准确定位目标微生物仍有待解决,于是有科学家提出了拉曼活化细胞分选(raman activated cell sorting,RACS)这一种无需标记的细胞分选方法[31]。2012年Stöckel等[46]利用芽孢的特殊拉曼光谱从7种粉末与芽孢杆菌的混合样品中准确找到芽孢杆菌;2017年,Song等[43]利用类胡萝卜素的特殊拉曼光谱从复杂的海水样品中筛选光合细菌。2018年,Jing等[47]以13C底物培养海水样品中的微生物,成功利用RACS方法从中分离出了原位固碳单细胞群,并测定其DNA序列,以重构其基因组。
综上所述,拉曼光谱结合单细胞分选技术可绕过纯菌株培养阶段,直接实现活体单细胞的分离,快速便捷且省时省力。但此种方法也存在相应问题:首先,自发的拉曼散射较弱,约107个入射光子中仅有1个进行拉曼散射[48],因此实验需要较长的时间(通常为几秒钟)来获取细胞的拉曼光谱,从而限制了RACS高通量的实现;其次,经LIFT分选技术弹射出的细胞活性会受一定程度影响,自身抗逆性差的微生物可能无法在分选后存活,从而使得后续培养实验无法进行。因此,拉曼光谱结合单细胞分选技术想要发展成熟仍有很长的路要走。
2.4 基于原位培养的细胞分离技术经过科学家几十年来不断对可培养微生物的重复培养、筛选后,想从中分离出结构独特、活性显著的新物种越来越困难。通过长期研究,人们已经认识到目前尚可培养的微生物仅占微生物总数的小部分[49],而迄今为止无法在实验室纯培养的微生物包含着丰富的物种多样性与巨大的可利用潜能。为了解决这一问题,科学家突破了传统的培养方式,提出了原位培养技术。该技术的原理是通过合理模拟某种微生物生长繁殖的自然条件,以自然环境作为培养基,即可在不了解其生长所需全部元素组成的情况下对其进行培养。该方法作为对传统菌种筛选方法的改良,也遵循先培养后筛选的主要思路。
2002年,Kaeberlein等[50]设计了扩散室(diffusion chamber)用于原位培养海洋潮汐带沉积物中的微生物,扩散室中的微生物细胞可通过滤膜与潮汐带沉积物进行物质信息交流(图 2),研究发现该方法的微生物获取率比传统培养方法高300倍,且培养出了无法在传统培养基中生存的MSC2。2008年,Gavrish等[51]以放线菌菌丝可以穿透0.22 μm孔径的半透膜为基础,发明了用于捕获及原位培养丝状放线菌的“陷阱”——trap装置,实验证明该方法分离到的放线菌数量与多样性较传统方法都有很大提高,且能获得一些稀有放线菌。2015年,姜明国等[52]利用原位培养方法从红树林根际土壤中分离出了包含疑似新属放线菌在内的多种微生物,研究表明该方法分离出的微生物较传统方法拥有更高的多样性与活性。2017年,Berdy等[53]研究出了高通量的微型扩散室——隔离芯片(isolation chip,IChip),该装置也可使微生物回收率提高至300倍且已成为分离新型抗菌微生物的主要培养工具。

|
(A:扩散室是由两个0.03 μm的聚碳酸酯膜及夹在二者之间的垫圈组成的;B:扩散室被培养在海洋沉积物的表面) 图 2 扩散室原位培养环境微生物示意图[50] Fig. 2 Diffusion growth chamber for in situ cultivation of environmental microorganisms[50] |
原位培养的关键优势在于科学家无需猜测新物种的生长需求,直接在满足其生长繁殖需求的自然条件中进行培养,使用自然环境中的生长因子作为生长介质[50, 53]。与此同时,原位培养也存在一个问题,即依旧无法于复杂的自然环境中了解微生物生存所必需的物质。Kaeberlein等[50]提出一种信号假说,即微生物可能需要邻近微生物发出的特定信号,表明此为熟悉环境后再生长繁殖,其隐含意义为即使提供适当的营养条件,微生物也无法在陌生的环境中生长。这可在一定程度上解释人工配制的传统培养基为何无法培养出大多微生物。这个假说为今后的研究提供了一种可能的方向,而真实的原因还有待科学家不断探究与突破,从而进一步推动基于原位培养的微生物分离技术的发展。
3 新技术发展展望任何单一技术都不是完美的。从复杂环境中筛选目标微生物,虽然传统菌种筛选方法费时费力,但现代主流基于单细胞分选的技术也存在难以完成精准筛选,即靶标性差等问题。因此,研究实践中不能偏重或偏废某个单一技术,更应该结合多个技术,取长补短。只有耦合多个技术的优势,才能真正地推进微生物菌种的筛选。在此,提出如下几个初步想法:
1) 传统方法与现代方法有机结合。使用传统方法初筛富集后再利用现代方法精准筛选,将二者相结合做到既快速又准确。传统方法在筛选菌种的过程中仍起到非常重要的作用,通过传统方法可于复杂环境中富集目标微生物或者弱化非目标微生物,为现代方法的实施提供了便利性和成功率。反之,现代方法可以由人工智能提供自动化的手段,大大降低了因人工操作导致的低效率和可能的错误率。两者结合将极大促进菌种筛选的过程。
2) 加强源头创新。选择有代表性的供试土壤有助于提高筛选目标微生物成功率,利用大自然已经长期驯化富集群落的场地将会使得筛选过程事半功倍。
3) 关注种间互作。物种间相生相克的机制也是筛选土壤微生物的困难之一,特别是营养缺陷型物种。应多关注微生物的种间互作,例如从海洋潮汐带沉积物中分离出的纯菌株MSC1和MSC2只能通过共培养于培养皿中生长[50]。
4) 应用快速检测。随着今后菌种筛选通量的增大,需考虑结合快速检测方法(例如酶活显色反应[54]、核酸测定[55]等),以便精准快速地从复杂环境中锁定目标菌种,特别是具有高活性和特定功能的菌种,进而为下游分析提供良好的材料。
5) 统筹下游分析。在筛得大量菌种后,还需思考如何更好地耦合下游分析,例如如何将细胞分选技术与单细胞测序、宏基因分析和宏转录组分析等结合起来。
6) 利用分选技术人工构建微生物群落,以更好地揭示微生物群落对生态系统的影响。
总之,只有多角度、多方位、多技术地整合才能精准、高通量、快速地从大自然中获得菌种。只有充分获得微生物资源,才能够更好地推动土壤微生物学,以及相关学科的进一步发展,才能真正地利用大自然赋予的瑰宝——微生物,反哺于人类、社会和环境的可持续性发展。
| [1] |
杨珍, 戴传超, 王兴祥, 等. 作物土传真菌病害发生的根际微生物机制研究进展[J]. 土壤学报, 2019, 56(1): 12-22 (  0) 0) |
| [2] |
聂三安, 王祎, 王飞, 等. 稻田土壤厌氧氨氧化菌群落结构对长期不同施肥的响应[J]. 土壤学报, 2018, 55(3): 744-753 (  0) 0) |
| [3] |
Dubilier N, Mcfall-Ngai M, Zhao L P. Microbiology: Create a global microbiome effort[J]. Nature, 2015, 526(7575): 631-634 DOI:10.1038/526631a (  0) 0) |
| [4] |
Rainey P B, Bailey M J. Physical and genetic map of the Pseudomonas fluorescens SBW25 chromosome[J]. Molecular Microbiology, 1996, 19(3): 521-533 DOI:10.1046/j.1365-2958.1996.391926.x (  0) 0) |
| [5] |
Chen Y P, Rekha P D, Arun A B, et al. Phosphate solubilizing bacteria from subtropical soil and their tricalcium phosphate solubilizing abilities[J]. Applied Soil Ecology, 2006, 34(1): 33-41 DOI:10.1016/j.apsoil.2005.12.002 (  0) 0) |
| [6] |
Yadav A N, Sachan S G, Verma P, et al. Prospecting cold deserts of north western Himalayas for microbial diversity and plant growth promoting attributes[J]. Journal of Bioscience and Bioengineering, 2015, 119(6): 683-693 DOI:10.1016/j.jbiosc.2014.11.006 (  0) 0) |
| [7] |
Daims H, Lebedeva E V, Pjevac P, et al. Complete nitrification by Nitrospira bacteria[J]. Nature, 2015, 528(7583): 504-509 DOI:10.1038/nature16461 (  0) 0) |
| [8] |
Omura S. The antibiotic cerulenin, a novel tool for biochemistry as an inhibitor of fatty acid synthesis[J]. Bacteriological Reviews, 1976, 40(3): 681-697 DOI:10.1128/BR.40.3.681-697.1976 (  0) 0) |
| [9] |
Campbell W C, Fisher M H, Stapley E O, et al. Ivermectin: A potent new antiparasitic agent[J]. Science, 1983, 221(4613): 823-828 DOI:10.1126/science.6308762 (  0) 0) |
| [10] |
Robertson L A, Cornelisse R, de Vos P, et al. Aerobic denitrification in various heterotrophic nitrifiers[J]. Antonie Van Leeuwenhoek, 1989, 56(4): 289-299 DOI:10.1007/BF00443743 (  0) 0) |
| [11] |
Dean-Ross D, Moody J, Cerniglia C E. Utilization of mixtures of polycyclic aromatic hydrocarbons by bacteria isolated from contaminated sediment[J]. FEMS Microbiology Ecology, 2002, 41(1): 1-7 DOI:10.1111/j.1574-6941.2002.tb00960.x (  0) 0) |
| [12] |
Hong K, Gao A H, Xie Q Y, et al. Actinomycetes for marine drug discovery isolated from mangrove soils and plants in China[J]. Marine Drugs, 2009, 7(1): 24-44 DOI:10.3390/md7010024 (  0) 0) |
| [13] |
Mitra S, Pramanik K, Sarkar A, et al. Bioaccumulation of cadmium by Enterobacter sp. and enhancement of rice seedling growth under cadmium stress[J]. Ecotoxicology and Environmental Safety, 2018, 156: 183-196 DOI:10.1016/j.ecoenv.2018.03.001 (  0) 0) |
| [14] |
DeLong E F. Marine microbial diversity: The tip of the iceberg[J]. Trends in Biotechnology, 1997, 15(6): 203-207 DOI:10.1016/S0167-7799(97)01044-5 (  0) 0) |
| [15] |
张红芳, 何刚, 吴绿英, 等. 一株高效解钾脱硅真菌的筛选、鉴定及培养条件优化[J]. 土壤, 2018, 50(5): 934-941 (  0) 0) |
| [16] |
廖青, 刘永贤, 邢颖, 等. 土壤富硒细菌的筛选、鉴定[J]. 土壤, 2018, 50(6): 1203-1207 (  0) 0) |
| [17] |
Davis K E R, Joseph S J, Janssen P H. Effects of growth medium, inoculum size, and incubation time on culturability and isolation of soil bacteria[J]. Applied and Environmental Microbiology, 2005, 71(2): 826-834 DOI:10.1128/AEM.71.2.826-834.2005 (  0) 0) |
| [18] |
Song J, Oh H M, Cho J C. Improved culturability of SAR11 strains in dilution-to-extinction culturing from the East Sea, West Pacific Ocean[J]. FEMS Microbiology Letters, 2009, 295(2): 141-147 DOI:10.1111/j.1574-6968.2009.01623.x (  0) 0) |
| [19] |
Tamaki H, Sekiguchi Y, Hanada S, et al. Comparative analysis of bacterial diversity in freshwater sediment of a shallow eutrophic lake by molecular and improved cultivation-based techniques[J]. Applied and Environmental Microbiology, 2005, 71(4): 2162-2169 DOI:10.1128/AEM.71.4.2162-2169.2005 (  0) 0) |
| [20] |
Connon S A, Giovannoni S J. High-throughput methods for culturing microorganisms in very-low-nutrient media yield diverse new marine isolates[J]. Applied and Environmental Microbiology, 2002, 68(8): 3878-3885 DOI:10.1128/AEM.68.8.3878-3885.2002 (  0) 0) |
| [21] |
Köpke B, Wilms R, Engelen B, et al. Microbial diversity in coastal subsurface sediments: A cultivation approach using various electron acceptors and substrate gradients[J]. Applied and Environmental Microbiology, 2005, 71(12): 7819-7830 DOI:10.1128/AEM.71.12.7819-7830.2005 (  0) 0) |
| [22] |
Kim L. Persister cells, dormancy and infectious disease[J]. Nature Reviews Microbiology, 2007, 5(1): 48-56 DOI:10.1038/nrmicro1557 (  0) 0) |
| [23] |
Colwell R R. Viable but nonculturable bacteria: a survival strategy[J]. Journal of Infection and Chemotherapy, 2000, 6(2): 121-125 DOI:10.1007/PL00012151 (  0) 0) |
| [24] |
Zhang H, Liu K K. Optical tweezers for single cells[J]. Journal of the Royal Society Interface, 2008, 5(24): 671-690 DOI:10.1098/rsif.2008.0052 (  0) 0) |
| [25] |
Raman C V, Krishnan K S. A new type of secondary radiation[J]. Nature, 1928, 121(3048): 501-502 (  0) 0) |
| [26] |
Huang W E, Griffiths R I, Thompson I P, et al. Raman microscopic analysis of single microbial cells[J]. Analytical Chemistry, 2004, 76(15): 4452-4458 DOI:10.1021/ac049753k (  0) 0) |
| [27] |
Huang W E, Ward A D, Whiteley A S. Raman tweezers sorting of single microbial cells[J]. Environmental Microbiology Reports, 2009, 1(1): 44-49 DOI:10.1111/j.1758-2229.2008.00002.x (  0) 0) |
| [28] |
Berry D, Mader E, Lee T K, et al. Tracking heavy water (D2O) incorporation for identifying and sorting active microbial cells[J]. PNAS, 2015, 112(2): E194-E203 DOI:10.1073/pnas.1420406112 (  0) 0) |
| [29] |
黄超, 王强, 姚辉璐, 等. 微流控芯片-拉曼光镊的红细胞光谱检测技术[J]. 分析化学, 2007, 35(10): 1410-1414 DOI:10.3321/j.issn:0253-3820.2007.10.004 (  0) 0) |
| [30] |
Casabella S, Scully P, Goddard N, et al. Automated analysis of single cells using Laser Tweezers Raman Spectroscopy[J]. The Analyst, 2016, 141(2): 689-696 DOI:10.1039/C5AN01851J (  0) 0) |
| [31] |
Song Y Z, Yin H B, Huang W E. Raman activated cell sorting[J]. Current Opinion in Chemical Biology, 2016, 33: 1-8 DOI:10.1016/j.cbpa.2016.04.002 (  0) 0) |
| [32] |
Pilát Z, Ježek J, Šerý M, et al. Optical trapping of microalgae at 735-1064 nm: Photodamage assessment[J]. Journal of Photochemistry and Photobiology B: Biology, 2013, 121: 27-31 DOI:10.1016/j.jphotobiol.2013.02.006 (  0) 0) |
| [33] |
Ayano S, Wakamoto Y, Yamashita S, et al. Quantitative measurement of damage caused by 1064-nm wavelength optical trapping of Escherichia coli cells using on-chip single cell cultivation system[J]. Biochemical and Biophysical Research Communications, 2006, 350(3): 678-684 DOI:10.1016/j.bbrc.2006.09.115 (  0) 0) |
| [34] |
Bang H, Chung C, Kim J K, et al. Microfabricated fluorescence-activated cell sorter through hydrodynamic flow manipulation[J]. Microsystem Technologies, 2006, 12(8): 746-753 DOI:10.1007/s00542-005-0076-9 (  0) 0) |
| [35] |
Cho S H, Chen C H, Tsai F S, et al. Human mammalian cell sorting using a highly integrated micro-fabricated fluorescence-activated cell sorter (microFACS)[J]. Lab on a Chip, 2010, 10(12): 1567-1573 DOI:10.1039/c000136h (  0) 0) |
| [36] |
Schmid L, Weitz D A, Franke T. Sorting drops and cells with acoustics: acoustic microfluidic fluorescence-activated cell sorter[J]. Lab on a Chip, 2014, 14(19): 3710-3718 DOI:10.1039/C4LC00588K (  0) 0) |
| [37] |
Parks D R, Bryan V M, Oi V T, et al. Antigen-specific PNAS, 1979, 76(4): 1962-1966.
(  0) 0) |
| [38] |
Webster C, Pavlath G K, Parks D R, et al. Isolation of human myoblasts with the fluorescence-activated cell sorter[J]. Experimental Cell Research, 1988, 174(1): 252-265 DOI:10.1016/0014-4827(88)90159-0 (  0) 0) |
| [39] |
蔡海英, 柳林, 彭亮红. 荧光激活细胞分离技术在角膜缘干细胞研究中的应用[J]. 眼科研究, 2009, 27(12): 1064-1067 (  0) 0) |
| [40] |
Lawson D A, Bhakta N R, Kessenbrock K, et al. Single-cell analysis reveals a stem-cell program in human metastatic breast cancer cells[J]. Nature, 2015, 526(7571): 131-135 DOI:10.1038/nature15260 (  0) 0) |
| [41] |
金鑫, 林慧君, 吴茅. 多参数流式细胞术在慢性粒单核细胞白血病诊断中的应用[J]. 中国卫生检验杂志, 2018, 28(6): 682-685, 688 (  0) 0) |
| [42] |
Godin J, Lien V, Lo Y H. Demonstration of two-dimensional fluidic lens for integration into microfluidic flow cytometers[J]. Applied Physics Letters, 2006, 89(6): 061106 DOI:10.1063/1.2266887 (  0) 0) |
| [43] |
Song Y Z, Kaster A K, Vollmers J, et al. Single-cell genomics based on Raman sorting reveals novel carotenoid-containing bacteria in the Red Sea[J]. Microbial Biotechnology, 2017, 10(1): 125-137 DOI:10.1111/1751-7915.12420 (  0) 0) |
| [44] |
Wang Y, Ji Y T, Wharfe E S, et al. Raman activated cell ejection for isolation of single cells[J]. Analytical Chemistry, 2013, 85(22): 10697-10701 DOI:10.1021/ac403107p (  0) 0) |
| [45] |
Song Y Z, Cui L, López J Á S, et al. Raman-Deuterium Isotope Probing for in situ identification of antimicrobial resistant bacteria in Thames River[J]. Scientific Reports, 2017, 7: 16648 DOI:10.1038/s41598-017-16898-x (  0) 0) |
| [46] |
Stöckel S, Meisel S, Elschner M, et al. Raman spectroscopic detection of anthrax endospores in powder samples[J]. Angewandte Chemie International Edition, 2012, 51(22): 5339-5342 DOI:10.1002/anie.201201266 (  0) 0) |
| [47] |
Jing X Y, Gou H L, Gong Y H, et al. Raman-activated cell sorting and metagenomic sequencing revealing carbon-fixing bacteria in the ocean[J]. Environmental Microbiology, 2018, 20(6): 2241-2255 DOI:10.1111/1462-2920.14268 (  0) 0) |
| [48] |
Huang W E, Stoecker K, Griffiths R, et al. Raman-FISH: combining stable-isotope Raman spectroscopy and fluorescence in situ hybridization for the single cell analysis of identity and function[J]. Environmental Microbiology, 2007, 9(8): 1878-1889 DOI:10.1111/j.1462-2920.2007.01352.x (  0) 0) |
| [49] |
Amann R I, Ludwig W, Schleifer K H. Phylogenetic identification and in situ detection of individual microbial cells without cultivation[J]. Microbiological Reviews, 1995, 59(1): 143-169 DOI:10.1128/MR.59.1.143-169.1995 (  0) 0) |
| [50] |
Kaeberlein T, Lewis K, Epstein S S. Isolating "uncultivable" microorganisms in pure culture in a simulated natural environment[J]. Science, 2002, 296(5570): 1127-1129 DOI:10.1126/science.1070633 (  0) 0) |
| [51] |
Gavrish E, Bollmann A, Epstein S, et al. A trap for in situ cultivation of filamentous actinobacteria[J]. Journal of Microbiological Methods, 2008, 72(3): 257-262 DOI:10.1016/j.mimet.2007.12.009 (  0) 0) |
| [52] |
姜明国, 甘光华, 杨立芳, 等. 广西红树林根际土壤放线菌的原位培养分离及其活性筛选[J]. 海洋学报, 2015, 37(2): 55-64 DOI:10.3969/j.issn.0253-4193.2015.02.006 (  0) 0) |
| [53] |
Berdy B, Spoering A L, Ling L L, et al. In situ cultivation of previously uncultivable microorganisms using the ichip[J]. Nature Protocols, 2017, 12(10): 2232-2242 DOI:10.1038/nprot.2017.074 (  0) 0) |
| [54] |
Greis K D. Mass spectrometry for enzyme assays and inhibitor screening: An emerging application in pharmaceutical research[J]. Mass Spectrometry Reviews, 2007, 26(3): 324-339 DOI:10.1002/mas.20127 (  0) 0) |
| [55] |
Schaffitzel C, Hanes J, Jermutus L, et al. Ribosome display: An in vitro method for selection and evolution of antibodies from libraries[J]. Journal of Immunological Methods, 1999, 231(1/2): 119-135 (  0) 0) |
2. Changchun Institute of Optics, Fine Mechanics and Physics, Chinese Academy of Sciences, Changchun 130033, China;
3. HOOKE Instruments Ltd., Changchun 130033, China
 2021, Vol. 53
2021, Vol. 53


